edition des genomes de mammiferes pdf.pptx

!"#$%&'"()'*+&%,()'"('
,-,,#./0()'
'
Responsable: XX
!"#"$%&$"'()*%$"+'
,'-./0'1234'
5)67*"'8%)*)9%"':;<=/%-"#+.*"'>#%-.*"'
"+'5)6=*%0.?)#'@/=6%A?$"'

.02
Production of genetically modified vertebrates from early 80’s
1234' 35' 36' 37' 38' 33' 32' 24' 21' 29' 27' 28' 23' 22' 41' 45' 4:' 49' 46'
;0-&)*(&#<',%=)('
>#-&?'
,%=)('
;0-&)*(&#<'0-?)'
;0-&)*(&#<'<-@A('
43' 42' 14' 11'
44'
B(&$C#0-A'?0-&)*(&()#)'
#&',%=)(D')E((FD'F#*'
;0-&)F%)%&)'#&'F#*)'
G-HH#?'
B(&$C#0-A'
?0-&)*(&()#)'#&'
0-HH#?'
I#*'
JE((F'
K)E'
;0-&)F%)#$%&'#&'
0-HH#?)'
15' 1:' 19' 16' 17'
;0-&)*(&()#)'C#-'
?0-&)F%)%&)'#&'
,("-L-'
;0-&)*(&()#)'
C#-'
?0-&)F%)%&)'
#&',%=)('

.03
B7"**"0'-)6%CA.?)#0D''
@/)67%/"'6"0')/9.#%0-"0'9=#=?E7"-"#+'-)6%C=0'
57+.9=#&0"'
>66%?)#'6"'9&#"'
F7<</%-"/'*G";</"00%)#'6G7#'9&#"'
5)6%C"/'*G";</"00%)#'6G7#'9&#"'
@/)$)E7"/'*G";</"00%)#'6G7#'
9&#"'#)7$".7'
@)7/'H.%/"'E7)%D'
I' J'J'>'>'>' !'J' I' J'J'>'>'>' >'J'
★'

.04
B7"**"0'-)6%CA.?)#0D''
@/)67%/"'6"0')/9.#%0-"0'9=#=?E7"-"#+'-)6%C=0'
57+.9=#&0"'
>66%?)#'6"'9&#"'
F7<</%-"/'*G";</"00%)#'6G7#'9&#"'
5)6%C"/'*G";</"00%)#'6G7#'9&#"'
@/)$)E7"/'*G";</"00%)#'6G7#'
9&#"'#)7$".7'
@)7/'H.%/"'E7)%D'
I' J'J'>'>'>' !'J' I' J'J'>'>'>' >'J'
★'
>9"#+0'AK%-%E7"0'
L//.6%.?)#'

.05
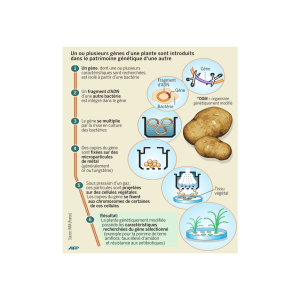
@/)67%/"'6"0')/9.#%0-"0'9=#=?E7"-"#+'-)6%C=0M'
M:#'A%N*.#+'*.'-)6%CA.?)#'9=#=?E7"'
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
1
/
40
100%