Micro-ARN et inflammation : ce que l`étude du rejet aigu

Le Courrier de la Transplantation - Vol. XII - n° 1 - janvier-février-mars 2012
22
Dossier thématique
Biomarqueurs
Résumé
Summary
»
L’implication des microARN dans la régulation de l’immunité
innée et adaptative a récemment été identifi ée. Nos travaux, qui
ont été parmi les premiers à faire entrer les miARN dans le champ
de la biologie de la transplantation, suggèrent leur implication
potentielle comme biomarqueurs de rejet, et pourraient également
améliorer notre compréhension de la régulation des gènes au cours
du processus physiopathologique d’agression allo-immune et, plus
largement, de réponse rénale à l’infl ammation
Mots-clés : MicroARN – Régulation – Infl ammation – Rejet.
The implication of microRNAs as master regulators of innate
and adaptive immune responses has been recently identifi ed.
We were among the fi rst to introduce microRNAs in the fi eld
of transplantation immunobiology. Our work suggested
that microRNAs might be considered as biomarkers of acute
rejection of the renal allograft, but may also improve our
understanding of the regulation of gene expression during
immune injury
Keywords: MicroRNA – Regulation – Infl ammation – Rejection.
Micro-ARN et inflammation :
ce que l’étude du rejet aigu
du greffon rénal peut nous apprendre
MicroRNAs and inflammation: what we learnt from the study
of acute rejection of the renal allograft
Lucile Amrouche*, Clémentine Rabaté*, Dany Anglicheau*
* Inserm U845 ; université
Paris-Descartes, Sorbonne
Paris Cité ; service de
transplantation rénale et
de soins intensifs, hôpital
Necker, AP-HP, Paris.
Micro-ARN et régulation
de l’expression des gènes
Les micro-ARN (miARN) représentent une classe abon-
dante d’ARN naturels non codants de petite taille qui
jouent un rôle fondamental dans la régulation de
l’expression des gènes. Même si le premier miARN a
été décrit en 1993, ce n’est qu’au cours des dernières
années que la reconnaissance de leur rôle important
dans la régulation des gènes a conduit à un intérêt
croissant pour leur caractérisation (1).
Synthèse des micro-ARN
Les gènes des miARN peuvent être codés par des
unités transcriptionnelles indépendantes, dans des
clusters polycistroniques ou dans les introns de
gènes codant pour des protéines (2). Les miARN
sont d’abord transcrits par une ARN polymérase II
dans le noyau, sous la forme de longs précurseurs
(primary miRNA [pri-miARN]) qui sont alors clivés en
un produit intermédiaire, appelé pré-miARN (precursor
miRNA), par l’enzyme nucléaire Drosha, une ribonu-
cléase spécifi que des ARN doubles-brins (fi gure) [3, 4].
Indépendamment de Drosha, une fraction des pré-
miARN d’origine intronique peut aussi être générée
par l’action combinée du splicéosome et d’une enzyme
appelée lariat-debranching enzyme (LDBR) [6]. Le pré-
miARN est un ARN d’environ 70 nucléotides, replié
en tige-boucle imparfaite par complémentarité de
bases entre la première moitié et la deuxième moitié
de sa séquence. Le pré-miARN est ensuite transporté
dans le cytoplasme via un mécanisme dépendant
de l’exportine-5, où il est clivé par une enzyme de la
famille Dicer qui le sépare de la boucle, pour libérer
un petit ARN double-brin de 19 à 25 nucléotides : dans
ce petit duplex, le futur miARN est imparfaitement
apparié à une molécule similaire (petit ARN, long
lui aussi d’une vingtaine de nucléotides). Au cours

Le Courrier de la Transplantation - Vol. XII - n° 1 - janvier-février-mars 2012 23
Micro-ARN et infl ammation :
ce que l’étude du rejet aigu du greff on rénal peut nous apprendre
de la dernière étape de la maturation du miARN, ce
duplex est ouvert ; un des brins de cette molécule
s’associe avec une protéine de la famille Argonaute,
formant ainsi le complexe RISC (RNA-Induced Silencing
Complex), alors que le fragment complémentaire est
dégradé.
Mécanisme d’action des micro-ARN
Quand le complexe RISC identifi e l’ARNm cible com-
plémentaire ou partiellement complémentaire de la
séquence du miARN, il inhibe l’expression du gène
par répression de la traduction ou par dégradation de
l’ARNm. Les miARN ne sont souvent que partiellement
complémentaires de leurs ARN cibles. Si la complémen-
tarité est parfaite entre le miARN et l’ARNm, l’ARNm cible
est clivé et dégradé. Si la complémentarité est impar-
faite, le mécanisme conduisant à l’inhibition de l’expres-
sion du gène est encore discuté, et pourrait impliquer
l’inhibition de la traduction au niveau de l’initiation
ou de l’étape d’élongation, la dégradation rapide de la
protéine nouvellement synthétisée, la séquestration de
l’ARNm dans des structures cytoplasmiques de stockage
et de dégradation des ARNm appelées corps P (proces-
sing bodies), et/ou la dégradation de l’ARNm (7). Les
sites de fi xation des miARN sont généralement situés
vers l’extrémité 3’ non traduite (UnTranslated Region
[UTR]) des ARNm cibles.
Fonctions des micro-ARN
L’impact de la régulation de l’expression des gènes
par les miARN semble particulièrement vaste : des
centaines de miARN ont été clonés et des milliers ont
été prédits par analyse bio-informatique (8). Il a été
montré qu’un seul miARN est directement responsable
de la répression de centaines de protéines et régule
le niveau de milliers d’autres (9). Les miARN seraient
capables de réguler au moins un tiers de l’ensemble du
génome humain. On prédit également par analyse bio-
informatique que la plupart des miARN sont capables
de réguler des dizaines de gènes cibles. Enfi n, de nom-
breux gènes peuvent avoir de multiples sites de fi xa-
tion de miARN, qui représentent des cibles pour un
ou plusieurs miARN.
Les miARN semblent jouer un rôle clé dans des pro-
cessus biologiques divers comme le développe-
ment (10), la prolifération cellulaire, la diff érenciation,
l’apoptose, l’oncogenèse (11)… En particulier, les miARN
sont apparus très impliqués dans le développement
et l’homéostasie des cellules de l’immunité (lire ci-
dessous). La dérégulation de l’expression de miARN
a aussi été associée à plusieurs cancers et à d’autres
types de maladie (12).
Implication des micro-ARN
dans le système immunitaire
Des données récentes suggèrent le rôle des miARN
dans la régulation du développement des cellules de
l’immunité et dans la modulation des immunités innée
et adaptative (tableau, p. 24) [14, 15].
Micro-ARN et développement et maturation
des cellules de l’immunité
Des études récentes montrent le rôle des miARN dans le
développement et la maturation lymphocytaire (16, 17)
et dans la différenciation des lignées hématopoïé-
tiques (18). Par exemple, la caractérisation du réper-
toire de miARN de cellules lymphocytaires T à diff érents
Figure. Biogenèse des miARN
(4)
. Les miARN sont transcrits par une ARN polymérase II dans
le noyau sous la forme de primary miRNA (pri-miARN), puis clivés en precursor miRNA (pré-
miARN) par un complexe protéique, le Microprocessor complex, comportant la ribonucléase
de type III Drosha et son cofacteur, la protéine DiGeorge syndrome Critical Region gene 8
(DGCR8). Le pré-miARN, replié en tige-boucle imparfaite par complémentarité de bases, est
transporté dans le cytoplasme par l’exportine-5. Il est ensuite clivé par un complexe protéique
comportant la ribonucléase de type III Dicer, la protéine TAR RNA-Binding Protein (TRBP) ou
la Protein
kinase R ACTivator (PACT) et une des protéines Argonaute 1 à 4, libérant 1 ARN
double-brin d’environ 22 nucléotides. Ce duplex est alors ouvert, un des brins s’associant à la
protéine Argonaute pour former le complexe RISC (RNA-Induced Silencing Complex), alors que
le fragment complémentaire appelé miARN* est le plus souvent dégradé. Le miARN comporte
en son extrémité 5’ la
seed sequence
, séquence clé qui va servir de guide au complexe RISC
afi n d’identifi er la séquence complémentaire présente à l’extrémité 3’ UTR de l’ARN messager
(ARNm) cible du miARN. RISC inhibe alors l’expression du gène par répression de la traduction
ou par dégradation de l’ARNm
(5)
.
Ribosome
ARNm cible ARNm cible
Exportine-5
Cytoplasme
Noyau
ADN génomique
Pri-miARN
Transcription
ARN polymérase II
Drosha/DGCR8
Dicer/Ago1-4/
TRBP ou PACT
Inhibition
de la traduction
Pré-miARN
AAAn
Homologie
de séquence
partielle
Homologie
de séquence
parfaite
Complexe
RISC
AAAn
Ribosome
Déstabilisation
de l’ARNm

Le Courrier de la Transplantation - Vol. XII - n° 1 - janvier-février-mars 2012
24
Dossier thématique
Biomarqueurs
stades de maturation a révélé des modifi cations dyna-
miques défi nissant une signature de miARN spécifi que
de chaque stade (19). En particulier, le miARN miR-181
est élevé au stade de cellules double-positives, quand
les thymocytes exprimant à la fois le CD4 et le CD8
subissent la sélection positive et la sélection négative,
ce qui suggère le rôle de miR-181 dans ce processus.
L’étude du profi l d’expression des miARN dans les cel-
lules T naïves, eff ectrices et mémoires a montré que
l’expression de miARN est modifi ée au cours de la dif-
férenciation des cellules T (20). Il a aussi été observé
que les cellules T régulatrices sont caractérisées par
un profi l d’expression des miARN distinct de celui des
lymphocytes T CD4+ conventionnels (21).
Micro-ARN et immunité innée
Une étude récente utilisant la technologie des microar-
rays sur des macrophages activés de souris a caractérisé
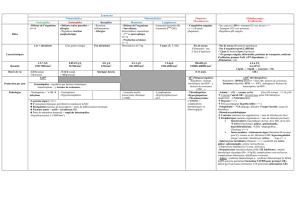
Tableau. MicroARN, gènes cibles et régulation de l’immunité (13).
Micro-ARN Gène(s) cible(s) Fonction et processus régulés
let-7e TLR4 Régulation de l’immunité innée
miR-9 NF-κB1 Régulation de la réponse à l’infl ammation
miR-16 TNFα Fixation à des motifs ARE des régions 3’ non traduites; induit la dégradation du TNFα
miR-17-5p RUNX1 Inhibition de la prolifération, de la diff érenciation et de la maturation des monocytes
miR-17~92 (cluster) BIM, PTEN Régulation de la transition de prolymphocytes à prélymphocytes des lignées B et T ;
promotion de la survie des cellules B et T
miR-20a AML-1 Inhibition de la prolifération, de la diff érenciation et de la maturation des monocytes
miR-21 PTEN, PDCD4, IL12A Expression augmentée dans les macrophages en réponse à l’infl ammation; induit par les TLR;
régule négativement l’activation macrophagique
miR-32 PFV1, ORF2 Inhibition de la réplication de PFV1 en ciblant le génome viral
miR-34 JAG1, WNT1, FOXP1 Régule la diff érenciation des cellules dendritiques d’origine myéloïde
miR-106a AML-1 Inhibition de la prolifération, de la diff érenciation et de la maturation des monocytes
miR-122 – Requis pour la réplication du VHC
miR-121/miR-122 KIT Régule la prolifération des cellules souches et des cellules progénitrices
miR-125b TNFα Diminue au cours de la réponse à l’infl ammation, induisant ainsi la production de TNFα
miR-126 HOXA9, PLK2 Induit l’expansion des cellules progénitrices
miR-132 – Régule la réponse immune aux infections bactériennes ; impliqué dans la voie de signalisation de CREB
miR-142 AC9 Réprimé par FOXP3, conduisant à une augmentation de la production d’AMPc et aux fonctions
suppressives des cellules T régulatrices
miR-146a TRAF6, IRAK1, IRAK2 Augmenté dans les macrophages et les cellules épithéliales en réponse à l’activation de TLR2, 4 et 5
ou à l’exposition au TNFα ou à l’IL-10; régule la réponse immune aux infections bactériennes
miR-150 Myb Régule la production de lymphocytes B matures ; régule la transition de pro- à pré-B;
régule l’activation des lymphocytes T
miR-155
PU.1, MAF, SHIP1, AID,
SOCS1, BACH1, CEBPB,
CSFR, TAB2, JARID2
Régule la réponse immune aux infections bactériennes et virales ; induit par la signalisation des TLR ;
régule TNFα ; requis pour les fonctions lymphocytaires normales, la réponse des centres germinatifs,
la commutation de classe, la génération de plasmocytes, la polarisation Th1/Th2, le développement
thymique des cellules T régulatrices, la prolifération des granulocytes et des monocytes
miR-181a DUSP5, DUSP6, SHP2,
PTPN22, BCL-2, CD69
Régule le développement des lymphocytes B et T ; module la sensibilité des lymphocytes T aux antigènes
en régulant les niveaux d’expression de plusieurs phosphatases impliquées dans la signalisation du TCR
miR-181b AID Impliqué dans la commutation de classe des lymphocytes B
miR-196b Famille HOX Rôle fonctionnel en modulant l’homéostasie des cellules souches et l’engagement dans les voies
de diff érenciation cellulaire
miR-223 MEF2C Régulation de la granulopoïèse
miR-326 ETS1 Promotion du développement des cellules T
H
17
miR-424 NFIA, SPI1 Impliqué dans la diff érenciation et la maturation des monocytes
ARE : AU-rich elements ; TLR : Toll-Like Receptor.

Le Courrier de la Transplantation - Vol. XII - n° 1 - janvier-février-mars 2012 25
Micro-ARN et infl ammation :
ce que l’étude du rejet aigu du greff on rénal peut nous apprendre
le miARN miR-155 comme la voie commune de plusieurs
types de médiateur de l’infl ammation (22), et 3 miARN,
miR-146a, miR-132 et miR-155, sont régulés en réponse
à la stimulation par des endotoxines (23). La fonction
des miARN dans les macrophages a été revue (24).
Micro-ARN et immunité adaptative
Des miARN ont été impliqués dans la sensibilité des
lymphocytes T aux pathogènes. Les cellules T qui surex-
priment miR-181 présentent une activation plus forte
de la cascade de signalisation du TCR en réponse à des
complexes MHC-peptide de faible affi nité (25).
Le phénotype de souris invalidées pour le gène miR-155
met en évidence le rôle complexe joué par miR-155
sur diff érents aspects de la réponse immune adapta-
tive. L’absence de miR-155 conduit à une altération
complexe de la réponse immune, évaluée par des
tests fonctionnels sur les lymphocytes B et T et les
cellules dendritiques, et à l’échec d’acquisition d’une
immunité protectrice contre des bactéries patho-
gènes (26). D’autres études montrent le rôle de miARN
au cours de la réponse à des infections bactériennes
ou virales (23, 26-28).
Micro-ARN et rejet du greff on rénal
L’implication des miARN dans l’homéostasie des cel-
lules de l’immunité a conduit à envisager leur impli-
cation au cours de la réaction allo-immune. Le rejet
aigu cellulaire du greff on rénal est caractérisé par
une infi ltration du greff on par des cellules mononu-
cléées alloréactives, dont le caractère agressif aboutit
à la dysfonction du greff on. L’implication des miARN
n’étant pas connue dans ce contexte, nous avons émis
l’hypothèse que le profil d’expression des miARN
pouvait être altéré au cours du rejet aigu d’allogreff e
rénale (29). Nous avons donc entrepris l’étude du profi l
d’expression des miARN dans des biopsies de greff ons
rénaux atteints ou non de rejet aigu cellulaire (29). Le
profi l d’expression global de 365 miARN a été analysé
dans 3 biopsies de greff ons rénaux atteints de rejet
aigu et dans 4 biopsies normales de greff ons rénaux
de patients à fonction rénale stable, utilisés comme
contrôles. Une étude par clustering hiérarchique non
supervisé de ces profi ls d’expression permettait de
séparer correctement les 2 groupes, suggérant une
modifi cation particulière du profi l de miARN au cours
du rejet aigu. Une analyse supervisée cherchant à
isoler les miARN dont l’expression était signifi cative-
ment diff érente dans les 2 groupes a permis de mettre
en évidence une signature d’expression de miARN
au cours du rejet aigu cellulaire du greff on rénal. Les
niveaux d’expression de 53 miARN étaient signifi ca-
tivement diff érents dans les 2 groupes de biopsies,
incluant 43 miARN moins exprimés (let-7c, miR-10a,
miR-10b, miR-125a, miR-200a, miR-30a-3p, miR-30b,
miR-30c, miR-30e-3p, miR-32, etc.) et 10 miARN plus
exprimés (miR-142-5p, miR-142-3p, miR-155, miR-
223, miR-146b, miR-146a, miR-342, miR-21, miR-650,
miR-525-5p) au cours du rejet. Cette signature a pu
être confi rmée sur des échantillons biopsiques indé-
pendants. Des analyses par courbes ROC (Receiver
Operating Characteristic) ont montré que le niveau
d’expression de miARN comme miR-142-5p ou miR-155
peut constituer un bon biomarqueur diagnostique de
rejet aigu cellulaire du greff on rénal, caractérisé par
une sensibilité et une spécifi cité de, respectivement,
100 % et 95 %.
Des analyses complémentaires ont montré que l’ex-
pression de plusieurs des miARN signifi cativement plus
exprimés (miR-142, miR-155 et miR-223) était corrélée
à l’expression du marqueur lymphocytaire T CD3, ce
qui suggère que ces miARN provenaient essentielle-
ment des cellules immunes infi ltrant le greff on. Des
expériences menées in vitro ont également montré
que l’expression de ces miARN était régulée par l’état
d’activation des cellules mononuclées, ce qui laisse
penser que les variations d’expression observées
n’étaient pas liées uniquement au degré d’infi ltra-
tion du greff on mais aussi au degré d’activation des
cellules. De façon similaire, l’expression de plusieurs
des miARN signifi cativement moins exprimés dans
les biopsies au cours du rejet aigu était corrélée à
l’expression du marqueur épithélial tubulaire NKCC2,
ce qui suppose que ces miARN provenaient essen-
tiellement des cellules parenchymateuses rénales.
De plus, des cellules épithéliales tubulaires rénales
en culture exposées à des cytokines pro-infl amma-
toires, mimant in vitro l’environnement infl ammatoire
du rejet, présentaient une altération de leur profi l
d’expression des miARN, suggérant l’implication de
ces derniers dans les altérations tubulaires induites
par l’infl ammation (29).
Régulation des micro-ARN
par l’infl ammation
Ces premiers résultats, suggérant une régulation de
l’expression des miARN tubulaires rénaux au cours
du rejet, ont conduit à suspecter une régulation
des miARN par l’inflammation dans les cellules
tubulaires.

Le Courrier de la Transplantation - Vol. XII - n° 1 - janvier-février-mars 2012
26
Dossier thématique
Biomarqueurs
Plusieurs stimuli infl ammatoires sont en eff et capables
de réguler l’expression de miARN (30). Des facteurs
transcriptionnels peuvent réguler des gènes porteurs
de séquences de miARN, modulant ainsi l’expression
de ces miARN. Dans le contexte de l’infl ammation, des
ligands de TLR, des antigènes ou des cytokines peuvent
faire varier l’expression de miARN par la régulation de
facteurs transcriptionnels spécifi ques. Dans les cellules
de l’immunité, plusieurs stimuli pro-infl ammatoires
peuvent induire des miARN. Par exemple, le ligand de
TLR4, le lipopolysaccharide (LPS), induit, dans la lignée
monocytaire humaine THP1, diff érents miARN dont
miR-155, miR-146 et miR-132, et l’interleukine 1β (IL-1β)
induit miR-146a (23). La phytohémagglutinine induit
miR-155 dans les cellules mononucléées circulantes
humaines (29).
Même si les données de la littérature sont beaucoup
plus limitées, le même type de régulation semble égale-
ment exister dans des cellules épithéliales. Par exemple,
la stimulation par l’IL-1β de cellules épithéliales alvéo-
laires humaines conduit à une augmentation du niveau
d’expression de miR-146a (31).
Implication des micro-ARN
dans les modifi cations épithéliales
phénotypiques induites par l’infl ammation
Au cours du rejet aigu, l’inflammation induit des
modifi cations phénotypiques des cellules résidentes
parenchymateuses du greff on. Les cellules tubulaires
activées produisent des cytokines, des chimiokines
et des molécules de costimulation en réponse à
l’infi ltration lymphocytaire (32). Par conséquent, les
cellules épithéliales activées régulent positivement
ou négativement l’activité destructrice des cellules
immunitaires alloréactives. D’autre part, l’infl amma-
tion peut également conduire à des modifi cations
phénotypiques suggestives de transition épithélio-
mésenchymateuse (33). Les mécanismes de régulation
mobilisés au niveau unicellulaire sont mal connus,
mais des données récentes suggèrent que les miARN
pourraient être impliqués, en ciblant des transcrits clés
de la signalisation intracellulaire et/ou en modifi ant
les molécules de surface exprimées par les cellules
épithéliales en réponse à l’infl ammation (34).
Dans les cellules épithéliales alvéolaires humaines qui
présentent une augmentation de miR-146a après sti-
mulation par l’IL-1β, la transfection de miR-146a avant
l’exposition aux cytokines réduit la synthèse d’IL-8 et
de RANTES par ces cellules, ce qui indique que miR-
146a est associé à la régulation de la synthèse de
médiateurs de l’infl ammation au cours de la réponse
immune précoce dans ce modèle (31). Les miARN ont
aussi été impliqués dans la régulation de l’expression
de protéines membranaires immunorégulatrices. Le
miARN miR-513 régule l’expression de la molécule de
costimulation B7-H1 induite par l’IFN-γ dans des cellules
épithéliales biliaires (35).
Ces données émergentes suggèrent l’implication de
miARN dans la réponse des cellules épithéliales à des
stimuli pro-infl ammatoires.
Implication des micro-ARN
dans la réponse épithéliale tubulaire
rénale à l’infl ammation
Nos résultats, obtenus sur des biopsies de greff ons
rénaux, associés aux données émergentes de la litté-
rature (cf. chapitre ci-dessus), suggéraient la possibilité
d’une régulation des miARN par l’infl ammation dans
les cellules épithéliales rénales (29). Nous avons donc
entrepris de tester l’hypothèse selon laquelle, dans le
contexte du rejet aigu d’allogreff e, l’infl ammation de
l’organe greff é pourrait aff ecter l’expression de miARN
non seulement dans les cellules infi ltrantes activées
mais aussi dans les cellules résidentes parenchyma-
teuses exposées à un milieu pro-infl ammatoire.
Une étude in vitro a donc été entreprise pour appré-
hender les conséquences de l’infl ammation sur l’ex-
pression des miARN tubulaires et pour déterminer si
celles-ci ont des conséquences physiopathologiques sur
les lésions des cellules cibles. Nos résultats préliminaires
réalisés in vitro ont montré que l’exposition de cellules
épithéliales tubulaires rénales de la lignée HK-2 à du
surnageant de cultures de cellules mononucléées pré-
alablement activées par la phytohémagglutinine induit
une modifi cation du profi l d’expression de miARN, avec
notamment une augmentation de l’expression de miR-
146a. L’IL-1β et les ligands des TLR2 et 4 reproduisent
cette induction de miR-146a dans les cellules tubulaires
rénales et cette stimulation est en partie inhibée par
la répression de la voie NF-κB, suggérant son impli-
cation dans la régulation de miR-146a. Une analyse
transcriptomique montre que l’expression de l’IL-8 et
de plusieurs chimiokines est réduite par la surexpres-
sion de miR-146a : il y aurait un rôle de rétrocontrôle
négatif de miR-146a sur la voie d’activation de NF-κB,
l’expression de miR-146a limitant les conséquences
des stimuli infl ammatoires sur l’activation de ce facteur
transcriptionnel. Des études fonctionnelles sont en
cours pour identifi er les mécanismes de cet eff et régu-
lateur de la voie NF-κB.
 6
6
1
/
6
100%