Biomarqueurs du cancer bronchique non à petites cellules : du concept à la pratique clinique MISE AU POINT

210 | La Lettre du Cancérologue • Vol. XVII - n° 5 - mai 2008
MISE AU POINT
Biomarqueurs du cancer bronchique
non à petites cellules :
du concept à la pratique clinique
Biomarkers for non-small-cell lung cancer:
from concept to molecular analysis-based treatment strategies
M. Pérol*, D. Arpin*, J.C. Soria**
* Unité d’oncologie thoracique,
hôpital de la Croix-Rousse, Lyon.
** Département de médecine,
institut Gustave-Roussy, Villejuif.
L
e cancer broncho-pulmonaire représente
toujours la première cause de mortalité par
cancer en France, en étant responsable de près
de 28 000 décès chaque année. Malgré l’amélioration
des traitements classiques du cancer bronchique
que représentent la chirurgie, la radiothérapie et
la chimiothérapie, les progrès enregistrés demeu-
rent modestes, avec une certaine stagnation des
données de survie, toutes formes confondues. Durant
la dernière décennie, les progrès dans la connaissance
de la biologie des cellules cancéreuses ont été cepen-
dant considérables, catalysant ainsi le passage des
traitements classiques du cancer aux thérapeutiques
ciblées. Ces progrès permettent d’envisager le déve-
loppement de marqueurs biologiques susceptibles
d’intervenir à tous les stades de la prise en charge des
patients atteints de cancer bronchique ou suspectés
de l’être, depuis le diagnostic des lésions précancé-
reuses ou des formes précoces jusqu’à l’individua-
lisation de la thérapeutique pour chaque patient.
Nous exposerons dans cette revue l’état actuel et
les perspectives de l’utilisation des biomarqueurs
dans la prise en charge des cancers bronchiques non
à petites cellules (CBNPC).
Définition, objectifs
et différentes catégories
de biomarqueurs
Au sens strict du terme, un biomarqueur est un état
physique, biologique ou moléculaire identifiant un
état physiologique spécifique. En cancérologie,
un biomarqueur peut se définir comme un para-
mètre biologique identifiant une entité nosologique
distincte ou un sous-groupe de pronostic homogène
au sein d’une population de patients atteints d’une
maladie donnée, ou permettant de prédire la réponse
à un traitement donné.
Dans le domaine du cancer bronchique, le champ
d’application des biomarqueurs est très vaste :
marqueurs de risque de développer un cancer bron-
chique au sein de la population générale ou à risque
(tabagisme), marqueurs de dépistage des lésions
précancéreuses et de diagnostic des formes précoces
accessibles à un traitement curatif, éventuellement
en combinaison avec les techniques endoscopiques
ou radiologiques, biomarqueurs tumoraux pronos-
tiques permettant l’évaluation de la probabilité de
récidive ou de progression tumorale, par exemple
après exérèse chirurgicale d’un cancer bronchique
avant tout traitement adjuvant, biomarqueurs
prédictifs de la réponse, de la résistance ou de la
tolérance à un traitement (chimiothérapie, théra-
peutique ciblée), marqueurs de monitoring post-
traitement utilisés pour la détection de la récidive
tumorale (surveillance des cancers opérés). L’iden-
tification de biomarqueurs fait également partie
intégrante du développement des thérapeutiques
ciblées ; cette étape paraît en effet fondamentale,
ayant pour but de caractériser au mieux les patients
susceptibles de bénéficier de ces traitements ou
d’individualiser des critères biologiques de réponse
au traitement (marqueurs pharmacodynamiques)
[1]. L’absence de sélection adéquate de la population
cible d’une thérapeutique peut en effet expliquer en
LK5-NEW + pub.indd 210 27/05/08 17:07:49

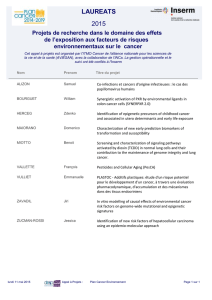
Suspicion de cancer
bronchique
Diagnostic histologique Classe
pronostique
Traiter
ou ne pas traiter ?
Prédictifs
Quel traitement ?
Cibles
Risque
métastatique
Génomique
ERCC1
BRCA1
RRM1
Oncogenèse
EGFR
K-ras
p53
Rb
mTor
Thérapeutiques
TS
ERCC1
RRM1
BRCA1
β-tubuline
Toxicité
Épidermoïde
Grandes cellules
Adénocarcinome
Classe génomique
Prélèvement
Biomarqueurs
Figure. Utilisations potentielles des biomarqueurs dans le CBNPC.
La Lettre du Cancérologue • Vol. XVII - n° 5 - mai 2008 | 211
Résumé
partie certains échecs de développement en phase III
de traitements ciblés (2). La figure illustre l’utilisa-
tion potentielle des biomarqueurs en cancérologie
thoracique dans la perspective d’une individualisa-
tion thérapeutique.
L’un des principaux domaines d’application des
biomarqueurs est la prédiction de l’efficacité de
la chimiothérapie ou d’une thérapeutique ciblée.
La valeur prédictive d’un biomarqueur est souvent
confondue ou intriquée avec sa valeur pronostique.
Un facteur pronostique est une caractéristique du
patient ou de la tumeur prédisant le devenir du
patient (survie) indépendamment du traitement ;
il s’agit donc d’un marqueur de l’histoire naturelle
de la maladie qui définit l’effet de la tumeur sur le
patient. Un facteur prédictif est un facteur clinique
ou biologique associé à l’efficacité d’un traitement
donné ; il définit l’effet du traitement sur la tumeur.
Il implique donc un bénéfice différent du traitement
selon qu’il est présent ou non. En termes statisti-
ques, il peut se définir par une interaction entre le
bénéfice thérapeutique et le statut du biomarqueur
(3). La détermination de la signification prédictive
ou pronostique d’un marqueur nécessite une étude
randomisée avec un groupe contrôle ; en effet, la
mise en évidence d’un devenir différent de deux
sous-groupes définis par la présence ou l’absence
d’un biomarqueur au sein d’une cohorte de patients
recevant tous le traitement évalué ne suffit pas pour
différencier la valeur prédictive ou pronostique de ce
biomarqueur. Cependant, l’interprétation des études
est parfois rendue difficile par l’intrication d’une
valeur prédictive et d’une signification pronostique
du même marqueur.
Évolution actuelle des concepts
et de la technologie
liés aux biomarqueurs :
des marqueurs tumoraux
sériques à la génomique
Les biomarqueurs peuvent être recherchés dans
les tissus normaux ou tumoraux, dans le sérum, le
plasma, le sang, le liquide pleural ou péritonéal,
avant ou après le traitement. Les premiers biomar-
queurs utilisés ont été les marqueurs tumoraux
L’amélioration de la compréhension des mécanismes de la carcinogenèse et de la progression tumorale dans les cancers
broncho-pulmonaires a permis l’identification de gènes, de profils d’expression et de voies de signalisation intracellulaires
constituant la base du développement de biomarqueurs pronostiques ou prédictifs de l’efficacité des traitements tradi-
tionnels ou ciblés. L’étude du profil d’expression génique des tumeurs bronchiques pourrait conduire à une taxonomie
moléculaire de ces tumeurs en rapport avec les différents mécanismes d’oncogenèse. Plusieurs marqueurs apportent des
informations pronostiques indépendantes des facteurs cliniques classiques pour les cancers bronchiques non à petites
cellules opérés, comme l’expression d’ERCC1, de RRM1 ou de BRCA1, ou certains profils d’expression génique. D’autres
facteurs interviennent par leur capacité à prédire l’efficacité d’une thérapeutique donnée, comme les mutations de K-ras
ou de l’EGFR pour les inhibiteurs de tyrosine kinase de l’EGFR, ERCC1 pour les sels de platine, RRM1 pour la gemcitabine,
la β-tubuline de classe III pour la vinorelbine ou les taxanes. De nombreux obstacles, dont la nécessité d’une validation
prospective, subsistent néanmoins avant que l’on ne voie apparaître ces marqueurs en pratique clinique.
Mots-clés
Cancer bronchique
non à petites cellules
Biomarqueurs
Génomique
Summary
Improved understanding in
carcinogenesis and biology
of tumour progression in
lung cancer has resulted in
the identification of genes,
gene expression profiles and
signalling pathways leading to
new prognostic or predictive
biomarkers for usual treat-
ment as chemotherapy or
targeted therapies. Studies of
gene expression profiles might
define a new molecular taxo-
nomy of lung cancer. Several
markers give prognostic
information independent of
standard clinical parameters
in non-small-cell lung cancer
after surgical resection as
ERCC1, RRM1, BRCA1 or some
gene expression profiles. Some
markers with a predictive value
can impact therapeutic efficacy
as K-ras or EGFR mutations for
EGFR tyrosine-kinase inhibitors,
ERCC1 for platinum, RRM1 for
gemcitabine, class III β-tubulin
for vinorelbine and taxanes.
However, there are still many
barriers before implementation
of these technologies into the
clinic, particularly the need of
a prospective validation.
Keywords
Non-small-cell lung cancer
Biomarkers
Genomics
LK5-NEW + pub.indd 211 27/05/08 17:07:49

212 | La Lettre du Cancérologue • Vol. XVII - n° 5 - mai 2008
Biomarqueurs du cancer bronchique non à petites cellules :
du concept à la pratique clinique
MISE AU POINT
sériques, dont l’intérêt clinique, que ce soit pour
le dépistage ou le monitoring thérapeutique, s’est
révélé trop faible. L’avènement des techniques
immunohistochimiques, de réalisation facile mais
de reproductibilité plus aléatoire, a permis la mise
en évidence semi-quantitative de l’expression de
protéines par la tumeur, avec la perspective d’une
standardisation et d’une généralisation plus aisées
que les techniques plus sophistiquées. Les progrès
réalisés dans la connaissance des modifications géno-
miques et moléculaires impliquées dans le processus
de carcinogenèse et de croissance tumorale ont
permis l’individualisation de biomarqueurs poten-
tiels, tant pour la détection précoce d’un cancer
que pour la personnalisation des thérapeutiques
(4). Actuellement, il est en effet techniquement
possible au niveau de la cellule tumorale d’évaluer
les modifications du génome en termes de contenu
ou de séquences d’ADN, la transcription génique
par la mesure des ARN messagers (ARNm) ou des
microARN, ainsi que la synthèse des protéines ou
des métabolites. Les techniques utilisées peuvent
s’intéresser à un gène cible en particulier, comme,
par exemple, le gène de l’epidermal growth factor
receptor (EGFR), en recherchant une amplification
génique (par hybridation in situ), son niveau d’ex-
pression par la mesure de son ARNm, des mutations
par séquençage ou un polymorphisme touchant un
nucléotide (single nucleotide polymorphism, SNP).
Les nouvelles technologies telles que les puces à
ADN à haut débit permettent désormais d’avoir
une véritable signature moléculaire de la tumeur,
avec la mise en évidence par l’hybridation géno-
mique comparative de gains ou de pertes de matériel
génétique, de mutations ou de polymorphismes de
nucléotides sur l’ensemble du génome, de l’expres-
sion simultanée de milliers de gènes tumoraux par
l’analyse du transcriptome (ARNm) [5], voire de
prendre en compte les modifications post-trans-
criptionnelles par l’analyse des protéines synthéti-
sées par la cellule tumorale grâce aux techniques de
protéomique. Cette caractérisation des tumeurs au
niveau moléculaire pose néanmoins des problèmes
importants de coût, de reproductibilité, d’analyse
statistique (interprétation d’un très grand nombre
de variables – allant parfois jusqu’à plusieurs milliers
de gènes pour chaque échantillon tumoral – chez un
nombre limité de patients) ; ces difficultés rendent
pour l’instant la perspective d’une utilisation clinique
assez lointaine (5).
D’une façon plus générale, la recherche de biomar-
queurs dans les tumeurs solides se heurte à plusieurs
obstacles : hétérogénéité tumorale spatiale avec
une expression différente des marqueurs entre la
tumeur primitive et ses métastases (6), hétéro-
généité temporelle avec une possible discordance
entre les biopsies tumorales faites au moment
du diagnostic initial et celles faites au moment
de la rechute au stade métastatique, difficultés à
réaliser de nouveaux prélèvements biopsiques chez
un patient en rechute, taille limitée des prélève-
ments en oncologie thoracique (à l’exception des
pièces opératoires), reproductibilité incertaine des
techniques utilisées pouvant résulter de différences
dans les conditions de prélèvement et de conserva-
tion de ceux-ci. Des technologies non invasives se
développent pour contourner la difficulté d’effec-
tuer des prélèvements tumoraux itératifs, comme
l’analyse des cellules tumorales circulantes ou
de l’ADN circulant ou l’analyse protéomique sur
le sérum. Par ailleurs, l’interprétation statistique
des études de biomarqueurs concerne souvent
des études rétrospectives, sans bras contrôle, ne
permettant pas de distinguer la valeur réellement
prédictive du marqueur étudié de son éventuelle
signification pronostique. La validation prospective
des nouveaux biomarqueurs du CBNPC n’en est qu’à
ses débuts pour certains d’entre eux (mutations de
l’EGFR, pharmacogénomie). Les résultats présentés
doivent être interprétés avec prudence à cause de
ces limitations.
Biomarqueurs
du cancer bronchique
non à petites cellules
Marqueurs tumoraux sériques
Des marqueurs onco-fœtaux, comme l’antigène
carcino-embryonnaire (ACE), ou des marqueurs de
différenciation, comme la neuron specific enolase
(NSE) ou le CYFRA 21-1, ont été étudiés dans le
CBNPC (ou le cancer à petites cellules pour la NSE) :
leur intérêt clinique est faible. Ces marqueurs sont
plus ou moins directement le reflet de la masse
tumorale à l’échelon individuel, avec une sensibilité
et une spécificité insuffisantes pour recommander
leur utilisation en pratique courante, que ce soit dans
le cadre du dépistage, du monitoring du traitement
ou de la surveillance post-thérapeutique (7). De
même, leur valeur pronostique ne semble pas indé-
pendante des autres variables cliniques pronostiques,
sauf peut-être pour le CYFRA 21-1 dans les cancers
épidermoïdes (8).
LK5-NEW + pub.indd 212 27/05/08 17:07:50

La Lettre du Cancérologue • Vol. XVII - n° 5 - mai 2008 | 213
MISE AU POINT
Marqueurs de l’EGFR
Mutations de l’EGFR
Le séquençage du gène de l’EGFR (9-11) a permis
d’individualiser des mutations somatiques hétéro-
zygotes situées sur le domaine tyrosine kinase (TKI)
du récepteur chez 80 à 90 % des patients répon-
deurs au gefitinib ou à l’erlotinib (exons 18 à 21).
Deux types de mutation représentent 85 à 90 % des
mutations décrites et sont corrélés à la sensibilité
des cellules tumorales mutées aux inhibiteurs de la
tyrosine kinase de l’EGFR (EGFR TKI) : délétions dans
l’exon 19 en position 746-750, substitution L858R
dans l’exon 21. Les tumeurs portant ces mutations
activatrices de l’EGFR paraissent dépendantes de
la voie de l’EGFR, dont l’inhibition par les EGFR TKI
entraîne une mort cellulaire par apoptose (9-11). La
fréquence de ces mutations semble plus importante
chez les femmes, en l’absence de tabagisme, pour
les adénocarcinomes et chez les patients d’origine
asiatique, ce qui est cohérent avec la relation existant
entre présence d’une mutation et réponse au traite-
ment. La probabilité de réponse aux TKI en présence
d’une mutation est de l’ordre de 70 à 80 % (12). On
constate cependant environ 10 % de répondeurs
parmi les patients dont la tumeur n’exprime pas
de mutations : celles-ci ne constituent donc pas le
déterminant unique de la réponse aux EGFR TKI.
L’analyse de la valeur des mutations de l’EGFR pour
prédire un bénéfice de survie sous EGFR TKI a fait
l’objet de plusieurs études rétrospectives portant
sur des séries de patients traités majoritairement
par géfitinib (13) ainsi que sur l’essai BR21 (14), qui
comporte un bras contrôle. Les résultats sont discor-
dants, probablement en raison de l’hétérogénéité des
séries, notamment en ce qui concerne la prévalence
des mutations, le matériel tumoral et les techniques
utilisées pour la recherche des mutations. Les essais
randomisés disposant d’un bras contrôle (placebo ou
chimiothérapie + placebo) suggèrent que les muta-
tions activatrices de l’EGFR ont une signification
pronostique favorable, quel que soit le traitement
administré – placebo, EGFR TKI en monothérapie,
chimiothérapie seule ou associée aux TKI (15, 16) ;
il est difficile dans ces études d’affirmer la valeur
prédictive des mutations concernant un gain de
survie sous EGFR TKI par rapport aux tumeurs dont
l’EGFR est de type “sauvage” (14, 15). Actuellement,
il n’est donc pas indiqué de sélectionner les patients
susceptibles de recevoir un traitement par TKI en
seconde ou troisième ligne sur la seule présence de
mutations de l’EGFR, car celles-ci (qui concernent
moins de 10 % des patients d’origine caucasienne et
environ 30 % des patients originaires d’Asie de l’Est)
◆
n’expliquent pas la totalité du bénéfice de survie
obtenu avec l’erlotinib, d’une part, et, d’autre part,
ne constituent pas le déterminant unique de la
réponse au traitement. La recherche de mutations
permettra en revanche de sélectionner une popu-
lation de patients dont la tumeur est extrêmement
sensible aux EGFR TKI, susceptibles de bénéficier de
leur utilisation en première ligne avec des taux de
réponse considérables (17). La validation prospective
en première ligne de cette sélection fondée sur la
présence de mutations du récepteur de l’EGFR fait
actuellement l’objet d’essais de phase III.
Augmentation du nombre de copies
du gène de l’EGFR
L’augmentation du nombre de copies du gène de
l’EGFR, évaluée par hybridation in situ (FISH), peut
résulter soit d’un degré élevé de polysomie, soit d’une
véritable amplification du gène (18). Les deux essais
randomisés ISEL (2) et BR21 (14), comparant un EGFR
TKI à un placebo, témoignent d’un bénéfice de survie
lié au géfitinib ou à l’erlotinib supérieur pour les
patients dont la tumeur contient un nombre de copies
élevé du gène de l’EGFR, par rapport aux patients
dont la tumeur est “FISH–”, pour lesquels il n’y a pas
de bénéfice de survie. Il existe probablement une
corrélation entre la présence de mutations et l’am-
plification du gène (19) ; l’incertitude quant au rôle
prédictif réel de l’amplification tient à l’absence de
distinction entre l’amplification des allèles sauvages
et celle des allèles mutés (12). Les tumeurs FISH+
(qui touchaient environ 30 % des patients de l’étude
ISEL) définissent une population cliniquement moins
“ciblée” que les mutations de l’EGFR (19) : sex-ratio
voisin de 1, présence de carcinomes épidermoïdes,
patients fumeurs ; l’augmentation du nombre de
copies du gène de l’EGFR définie par la positivité du
test FISH (18) pourrait ainsi constituer un marqueur
prédictif de bénéfice en termes de survie dans l’utili-
sation des EGFR TKI en seconde ou troisième ligne, car
elle caractérise à la fois les patients répondeurs et les
patients stabilisés par le traitement. Néanmoins, la
reproductibilité de ces résultats, obtenus par une seule
équipe, reste à démontrer, de même que l’application
en routine de cette technique délicate.
Expression de l’EGFR
évaluée par immunohistochimie
Le rôle prédictif d’un bénéfice de l’expression de
la protéine EGFR évaluée par immunohistochimie
a été étudié dans les deux études ISEL et BR21 (2,
15, 19). L’erlotinib procure, par rapport au placebo,
un bénéfice de survie lié aux TKI significatif pour les
◆
◆
LK5-NEW + pub.indd 213 27/05/08 17:07:50

214 | La Lettre du Cancérologue • Vol. XVII - n° 5 - mai 2008
Biomarqueurs du cancer bronchique non à petites cellules :
du concept à la pratique clinique
MISE AU POINT
patients dont la tumeur exprime l’EGFR en immuno-
histochimie, avec une médiane de survie à 10,7 mois
versus 3,8 mois avec le placebo (15) ; on note une
tendance semblable avec le géfitinib dans l’essai
ISEL (19). Il n’y a aucun bénéfice avec le géfitinib ou
l’erlotinib pour les patients dont la tumeur n’exprime
pas l’EGFR en immunohistochimie, mais le test d’in-
teraction entre l’effet de l’EGFR TKI et l’expression
de l’EGFR n’est positif que dans l’étude ISEL (19).
Une réserve importante concerne la variabilité des
résultats obtenus avec l’immunohistochimie selon
le type d’anticorps utilisé et l’échelle retenue pour
affirmer la positivité d’un échantillon (20). De ce fait,
il est difficile d’utiliser l’immunohistochimie comme
seul moyen de sélection des patients candidats à
une monothérapie de seconde ou troisième ligne
par erlotinib, les données actuelles ne permettant
pas d’exclure un bénéfice potentiel pour des tumeurs
négatives.
Marqueurs de résistance aux EGFR TKI
La présence d’une mutation de K-ras, souvent asso-
ciée au tabagisme, constitue un facteur de résistance
primaire au géfitinib et à l’erlotinib (21), l’activation
des voies d’aval étant alors assurée indépendamment
de l’activation de l’EGFR. Les mutations de K-ras et
celles de l’EGFR semblent ainsi s’exclure mutuel-
lement. La quasi-totalité des patients répondeurs
à l’erlotinib ou au géfitinib voient, au terme d’un
délai variable, leur tumeur progresser à nouveau
sous traitement. Plusieurs mécanismes expliquant
l’émergence d’une résistance acquise à l’erlotinib
ou au géfitinib ont été décrits. L’apparition ou la
sélection d’une seconde mutation dans l’exon 20
(mutation T790M) semble responsable d’environ
la moitié des résistances en restaurant la fonction-
nalité du récepteur par empêchement de l’ancrage
de l’erlotinib ou du géfitinib sur le site de fixation de
l’adénosine triphosphate (ATP) [22] ; une autre muta-
tion de l’exon 19 (D761Y) semble exercer le même
rôle. Le développement d’inhibiteurs irréversibles
de l’EGFR, comme l’HKI 272, actifs même en cas de
mutation T790M, permet d’espérer pouvoir traiter
efficacement ce type de rechute. Plus récemment,
l’amplification de l’oncogène MET, rétablissant la
fonctionnalité de la voie de pAkt par le biais d’Erb-B3
malgré le blocage de l’EGFR, a été décrite comme
un mécanisme isolé ou associé à la mutation T790M
de résistance (23). La détermination du mécanisme
de résistance pouvant avoir dans l’avenir des impli-
cations thérapeutiques, la réalisation d’un nouveau
prélèvement au moment de la rechute sera sans
doute nécessaire.
◆
Biomarqueurs pharmacogénomiques
La pharmacogénomie est l’étude des gènes qui
influencent l’activité ou la toxicité des drogues, en
particulier des cytotoxiques ; elle s’intéresse, par
exemple, à des gènes codant pour la cible enzy-
matique d’un cytotoxique comme la thymidylate
synthétase, pour le 5-fluoro-uracile ou le pémé-
trexed ou pour des enzymes intervenant dans les
mécanismes de réparation des dommages créés
par le cytotoxique sur l’ADN, comme l’excision
repair cross-complementary group 1 (ERCC1) pour
les lésions induites par les sels de platine. La phar-
macogénomie a ainsi pour objectif d’adapter le
traitement à chaque tumeur et à chaque individu
en prédisant la réponse à la chimiothérapie pour
en limiter les effets indésirables sévères ; un plus
grand nombre de patients pourrait ainsi bénéficier
en théorie d’un traitement avec une optimisation
du rapport bénéfice/risque.
ERCC1
ERCC1 est une enzyme appartenant à l’un des
systèmes de réparation de l’ADN appelé NER
(nucleotide excision repair), qui permet l’excision
des adduits formés par les sels de platine sur l’ADN et
la synthèse d’un brin d’ADN de remplacement. Une
forte capacité de réparation de l’ADN par la cellule
tumorale peut ainsi être corrélée à une résistance
au cisplatine. La valeur prédictive d’une résistance
au cisplatine de l’expression d’ERCC1 (ARNm) a été
testée de façon prospective dans les CBNPC avancés
lors d’une étude de phase III où une chimiothé-
rapie standard de type cisplatine docétaxel a été
comparée à un traitement adapté en fonction de
l’expression d’ERCC1 : cisplatine-docétaxel en cas
de faible expression d’ERCC1, schéma sans platine
docétaxel-gemcitabine en cas de forte expression
d’ERCC1 (24). Cette première étude prospective
a montré une augmentation significative des taux
de réponses avec l’adaptation de la chimiothérapie
selon le niveau d’expression d’ERCC1, sans impact
néanmoins sur la survie sans progression ni sur la
survie. La valeur pronostique et prédictive de l’ex-
pression d’ERCC1 en situation adjuvante, évaluée
par immunohistochimie, a été étudiée de manière
rétrospective sur les prélèvements de 761 patients
représentatifs de la population totale de l’essai
IALT, qui a démontré un bénéfice de survie avec la
chimiothérapie à base de cisplatine (25). La première
constatation est qu’une forte expression d’ERCC1 a
une valeur pronostique favorable chez les patients du
bras observation. À l’inverse, le bénéfice de la chimio-
◆
LK5-NEW + pub.indd 214 27/05/08 17:07:51
 6
6
 7
7
 8
8
 9
9
 10
10
1
/
10
100%