Biologie fondamentale C éi i i i C- les protéines Dominique Rainteau

Biologie fondamentale
Les Molécules du vivant
Première partie : de l’atome à la molécule . Philippe Karoyan
A
L’ J ëll M li h
Deuxième partie : les biomolécules du compartiment sanguin
A
-
L’
eau.
J
o
ëll
e
M
as
li
a
h
B- les ions du compartiment plasmatique Joëlle Masliah
C
éi i i i
D- les lipides Joëlle Masliah
C
-les prot
éi
nes Dom
i
n
i
que Ra
i
nteau
E- les glucides Jean Pierre Tresca
F- l’ox
yg
ène Jean Pierre Tresca
yg
1
DR 9 septembre 2010

Introduction : de la fonction à la structure
I. Les protéines du plasma
II. La structure tridimensionnelle d’une protéine
la sérum albumine
les immunoglobulines
Rappel
III. La chaîne polypeptidique: la liaison peptidique
IV. Les structures régulières: hélice a, feuillets b
V L li t d l h î l tidi
V
.
L
es rep
li
emen
t
s
d
e
l
a c
h
a
î
ne po
l
ypep
tidi
que
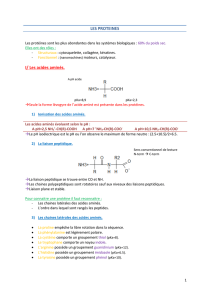
les amino acides
VI L
VI
L
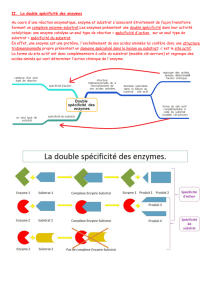
es enzymes
Mécanisme d’une réaction enzymatique
Le site actif
La protéolyse ménagée
La
protéolyse
ménagée
Les isoenzymes
DR 9 septembre 2010

Rappel
Hydrophile Æcapable d’établir des interactions stabilisantes avec les
molécules d’eau Æliaisons hydrogène H-
O
-H
H-O-H
molécule hydrophile
H
O
H
molécule
hydrophile
liaison hydrogène
liaison covalente

Molécule hydrophile Æcapable d’établir des interactions stabilisantes avec les ions Æ
liaisons ioni
q
ues
q
Cl
Na
Cl
Na
+
+
liaison ionique
Cl
Na
Cl
-
Na
+
+
liaison
ionique
-
+
+
-
COO
H
3
N
--
+
H
3
N
-
-
COO
-
+
+
COO
H
3
N
+
H
3
N
COO
liaison ioni
q
ue
q

Hydrophobe capable d’établir des interactions hydrophobes
radical CH3
acide gras (huile)
acide
gras
(huile)
interaction hydrophobe
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
1
/
35
100%