Les protéines

1
LES PROTEINES
Les protéines sont les plus abondantes dans les systèmes biologiques : 60% du poids sec.
Elles ont des rôles :
- Structuraux : cytosquelette, collagène, kératines.
- Fonctionnel : (nanomachines) moteurs, catalyseur.
I/ Les acides aminés.
pKa=8,9 pKa=2,3
Seule la forme lévogyre de l’acide aminé est présente dans les protéines.
1) Ionisation des acides aminés.
Les acides aminés évoluent selon le pH :
A pH=2,5 NH3+-CH(R)-COOH A pH=7 +NH3-CH(R)-COO- A pH=10,5 NH2-CH(R)-COO-
Le pH isoélectrique est le pH ou l’on observe le maximum de forme neutre : (2.5+10.5)∕2=6.5.
2) La liaison peptidique.
La liaison peptidique se trouve entre CO et NH.
Les chaines polypeptidiques sont rotatoires sauf aux niveaux des liaisons peptidiques.
Liaison plane et stable.
Pour connaitre une protéine il faut reconnaitre :
- Les chaines latérales des acides aminés.
- L’ordre dans lequel sont rangés les peptides.
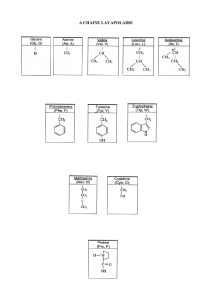
3) Les chaines latérales des acides aminés.
La proline empêche la libre rotation dans la séquence.
La phénylalanine est légèrement polaire.
La cystéine comporte un groupement thiol (pKa=8).
Le tryptophane comporte un noyau indole.
L’arginine possède un groupement guanidinium (pKa=12).
L’histidine possède un groupement imidazole (pKa=6.5).
La tyrosine possède un groupement phénol (pKa=10).
Sens conventionnel de lecture
N-term C-term
A pH acide

2
II/ La structure des protéines.
1) La structure primaire.

3
La structure primaire :
- Est la plus simple : enchainements de peptides (ex : aspartame).
- Correspond à la séquence.
Une protéine ne restera jamais sous cette forme, elle va en permanence avoir tendance à se
replier dans l’espace pour donner des niveaux de structure plus compliques.
Aspartate + phénylalanine + méthanol aspartame.
2) La structure secondaire.
La structure secondaire est due aux liaisons hydrogènes intramoléculaires qui permet les
repliements entre les CO et NH, la structure tridimensionnelle est en grande partie déterminée
par les interactions électrostatiques entre O- et H+.
Pauling et Corey ont répertorié toutes les structures secondaires.
a) L’hélice alpha :
L’hélice alpha:
- Est stable : les liaisons CO et NH pointe dans la direction de l’hélice. (1aa présente 100° de
rotation ; 1.5A).
- Elle définit une conformation du squelette polypeptidique qui se répète régulièrement.
- Droite est formée par des acides aminés L c’est la plus stable.
- Sa distorsion est induite par le solvant (H2O).
b) Les structures béta :
Les structures béta :
- Forment des liaisons hydrogènes entre différentes parties de leur chaine polypeptidique en
se tordant.
- Sont stables, les feuillets sont liés entre eux grâce à des liaisons hydrogènes et les
groupements R qui pointent en dehors des structures.
- Présentent un encombrement stérique limité.
Subissent une condensation pour donner ce que l’on appelle la structure tertiaire de la protéine.
Il faut donc comprendre que la structure primaire donne à différents endroits de la protéine des
structures secondaires, qui sont un arrangement répétitif et local de la chaine.
3) La structure tertiaire.
C’est dans cet état de structure que la protéine obtiendra sa structure tridimensionnelle et permet
leurs fonctionnalités.
C’est dans cette structure que l’on retrouve la majorité des protéines.

4
4) La structure quaternaire.
C’est un assemblage de plusieurs sous-unités (chaînes polypeptidiques = SU) pour former une
supermolécule.
III/ Mécanisme du repliement.
Tous types de liaisons interviennent dans le processus de repliement dont la majorité est les
liaisons covalentes (ponts disulfures).
L’information pour le repliement se trouve dans la structure de la protéine.
La dénaturation de la protéine se fait par :
- Modification du pH : change l’état de protonation des chaînes latérales.
- Chauffage de la protéine : mouvements thermiques dérangent les interactions.
- Utilisations de dénaturants :
Sels : déstabilisent les liaisons ioniques.
Les détergents : stabilisation de la protéine sous forme dénaturée.
- Les compétiteurs de liaisons hydrogènes.
- Les oxydants et réducteurs (réduisent les ponts disulfures).
Le repliement se fait thermodynamiquement : la plus basse énergie est adoptée.
IV/ Méthode d’étude des protéines.
Echantillon biologique homogénéisationhomogénatpurificationprotéine
1) Technique de purification des protéines.
Tampon de lyse:
- Isotonique (concentration en sel).
- pH physiologique affecte la structure des protéines (empêche action protéases).
- Oxydation (utilisation agents réducteurs).
- Protéase coupe les cellules (utilisation anti-protéase).
Méthode douce:
- Choc osmotique déstructuration de la membrane (sortie de l’eau de la cellule).
- Les détergents entre et solubilise la membrane.
- Le Dounce et le Potter éclatement des membranes.

5
Méthode forte :
- La French Press explosion des cellules par compression puis relâchement (50000 jet).
- La bombe a disruption éclatement des cellules par injection d’azote sous pression.
- Bead beater (billes de verres).
- La sonication destruction des cellules par ultrasons.
2) Reconnaissance des protéines.
a) La taille :
La centrifugation sépare les compartiments selon leur taille de par leur vitesse.
La dialyse sépare les protéines selon leur taille (garde les grosses).
Le tamisage moléculaire ou gel-filtration sépare les protéines par leur vitesse de descente.
b) La charge :
La chromatographie échangeuse d’ions (positive ou négative), sépare les protéines en fonction de
leur charge par un excès d’ions.
c) La fonction :
Chromatographie d’affinité : par ajouts de ligands spécifiques on peut en une seule étape éliminer
toutes les autres protéines.
3) Techniques d’études des protéines.
On a plusieurs méthodes afin d’étudier les protéines :
- Spectrophotométrie : mesure l’absorbance de la protéine a λ=280nm par absorption à
260nm des AA aromatiques (T, W, F).
- Le clivage protéique : dégrade la protéine par hydrolyse acide ou clivage enzymatique et
chimique et libère les AA.
- Le séquençage protéique : suppression des ponts disulfures et identification des AA N et C-
term en plus de la composition globale de la protéine.
- Electrophorèse : migration des protéines en fonction de leurs charges puis comparaison par
rapport à une courbe étalon.
- Spectrométrie de masse : Indentification de la masse des fragments de protéine ionisés.
1
/
5
100%