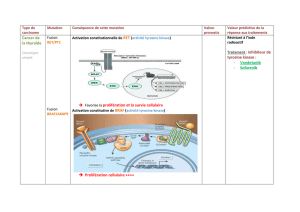
Lire l`article complet

Correspondances en Onco-Théranostic - Vol. I - n° 3 - juillet-août-septembre 2012
107
dossier thématique
ALK
Edimark SAS, 2, rue Sainte-Marie - 92418 Courbevoie Cedex - Tél. : 01 46 67 63 00 - Fax : 01 46 67 63 10
E-mail : [email protected] - www.edimark.fr
LE REGARD DE L’AVOCAT
1er Prix SPEPS 2011
de la meilleure
innovation numérique
M
e
Gilles Devers, avocat
et bénéficiez
d’un “libre accès
permanent” aux archives
et aux numéros en cours
des
et bénéficiez
d’un “libre accès
permanent” aux archives
et aux numéros en cours
des
ABONNEZ-VOUS !
2
O
publications du groupe
Réarrangements du gène ALK
dans les cancers du poumon
non à petites cellules
ALK gene rearrangement in non-small-cell lung carcinoma
Sylvie Lantuejoul*, Lénaïg Mescam-Mancini*,**, Anne McLeer-Florin**
* Département de
pathologie, pôle de
biologie et de pathologie,
hôpital Albert-Michallon ;
Inserm U823, institut
Albert-Bonniot, université
Joseph-Fourier, Grenoble.
** Plateforme de génétique
moléculaire des cancers,
pôle de biologie et de
pathologie, hôpital Albert-
Michallon, UMR-S 1036-
CEA, Grenoble, université
Joseph-Fourier, Grenoble.
RÉSUMÉ
Summary
»
Depuis 2004, la prise en charge des cancers du poumon a
subi une véritable révolution grâce à la découverte d’anomalies
génétiques pouvant être ciblées par des agents thérapeutiques.
Ainsi, dans les adénocarcinomes, après la mise en évidence des
mutations activatrices du gène de l’EGFR (Epidermal Growth Factor
Receptor), la découverte de translocations du gène ALK (Anaplastic
Lymphoma Kinase) fait espérer une amélioration du pronostic de ces
tumeurs. En effet, les cellules présentant un réarrangement d’ALK sont
dépendantes de l’activation de la kinase ALK, et une réponse favorable
après traitement par une petite molécule inhibitrice, le crizotinib, a été
rapportée dans des essais cliniques. Les patients ALK positifs sont le
plus souvent non fumeurs ou de petits fumeurs (moins de 10 paquets-
année) et porteurs d’un adénocarcinome d’architecture solide et
acinaire, souvent avec des massifs cribriformes riches en cellules
mucipares en bague à chaton exprimant le TTF1. Trois techniques
peuvent être utilisées pour la mise en évidence d’un réarrangement
d’ALK dans les cellules tumorales : l’immunohistochimie, la RT-PCR
(Real-Time Polymerase Chain Reaction) et la FISH (hybridation in situ en
fluorescence). La détection de ces réarrangements est proposée en
France par l’Institut national du cancer (INCa) au sein des plateformes
hospitalières de génétique moléculaire des cancers et s’inscrit dans
un algorithme de diagnostic décisionnel en cours de validation aux
niveaux national et international.
Mots-clés : Carcinome – Poumon – ALK – Immunohistochimie – FISH
– RT-PCR.
Since the discovery in 2004 of EGFR (Epidermal Growth Factor
Receptor) mutations, responsible for a specific sensitivity of
mutated tumors to tyrosine kinase inhibitors, the prognosis
of non-small-cell lung carcinoma (NSCLC) has improved
dramatically along with the development of targeted
therapies. In 2007, Soda et al. described a rearrangement of
the ALK (Anaplastic Lymphoma Kinase) gene in a subset of
lung adenocarcinomas, leading to a permanent activation of
the ALK kinase, specifically targeted by a small molecule raised
against ALK and MET, crizotinib. ALK gene rearrangements are
observed mainly in adenocarcinomas with solid and acinar
histology, rich in TTF1 positive signet-ring cells, which arise in
non or light smokers (< 10 pack year), with advanced stage
disease at diagnosis. Three diagnostic techniques can be
used: immunohistochemistry, RT-PCR (Real-Time Polymerase
Chain Reaction) and FISH (Fluorescence In Situ Hybridization),
the latter being the reference technique recommended by
the Institut national du cancer (INCa) program in France.
However, several national and international studies are
ongoing in order to determine a diagnostic algorithm for
a large-scale screening.
Keywords: Carcinoma – Lung – ALK – Immunohistochemistry
– FISH – RT-PCR.
L
e cancer du poumon reste la première cause de
décès par cancer dans le monde, et est responsable
de plus de 1,2 million de décès par an (1). Sur le plan
clinique, histologique et thérapeutique, les cancers du
poumon se divisent en 2 grands groupes : les carcinomes
à petites cellules, dont l’incidence est en diminution, et les
carcinomes bronchiques non à petites cellules (CBNPC),
constitués principalement des adénocarcinomes, qui
représentent environ 50 % des cancers du poumon, et
des carcinomes malpighiens (environ 30 %). Le pronostic
des cancers non à petites cellules reste redoutable, avec
une survie globale tous stades confondus n’excédant pas
5 % à 5 ans. La principale raison en est que, au moment du
diagnostic, 75 % de ces cancers sont métastatiques et donc
non opérables. Si la majorité des cancers du poumon sont
associés à une consommation tabagique, 15 % des cancers
chez les hommes et 53 % des cancers du poumon chez
les femmes surviennent chez des non-fumeurs ; il s’agit
surtout d’adénocarcinomes, dont 20 % ne sont pas liés au
tabac. Ces tumeurs présentent une carcinogenèse à part,
souvent marquée par une véritable addiction à une muta-
tion, une translocation ou une amplification activatrice

Correspondances en Onco-Théranostic - Vol. I - n° 3 - juillet-août-septembre 2012
108
dossier thématique
ALK
Réarrangements du gène ALK dans les cancers du poumon non à petites cellules
d’un oncogène (2, 3). Ainsi, en 2004 le rôle des mutations
activatrices (4) du récepteur à l’EGF (Epidermal Growth
Factor) a été démontré dans certains adénocarcinomes,
conférant à ces tumeurs une sensibilité aux inhibiteurs de
tyrosine kinase. L’intérêt est que ces oncogènes activent
des voies de signalisation stimulant la croissance cellulaire
et la résistance à l’apoptose des cellules tumorales uni-
quement, si bien que, lorsqu’une protéine oncogénique
est ciblée par une molécule inhibitrice, seules les cellules
malignes sont atteintes, et non les cellules normales (2).
En 2007, une autre anomalie génétique concernant le gène
codant pour la kinase ALK a été décrite par M. Soda et al.
dans les CBNPC ; cette anomalie procède d’une translo-
cation entre 2 gènes, le gène ALK (Anaplastic Lymphoma
Kinase) en 2p23 et le gène EML4 (Echinoderm Microtubule
associated protein Like 4) en 2p21 (5). Cette altération résulte
en fait d’une inversion au niveau du bras court du chromo-
some 2 et d’une fusion de la région codant pour la tyrosine
kinase (TK) ALK avec différentes portions N-terminales plus
ou moins tronquées du gène EML4 (figure 1). Ces réarran-
gements sont responsables d’une activité tyrosine kinase
permanente via la dimérisation du domaine TK, indé-
pendante de toute fixation à un ligand ; cette activation
constante est responsable de la prolifération des cellules
tumorales, de changements dans leur cytosquelette, de
leur migration et de leur survie (6). Comme pour les muta-
tions d’EGFR, les cellules qui présentent ce réarrangement
sont dépendantes de l’activation de la kinase ALK (7). De
multiples variants ont été décrits (environ 15 actuellement)
[8]. Ces variants impliquent l’exon 20 du gène ALK qui code
pour le domaine TK intracytoplasmique et la région d’EML4
qui code pour le domaine coiled coil aminoterminal d’EML4
nécessaire à l’activation d’ALK ; les variants EML4-ALK les
plus fréquents sont E13;A20 et E6a/b;A20, observés dans
33 et 29 % des cas (9). D’autres partenaires qu’EML4 ont
été rapportés, tels que TFG (TRK-fused gene), KIF5B (kine-
sin family member 5B) et, plus récemment, KLC1 (kinesin
light chain 1) [figure 2] (10-12). Ils seraient responsables
Figure 1. Schéma de la translocation EML4-ALK, illustrant l’inversion de la partie aminoterminale
d’EML4 et sa fusion avec la partie du gène ALK codant pour le domaine tyrosine kinase.
EML4
KINASE
EML4
EML4-ALK variant 1
EML4-ALK variant 1
ALK
ALK
1
1
1496
496
981
1 059
1 620
1 058
Domaine transmembranaire
Exon 21 Exon 13
Figure 2. Schéma des différents variants actuellement recensés du réarrangement du gène ALK avec ses différents partenaires.
EML4 ALK
ALK
ALK
ALK
ALK
ALK
ALK
ALK
ALK
EML4
EML4
EML4
EML4
EML4
EML4
EML4
Domaine tyrosine kinase
KLC1, TGF ou KiF5B
EML4-ALK
Variant 1 (33 %) - E13;A20
Variant 3a/b (29 %) - E6;A20
Variant 2 (9 %) - E20;A20
Variant 4’ et 7 (3 %) - E14;A20
Variant 5’ (2 %) - E18;A20
Variant 4 (2 %) - E15;A20
Variant 5 a/b (2 %) - E2;A20
E17;A20
KLC1, TFG ou KiF5B-ALK

Correspondances en Onco-Théranostic - Vol. I - n° 3 - juillet-août-septembre 2012
109
Réarrangements du gène ALK dans les cancers du poumon non à petites cellules
d’une activation de la kinase ALK similaire à celle issue
des variants EML4-ALK, avec une sensibilité potentielle
aux inhibiteurs d’ALK.
Données anatomocliniques
Les réarrangements du gène ALK sont observés dans
3 à 13 % des CBNPC selon les séries, avec une inci-
dence aux alentours des 5 % dans les études sur des
populations non sélectionnées. La plupart des patients
porteurs de ce réarrangement sont plus jeunes que
les autres patients atteints de cancer du poumon, et
sont de petits (< 10 paquets-année) fumeurs ou des
non-fumeurs (5, 7, 13). Du point de vue histologique,
les carcinomes ALK réarrangés sont majoritairement
des adénocarcinomes, d’architecture solide et acinaire,
souvent avec des massifs cribriformes riches en cellules
mucipares en bague à chaton TTF1 positives (figure 3A) ;
certains adénocarcinomes papillaires ALK réarrangés
ont également été décrits, ainsi que d’exceptionnels
carcinomes malpighiens ou des adénosquameux (10,
13, 14). Une réponse favorable après traitement par une
petite molécule inhibitrice d’ALK et de MET, le crizotinib
(PF-02341066), a été rapportée en essai de phase I ;
2 essais de phase II et III sont en cours, notamment en
première et deuxième lignes par comparaison avec
la chimiothérapie, et en troisième ligne et au-delà en
phase II. En France, le crizotinib est disponible en autori-
sation temporaire d’utilisation (ATU) après échec d’une
chimiothérapie (15, 16).
Tests diagnostiques
La mise en évidence d’un réarrangement d’ALK dans
les cellules tumorales peut être effectuée par diffé-
rentes méthodes : l’immunohistochimie met en évi-
dence une expression aberrante du domaine kinase
intracytoplasmique de la protéine, la RT-PCR permet
la détection des différents transcrits de fusion grâce à
l’utilisation d’amorces spécifiques, et les techniques
d’hybridation in situ permettent la mise en évidence
directe du réarrangement.
Immunohistochimie
Le clone ALK1 est classiquement utilisé pour le dia-
gnostic des lymphomes anaplasiques à grandes cel-
lules ; il présenterait une sensibilité de 90,0 % et une
spécificité de 97,8 % (17) pour le diagnostic des adéno-
carcinomes ALK réarrangés ; cependant, sa sensibilité
paraît inférieure à celle des 2 autres clones actuelle-
ment utilisés dans la littérature pour le diagnostic
de ces adénocarcinomes (10) : il s’agit du clone 5A4,
capable de détecter les protéines de fusion issues des
variants EML4-ALK v1, v2, v3, v6 et v7 et du variant
KiF5B-ALK, et du clone D5F3, présentant tous 2 une
sensibilité de 100 % et une spécificité de 99 % (9, 10,
18-21). Le marquage observé est souvent intense,
cytoplasmique et diffus (figure 3, C et D), mais, dans
de rares cas, il peut être faible et focal [18, 21].
RT-PCR
La mise en évidence par RT-PCR des différents transcrits
de fusion d’ALK nécessite l’utilisation d’une technique
“multiplex” utilisant une amorce commune pour la
mise en évidence de l’exon 20 d’ALK et des amorces
spécifiques variées pour la mise en évidence du par-
tenaire de fusion. Cette technique est très sensible,
mais la longueur des fragments amplifiés pouvant
être supérieure à 1 000 paires de bases, il est néces-
saire de disposer de matériel congelé ; c’est rarement
le cas (5). Des kits de détection par RT-PCR ont ainsi
été développés (ALK Fusion TaqMan® Gene expression
assays, Life Technologies), permettant la détection des
variants les plus fréquents (E6a/b;A20, E13;A20, E20;A20,
E17;A20 et E18;A20) à partir d’ARN extraits de tissus
fixés et inclus en paraffine. Environ soixante-dix pour
cent des variants EML4-ALK peuvent ainsi être mis en
Figure 3. A. Histologie classique solide et acinaire riche en cellules en bague à chaton des
adénocarcinomes ALK réarrangés. B. FISH ALK (sonde break-apart) montrant une séparation (ou
split) des sondes marquées en rouge et en vert (situées de part et d’autre du point de cassure
localisé dans l’exon 20 du domaine kinase d’ALK) et témoignant d’un réarrangement du gène
ALK. C et D. Immunohistochimie anti-ALK (clone 5A4).

Correspondances en Onco-Théranostic - Vol. I - n° 3 - juillet-août-septembre 2012
110
ALK
dossier thématique
Accédez à la pathologie moléculaire
avec Roche, votre partenaire pour l’avenir.
Porté par les valeurs d’innovation et de progrès, Roche Diagnostics s’investit chaque jour dans la recherche
et le développement pour proposer aux pathologistes le meilleur du diagnostic : une offre d’instruments
complète et une large gamme de marqueurs VENTANA®, des technologies telles que l’IHC et l’HIS.
Aujourd’hui Roche va plus loin et vous ouvre les portes du diagnostic moléculaire avec la PCR et le séquençage :
grâce à ce savoir-faire de pointe et à un accompagnement au quotidien, Roche acquiert une longueur d’avance
dans l’histopathologie et se place comme le partenaire d’aujourd’hui et de demain.
L’anticorps VENTANA® ALK (D5F3), bientôt disponible.
Dernière concrétisation de l’expertise Roche Diagnostics, l’anticorps VENTANA® ALK (D5F3),
utilisé dans le diagnostic broncho-pulmonaire, arrive prochainement sur le marché.
Pour être informé de sa mise à disposition, flashez le code ci-contre ou rendez-vous sur www.roche-alk.com.
Septembre 2012
AP RTD ALK EVDEF2.indd 1 04/09/12 12:18
évidence par cette technique, mais une confirmation
par une autre technique (IHC ou FISH) est nécessaire,
en particulier dans le cas de l’absence de variant, afin
d’éliminer tout risque de faux négatif.
FISH
La FISH est la technique de référence pour la mise en
évidence des réarrangements d’ALK dans les CBNPC,
la présence en FISH d’un réarrangement d’ALK dans
plus de 15 % des cellules tumorales étant le test dia-
gnostique utilisé pour l’inclusion des patients dans un
protocole de traitement par crizotinib à ce jour (22).
L’utilisation de sondes break-apart (ou split) permet la
mise en évidence d’un réarrangement d’ALK quel que
soit son partenaire (figure 3, B, p. 109), mais l’interpré-
tation du résultat est parfois difficile, surtout pour les
réarrangements intrachromosomiques conduisant à
une séparation des signaux FISH (indiquant un réar-
rangement) parfois ténue. Les sondes dites de fusion,
spécifiques de 2 partenaires connus, permettent d’iden-
tifier le partenaire de fusion d’ALK.
Conclusion : algorithme diagnostique
Les mutations d’EGFR et de KRAS et les réarrangements
du gène ALK étant considérées comme mutuellement
exclusives, l’INCa a privilégié dans ses programmes
de 2011 et de 2012 le testing ALK des adénocarci-
nomes métastatiques sans mutation d’EGFR ni de
KRAS ; cependant, cet algorithme diagnostique est
actuellement discuté, car plusieurs cas d’adénocarci-
nomes à la fois mutés pour EGFR et réarrangés pour
ALK ont été rapportés. La technique de référence
choisie dans ces programmes de l’INCa était la FISH,
mais, pour des raisons financières et de facilité d’uti-
lisation, la possibilité d’un screening à grande échelle
par immunohistochimie, dès le diagnostic histolo-
gique, est envisagée, avec la nécessité de confirmer les
cas positifs ou douteux par la FISH ou la RT-PCR. Une
étude nationale et plusieurs études internationales
de validation d’un tel algorithme diagnostique sont
en cours et devraient assez rapidement permettre de
statuer sur sa faisabilité. ■
1. Travis WD, Brambilla E, Noguchi M et al. International
Association for the Study of Lung Cancer/American Thoracic
Society/European Respiratory Society international multi-
disciplinary classification of lung adenocarcinoma. J Thorac
Oncol 2011;6(2):244-85.
2. Pirker R, Pereira JR, von Pawel J et al. EGFR expression as a
predictor of survival for first-line chemotherapy plus cetuxi-
mab in patients with advanced non-small-cell lung cancer:
analysis of data from the phase 3 FLEX study. Lancet Oncol
2011;13(1):33-42.
3. Shigematsu H, Gazdar AF. Somatic mutations of epidermal
growth factor receptor signaling pathway in lung cancers. Int
J Cancer 2006;118(2):257-62.
4. Mok TS, Wu YL, Thongprasert S et al. Gefitinib or carbopla-
tin-paclitaxel in pulmonary adenocarcinoma. N Engl J Med
2009;361(10):947-57.
5. Soda M, Choi YL, Enomoto M et al. Identification of the
transforming EML4-ALK fusion gene in non-small-cell lung
cancer. Nature 2007;448(7153):561-6.
6. Horn L, Pao W. EML4-ALK: honing in on a new target
in non-small-cell lung cancer. J Clin Oncol 2009;27(26):
4232-5.
7.
Koivunen JP, Mermel C, Zejnullahu K et al. EML4-ALK fusion
gene and efficacy of an ALK kinase inhibitor in lung cancer.
Clin Cancer Res 2008;14(13):4275-83.
8.
Sanders HR, Li HR, Bruey JM et al. Exon scanning by reverse
transcriptase-polymerase chain reaction for detection of
known and novel EML4-ALK fusion variants in non-small cell
lung cancer. Cancer Genet;204(1):45-52.
9. Sasaki T, Rodig SJ, Chirieac LR et al. The biology and treat-
ment of EML4-ALK non-small cell lung cancer. Eur J Cancer
2010;46(10):1773-80.
10.
Takeuchi K, Choi YL, Togashi Y et al. KIF5B-ALK, a novel
fusion oncokinase identified by an immunohistochemistry-
based diagnostic system for ALK-positive lung cancer. Clin
Cancer Res 2009;15(9):3143-9.
11. Rikova K, Guo A, Zeng Q et al. Global survey of phospho-
tyrosine signaling identifies oncogenic kinases in lung cancer.
Cell 2007;131(6):1190-203.
12. Togashi Y, Soda M, Sakata S et al. KLC1-ALK: a novel fusion
in lung cancer identified using a formalin-fixed paraffin-
embedded tissue only. PLoS One 2012;7(2):e31323.
13. Rodig SJ, Mino-Kenudson M, Dacic S et al. Unique cli-
nicopathologic features characterize ALK-rearranged lung
adenocarcinoma in the western population. Clin Cancer Res
2009;15(16):5216-23.
14. Inamura K, Takeuchi K, Togashi Y et al. EML4-ALK lung
cancers are characterized by rare other mutations, a TTF-1
cell lineage, an acinar histology, and young onset. Mod Pathol
2009;22(4):508-15.
15.
Kwak EL, Bang YJ, Camidge DR et al. Anaplastic lymphoma
kinase inhibition in non-small-cell lung cancer. N Engl J Med
2010;363(18):1693-703.
16. Ou SH, Bazhenova L, Camidge DR et al. Rapid and dramatic
radiographic and clinical response to an ALK inhibitor (crizoti-
nib, PF02341066) in an ALK translocation-positive patient with
non-small cell lung cancer. J Thorac Oncol 2010;5(12):2044-6.
17.
Yi ES, Boland JM, Maleszewski JJ et al. Correlation of IHC
and FISH for ALK gene rearrangement in non-small cell lung
carcinoma: IHC score algorithm for FISH. J Thorac Oncol
2011;6(3):459-65.
18.
Mino-Kenudson M, Chirieac LR, Law K et al. A novel, highly
sensitive antibody allows for the routine detection of ALK-
rearranged lung adenocarcinomas by standard immunohis-
tochemistry. Clin Cancer Res 2010;16(5):1561-71.
19. Paik JH, Choe G, Kim H et al. Screening of anaplastic lym-
phoma kinase rearrangement by immunohistochemistry in
non-small cell lung cancer: correlation with fluorescence in situ
hybridization. J Thorac Oncol 2011;6(3):466-72.
20. Jokoji R, Yamasaki T, Minami S et al. Combination of
morphological feature analysis and immunohistochemistry
is useful for screening of EML4-ALK-positive lung adenocar-
cinoma. J Clin Pathol 2010;63(12):1066-70.
21.
McLeer-Florin A, Moro-Sibilot D, Melis A et al. Dual IHC
and FISH testing for ALK gene rearrangement in lung ade-
nocarcinomas in a routine practice: a French study. J Thorac
Oncol 2012;7(2):348-54.
22. Camidge DR, Theodoro M, Maxson DA et al. Correlations
between the percentage of tumor cells showing an ALK (ana-
plastic lymphoma kinase) gene rearrangement, ALK signal copy
number, and response to crizotinib therapy in ALK fluorescence
in situ hybridization-positive nonsmall cell lung cancer. Cancer
2012;118(18):4486-94.
Références
1
/
4
100%