Mutation de KRAS

8ème journées de Cancérologie Digestive – PACA/Corse
Marseille 23 novembre 2012
Astrid Lièvre
Département d Oncologie Médicale
Institut Curie Hôpital René Huguenin (Saint-Cloud)
Université Versailles Saint-Quentin-en-Yvelines
Facteurs prédictifs d efficacité
des biothérapies
Nouveautés!

Mutation de KRAS: marqueur de résistance aux anticorps anti-EGFR
!"#$%&'&(')*+',)-.&%'/&0'12234'!"#$%&'&(')*+'5',*"-'6-.7*'12284'9)%):&;0'<'=->'5'?&@'
12284'AB)@7'5',*"-'6-.7*'12284'C)-',D(0&B'5',*"-'6-.7*'12EE4'F7G&B&H&%'A--'
6-.7*'12EE''

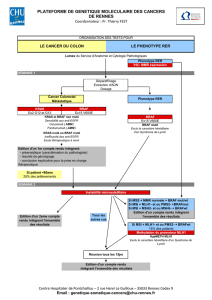
Statut mutationnel du gène KRAS
Paramètre essentiel dans la stratégie thérapeutique du CCRm
ESMO ASCO

COSMIC (Catalogue of Somatic Mutations in Cancer) !
http://www.sanger.ac.uk/genetics/CGP/cosmic/!
18% (G13D)
81%
23% (G12V)
35% (G12D)
Mutations de KRAS et cancer colorectal
Codons 12 et 13: hotspot de mutations (> 95% des cas)
Exon 2

CCRm mutés G13D: potentiellement sensibles au cetuximab?
579 pts chimiorésistants TTT par cetuximab
cetuximab SSP et SG chez les G13D vs autres mutations de KRAS (codon 12)
De Roock et al. JAMA 2010; 304:1812-1820
• Evaluation dans études randomisées OPUS and CRYSTAL
• 83 pts avec mut G13D KRAS (15% des tumeurs mutés KRAS, 6% des tumeurs)
cetuximab+chimio vs chimio
S. Tejpar et al., J Clin Oncol 2012; 30:3570-3577
Mutation G13D de KRAS: la polémique
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
1
/
26
100%