Maturation ARN pré-messagers et épissage - Cours

Minault Quentin
Cazals Cyril
Binome 5
Cours du 16/09/11 14h-16h
_____________________________________________________________________________________________
Maturation des ARN pré messagers – Epissage
« Mes exemples sont là pour illustrer le cours » la prof.
I) Maturation des ARN pré – messagers : 2 « retouches »
aux extrémités et un bouleversement à l’intérieur
1) De l’ADN à l’ARN
L’ADN contient environ 20 000 – 25 000 gènes.
La RNA polymérase permet le passage du gène à l’ARN, c’est la transcription.
Cet ARN est alors sous forme de pré messager qui va par la suite subir
plusieurs étapes avant de pouvoir devenir mature : formation d’une structure
cap (coiffe en 5’) ainsi qu’une coupure de l’ARN naissant en 3’ (clivage/ poly
adénylation).
De plus, après ou pendant la transcription, on assiste à une étape nommée
l’épissage : L’ARN une fois mature pourra être exporté en dehors du noyau
vers le cytoplasme afin d’y être traduit et dégradé.
Ces mécanismes sont importants car de nombreuses maladies humaines et
cancers sont dues à des altérations au niveau des différentes étapes citées ci –
dessus (Alzheimer, syndrôme du X fragile, cancers…).
2) Maturation de l’ARN pré messager
Chez les eucaryotes, l’ARNm possède une coiffe en 5’ ( le cap), des séquences
codantes (exons), des séquences non codantes (introns), une queue polyA en
3’, une séquence traduite ORF et des séquences non traduites (5’ et 3’ UTR : ce
sont les régions régulatrices).
a) Le 5’ capping
En 5’ de l’ARN transcrit, il reste une extrémité triphosphate. La synthèse du cap
se fait en 4 étapes :
- l’ARN 5’ triphosphatase enlève 1 phosphate
- la guanylyl transférase permet l’ajout d’un GTP et le rejet de PPi
- méthylation de cap sur la première guanine par la 7-guanine methyl transférase
- ajout de groupements méthyls sur les riboses grâce à la 2-O’ methyl transférase

La coiffe (cap) est rapidement ajoutée après l’initiation car toutes les enzymes
nécessaires se fixent directement sur la RNA polymérase. Ce mécanisme est
indépendant de la séquence AND il ne concerne que l’ARNprémessager
La coiffe sert à stabiliser l’ADN contre les nucléases et c’est également un site de
liaison pour différents facteurs.
La liaison entre la coiffe et le RNA est une liaison triphosphate 5’ => 5’ alors que
la liaison entre les nucléosides de l’ARN est une liaison phosphodiester 3’ => 5’.
b) La polyadénylation
Une séquence est reconnue par le complexe protéique. Celui ci clive
l’ARNm dégradation de la partie 3’ et ajout d’une queue polyA par une
polyA polymérase.
Rôles :
- Augmenter la stabilité du messager via eIF4G : l’ARNm prend une
conformation circulaire dans le cytoplasme, cela bloque l’activité des
nucléases.
- Favoriser la traduction.
II) L’épissage constitutif : Principes et Aspects
réactionnels
1) Principe
Epissage = splicing = enlever les introns + relier les exons
Les séquences consensus : 2 types (site fort si aucune variation de séquence et
site faible si variation de séquence).
Introns pas forcément épissé dans ordre.
Les séquences cis :
Au niveau de l’exon n : site donneur en 5’ (GU)
Au niveau de l’exon n+1 : site accepteur en 3’ (AG)
Chez l’homme il existe entre site branchement et site accepteur un pont
polypyrimidique
Sequences tres conservees chez levure d’ou travail sur levure.
Reconnues par le spliceosome ( conservé de la levure a homme).
Il existe d’autres séquences cis : inhibitrices (silencers) et activatrices
(enhancers).

2) Deux réactions de transestérification (voir diapo)
- Le groupement 2’OH du site accepteur(qui est fortement réactif) lance
une attaque nucléophilique sur la guanine du site donneur du premier
exon. L’adénine et la guanine sont alors reliées et forment une stucture
en lariat (lassot).
- Le groupement 3’ OH du site donneur lance une attaque nucléophilique
au niveau du site accepteur, il a relarguage de l’intron et ligation des
deux exons.
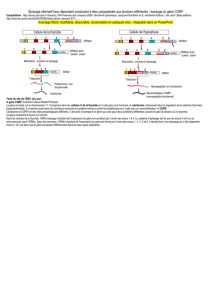
3) Composant du spliceosome (complexe d’épissage)
Le spliceosome correspond au complexe d’épissage.
C’est un complexe multi mega Dalton (60S) .
Il est composé de 5 ARN sn (small nuclerar) : U1, U2, U4, U5 et U6 qui sont
associés à des protéines sn (7) et à des protéines non snRNP (plus de 170 chez
l’homme).
5 U sont donc impliqués et auront des fonctions différentes :
- U1 reconnaît le site donneur
- U2 reconnaît le point de branchement
- U4 et U6 interagissent ensemble avec le site donneur et donc aussi avec U1
- U5 avec les sites en 5’ et 3’
Les protéines du spliceosome (snRNP) ont aussi différentes fonctions (liaison à
l’ARN, activité, interaction). Le spliceosome s’assemble de manière plus ou moins
séquentielle, c’est une structure dynamique présentant de nombreux
réarrangements, ce qui permet la stabilité et flexibilité de l’épissage.
a) Interactions ARN/ARNm
U1 s’hybride avec site donneur,formation d’une séquence double brin par
appariement.
U2 s’hybride avec le site de branchement mais le A ne s’apparie pas.
U4 et U6 s’apparient ensemble.
b) Réarrangement ARN/ARN
U2 au niveau du site de branchement.
U6 interagit avec le site 5’.
Conséquences juxtaposition du site donneur du premier exon et du
point de branchement, ce réarrangement ARN/ARN va rapprocher dans
l’espace les sites 5’ et 3’ (grâce à U5) et donc rapprocher les deux exons,
ce qui permet la deuxième réaction de transestérification.
c) Réarrangement protéine/protéine
Au cours de l’épissage, des protéines s’ajoutent, d’autres s’enlèvent.

4) Assemblage du spliceosome
- Complexe E : quand U1 est arrivé sur le site 5’ (site donneur) et est stabilisé,
l’ARN s’engage dans le cycle d’épissage
- Complexe A (pré spliceosome) : U2 vient se fixer sur le site de branchement
(le complexe commence à être stable)
- Complexe B (spliceosome pré catalytique) : U4 – U5 –U6 arrivent sous forme
de trimère. U6 par son activité hélicase commence à chasser U1 et U4. Il se
forme alors le complexe B actif où il n’y a pas encore de corps catalytique
- Complexe B (spliceosome actif catalytiquement) : Par de nombreux
réarrangements.
- Complexe C (spliceosome activé) : U4 est enlevé pour rendre U6 accessible.
U6 reconnaît l’ARN prémessager, fait des réactions avec U2 pour faire la
première étape de transestérification structure en lariat.
Le complexe C subit la 2ème réaction de transestérification ligation des
deux exons + relarguage de l’intron recyclage de protéines et des ARN du
spliceosome qui s’étaient dissociées.
III) Bases de la reconaissance des sites d’épissage in
vivo, par la machinerie d’épissage
Il existe plusieurs types de gènes :
- SUR2(cytochrome 450) : gène typique chez mammifères (40kb)
- Dystrophine humaine : plus grand gène, responsable de la dystrophie de
Duchenne et Drecker (2,4 Mb)
- Nébulline : 183 exons , 249 kb: le plus complexe
La caractéristique invariante est que les exons interne ont une taille régulière
(entre 50 - 150 pb) et les introns une taille variable ( de 100 à 300 pb).
Comment la cellule va t – elle reconnaître des séquences variables sur des
longueurs aussi grandes ?
Ce qui permet de reconnaître les exons des introns sont les séquences consensus
en 5’ et 3’.
Chez S. cerevisiae, les séquences sont bien conservées ainsi que le site de
branchement.
Chez H. sapiens, à part le GU (qui est une séquence invariable), tout le reste peut
être non conservé. Le site de branchement est également très peu conservé.

Mécanismes facilitant la reconnaissance des sites d’épissage :
1) Couplage de la transcription et de l’épissage
- Vitesse de polymérisation :
- 1500 à 2000 nts par min
- 2 à 3 min pour synthétiser un pré-mRNA de 5 kb
- 50 - 75 min pour un pré-mRNA de 100 kb
- Cinétique de l’épissage in vivo :
- 2 min pour 1 intron de 0.5 à 1 kb
»»» Un gène de moins de 5 kb peut être transcrit totalement, puis épissé.
»»» Un gène de plus de 5-6 kb est transcrit et épissé de manière
concomitante.
Seuls les gènes transcrits par la Pol II ont une structure en
exons/introns (gènes « morcelés ») et sont soumis à l’épissage.
Des gènes morcelés (normalement transcrits par Pol II), mais
fusionnés à un promoteur Pol III ou T7 sont transcrits in vivo mais sont
peu ou pas épissés.
Particularité de la Pol II par rapport aux autres Pol : présence d’un C-
terminal domain (CTD) : formé de 50 répétitions d’un heptapeptide (des
sérines en 2’ et en 5’). C’est une plateforme d’interactions avec d’autres
protéines.
Il faut savoir que la phosphorylation de la sérine 5’ initie le
transcription tandis que celle de la sérine 2’ initie l’élongation.
In vivo, une Pol II tronquée de son domaine CTD peut transcrire les
gènes, mais les pré-mRNA résultant sont maturés et épissés
inefficacement.
La Pol II active un épissage in vitro.
Ces données mettent en évidence le rôle du CTD dans l’épissage. Il
intervient comme plate-forme d’interactions avec de multiples facteurs de
maturation et d’épissage.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
1
/
11
100%