TD 1 épissage alternatif

TD1 : Epissage alternatif
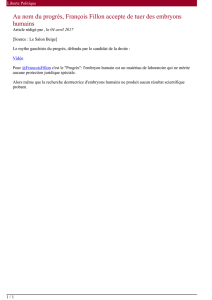
Le gène de la tropomyosine α de xénope possède trois exons 3' terminaux, appelés exon 9A9', 9B et
9D, dont la sélection différentielle en fonction du contexte tissulaire produit trois ARNm distincts
(Figure 1A). Dans les somites, l'exon 9A9' est utilisé en tant qu'exon terminal avec la sélection d'un
signal de polyadénylation appelé αe ce qui conduit à la production d'un ARNm appelé XTMα7. Dans
le coeur embryonnaire et les muscles striés adultes un site d'épissage 5' interne à l'exon 9A9' est
sélectionné. Il y a alors épissage de l'exon 9A à l'exon 3' terminal 9B ce qui conduit à la production
d'un ARNm appelé XTMα2. Finalement dans les tissus non musculaires il y a sélection de l'exon 3'
terminal 9D qui est épissé directement à l'exon 8 pour produite l'ARNm XTMO5.
Les auteurs décident d'étudier in vivo les mécanismes qui contrôlent l'utilisation de l'exon 9A9'.
1. A votre avis pourquoi les auteurs ont-ils choisi d'étudier la régulation de cet exon ?
La région génomique qui couvre les exons 7 à 9B a été clonée par PCR, séquencée et placée sous le
contrôle de différents promoteurs :
- le promoteur de l'actine cardiaque qui est activé spécifiquement dans les somites.
- le promoteur de la kératine embryonnaire qui est activé spécifiquement dans l'épiderme.
- le promoteur précoce du virus SV40 qui est actif dans l'ovocyte de xénope.
2. Comment appellent-on de telles constructions ?
Les constructions sous le contrôle des promoteurs actine et kératine ont été injectées dans l'embryon
de xénope au stade deux cellules. Après quelques heures de développement les embryons ont été
fixés et les ARN ont été extraits. La construction sous le contrôle du promoteur SV40 a été injectée
dans l'ovocyte de xénope. Après quelques heures d'incubation les ARN ont été extraits. Les ARNm
issus des minigènes ont été analysés par RT/PCR selon le principe décrit dans la figure 1B. Les
résultats d'une telle analyse sont donnés dans la figure 2.
3. Après avoir expliqué le principe de l'analyse par RT/PCR interpréter les résultats obtenus.
Les sites d'épissage 3' et 5' de l'exon 9A9' ont été détruits ou mutés en séquences consensuelles. Le
site de polyadénylation αe a été détruit ou remplacé par un site de polyadénylation synthétique appelé
SPA qui comporte des séquences optimales pour la réaction de polyadénylation/cleavage. Les
différents minigènes mutants ont été injectés dans les embryons et les ARNm produits ont été
analysés par RT/PCR. Les résultats sont donnés dans la figure 3.
4. Quelles sont les caractéristiques des sites d'épissages 5' et 3' de l'exon 9A9'? Quels sont
selon vous les paramètres importants pour obtenir un signal de polyadenylation optimal ?
Interprétez les résultats obtenus.
L'étude informatique de la région 3' de l'intron en amont de l'exon 9A9' a permis de mettre en
évidence cinq sites de branchement potentiels dans un région comprise entre -130 nt et -275 nt en
amont du site 3' d'épissage. Afin de cartographier le point de branchement utilisé in vivo les auteurs
ont mutés l'adénosine de ces cinq sites potentiels en thymidine ou cytosine. Les mutants placés sous
le contrôle du promoteur actine cardiaque ont été injectés dans des embryons au stade 2 cellules et
analysés par RT/PCR. Les résultats sont donnés dans la figure 4
5. Donner la séquence consensuelle d'un site de branchement chez les eucaryotes supérieurs.
Interpréter les résultats obtenus.
Pour déterminer si des séquences comprises entre le point de branchement et le site 3' d'épissage
sont importantes dans la régulation de l'exon 9A9', les auteurs ont construit trois mutants de délétion
appelés ∆1, ∆2 et ∆3 dans lesquels 80 nt, 150 nt et 230 nt respectivement ont été enlevés en amont
du site 3' d'épissage (Figure 5A). Pour éviter tout effet direct sur le site 3' d'épissage les délétions
maintiennent les 14 derniers nt de l'intron juste en amont de l'exon 9A9'. Ces différentes constructions

placées sous le contrôle des promoteur kératine et actine cardiaque ont été microinjectées dans
l'embryon de xénope et les ARN ont été analysés par RT/PCR. Les résultats sont donnés dans la
figure 5B et 5C.
6. Interprétez les résultats.
La région de 150 nt délétée dans le mutant ∆2 a été appelée 150PY. Elle a été remplacée par une
séquence de taille identique amplifiée à partir du vecteur pBS (appelée 150 pBS) pour produire le
mutant ∆2+pBS. La région 150PY possède quatre motifs de liaison (UCUU) à une protéine de liaison
aux ARN appelée Polypyrimidine tract binding protein ou PTB (Figure 4 A). Ces quatre motifs ont été
mutés en CCCC pour donner le mutant PTB1-4. Les mutants ∆2+pBS et PTB1-4 placés sous le
contrôle du promoteur kératine ont été microinjectés dans l'embryon de xénope et les ARN ont été
analysés par RT/PCR. Les résultats sont donnés dans la figure 5B et 5D.
7. A quelle fin a été réalisé le mutant ∆2+pBS. Que pouvez vous conclure de ces expériences.
Les ARN150PY sauvages et 150PY mutés au niveau des quatre sites de liaison à la PTB ont été
trancrits in vitro en présence de 32P αUTP. Les ARN radiomarqués ont été incubés avec de la PTB
recombinante. Les échantillons ont été exposés aux UV, traités à la RNase T1, analysés par
electrophorèse sur un gel d'acrylamide en présence de SDS et visualisés par autoradiographie. Les
résultats sont donnés dans la figure 6.
8. Exposer le principe d'une telle expérience. Que pouvez vous en conclure ?
Le minigène sauvage a été injecté dans l'ovocyte de xénope avec une quantité croissante
d'ARN150PY ou 150pBS. Après quelques heures d'incubation les ARN ont été extraits et analysés par
RT/PCR comme précédemment décrits. Les résultats sont donnés dans la figure 7.
9. Pourquoi les auteurs injectent-ils les ARN 150PY et 150pBS ?
Que pouvez vous en conclure ?
Il est possible de bloquer la traduction d'un ARNm dans l'embryon de xénope par l'injection
d'oligonucléotides modifiés antisens qui s'hybrident dans la région jouxtant le codon initiateur. De tels
oligonucléotides sont appelés des "morpholinos". Les auteurs ont injectés des morpholinos antisens
contrôles (C-MO) ou dirigés spécifiquement contre l'ARNm XPTB (XPTB-MO) dans des embryons au
stade deux cellules. La quantité de protéine XPTB présente dans des embryons au stade bourgeon
caudal a été analysée en western blot avec des anticorps anti XPTB (Figure 8 A).
Le minigène kératine a été co-injecté dans des embryons avec des morpholino C-MO ou XPTB-MO.
Le minigène kératine a également été co-injecté dans des embryons avec des morpholino XPTB-MO
et de l'ARNm XPTB dont la séquence jouxtant le codon initiateur a été modifiée. Après quelques
heures d'incubation les embryons ont été fixés. Les ARN ont été extraits et analysés par RT/PCR
comme précédemment décrit. Les résultats sont donnés dans la figure 8B.
10. Expliquer l'intérêt des différentes expériences réalisées. Interpréter ces résultats.

Figure 1
Figure 3
Figure 2
Figure 4

Figure 7
Figure 8
Figure 5
Figure 6

Figure 7
Figure 8
Figure 5
Figure 6
1
/
5
100%