BILAN D’ACTIVITE DES PLATEFORMES HOSPITALIERES DE GENETIQUE MOLECULAIRE Coordonnateur(s)

Plateforme hos
p
italière de
g
énéti
q
ue moléculair
e
Coordonnateur
(
s
)
CHU de Bordeaux et Institut Bergonié Pr JP MERLIO
BILAN D’ACTIVITE DES PLATEFORMES HOSPITALIERES DE GENETIQUE MOLECULAIRE
DES CANCERS POUR L’ANNEE 2011
Merci de faire parvenir ces données avant le 31 mars 2011 aux coordonnées suivantes :
Etienne Lonchamp

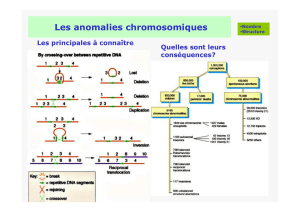
I- Données d’activité 2011 pour l'onco-hématologie : le document cible l’activité ayant un impact sur la prise en charge médicale.
Nombre de
patients avec
mutation ou
autre anomalie
Nombre de
patients sans
mutation ou
autre anomalie
Nombre de
patients avec un
résultat non
interprétable
Motif le plus
fréquent ayant
conduit aux
résultats non
interprétables
CH Ets privés
caryotype conventionnel 26 26 17 6 3
détection BCR-ABL RTPCR qualitative 227 227 155 72 0 52 39 14
suivi maladie
résiduelle quantification BCR-ABL RTPCR quantitative 2503 765 1251 184 75
suivi caryotype conventionnel 121 89 96 17 8
diagnostic et suivi anomalies de structure hors BCR
-
ABL FISH 2 2 20 0
LMC/LAL Suivi mutations ABL RT-PCR + séquençage 49 44 9 38 2 17 4 0
caryotype conventionnel 257 202 218 30 9
détection BCR-ABL RTPCR qualitative 40 40 4 36 0 0 38 2 0
détection divers transcrits de
fusion 114 54 21 33 0 0 114 0 0
autres anomalies de nombre et
structur
e
FISH 432 163 83 80 0 0 361 59 12
mutations FLT3 PCR et analyse de
fragment 66 66 14 52 0 66 0 0
mutations NPM PCR , analyse de
fragment et
séquençage 57 57 14 43 0 57 0 0
mutation CEPBA séquençage 37 37 4 33 0 37 0 0
mutations autres gènes
quantification BCR-ABL RTPCR quantitative 105 28 97 6
quantification WT1
quantification divers transcrits
de fusion
quantification d'un allèle muté
ou
g
ène h
yp
erex
p
rim
é
anomalies de nombre et
structur
e
FISH
LAL suivi maladie
résiduelle quantification IgH - TCR
caryotype conventionnel 92 92 50 26 16
anomalies de nombre et
structur
e
FISH 414 130 240 112 57
mutations somatiques IgVH
mutation p53
chimérisme post greffe - mise au
p
oin
t
chimérisme post greffe - suivi
Nombre de
patients
Nombre
d’examens
effectués en
2011
Suivi de la maladie
résiduelle
Test : Gène/marqueur
Diagnostic
Nombre de prescriptions établiss
e
plateforme
Technique utilisée1
Diagnostic
Résultats des tests (il n'est pas nécessaire de compléter les cases
grisées)
Niveau régional
Nombre de
prescriptions
établissements
de la plateforme
Pathologie
LMC
diagnostic
LAL/LAM
Leucémies suivi maladie
résiduelle
LLC
1 Si deux techniques distinctes sont utilisées au sein de la plateforme, merci de préciser le nombre de tests effectués par type de technique (créer autant de lignes que nécessaire).

Nombre de
patients avec
mutation ou
autre anomalie
Nombre de
patients sans
mutation ou
autre anomalie
Nombre de
patients avec un
résultat non
interprétable
Motif le plus
fréquent ayant
conduit aux
résultats non
interprétables
CH Ets privés
caryotype 32 31 25 3 4
mutations somatiques IgVH
anomalies de nombre et
structure FISH 670 398 150 88 313
anomalies de nombre et
structure FISH 81 35 49 27 5
détection remaniements
spécifiques (BCL1-JH, BCL2-JH,
autres…) FISH 26 26 10 0 14
détection cycline D1
quantification BCL1 ou2-JH pour
suivi
mutation JAK2 V617F PCR quantitative allèle
spécifique 890 842 280 562 0 0 304 208 56
quantification JAK2V617F
autres mutations HRM+sequençage 29 14 10 12 3
caryotype conventionnel 49 49 30 12 7
anomalies de nombre et
structure FISH 28 21 15 12 1
caryotype conventionnel 386 386 243 94 49
anomalies de nombre et
structure FISH 108 61 64 34 8
mutations diverses
caryotype conventionnel 8 8 33 2
anomalies de nombre et
structure FISH
CGH array
Nombre de prescriptions établiss
e
plateforme
Niveau régional
Pathologie
Nombre de
prescriptions
établissements
de la plateforme
lymphome
SMP non LMC
syndromes myélodysplasiques
myélomes et autres syndromes
lymphoprolifératifs
Résultats des tests (il n'est pas nécessaire de compléter les cases
grisées)
Test : Gène/marqueur
Nombre
d’examens
effectués en
2011
Nombre de
patients
Technique utilisée1

II- Données d’activité 2011 pour les tumeurs solides : le document cible l’activité ayant un impact sur la prise en charge médicale.
Niveau
interré
g
ional
Nombre de
patients avec
mutation ou
autre anomalie
Nombre de
patients sans
mutation ou
autre anomalie
Nombre de
patients avec un
résultat non
interprétable
Motif le plus
fréquent ayant
conduit aux
résultats non
interprétables
CH Ets privés
Activité pour des
établissements
dépendant d'une
autre plateforme
cancer de l'estomac amplification HER2 par ISH FISH 48 45 9 33 3 21 9 14 4
cancer colorectal mutations KRAS
HRM puis
séquençage si
anormal ou
séque
n
çage
seu
l
1108 1080 450 605 25 ADN non amplifiable 166 239 661 42
mutations KIT DHPLC puis
séquençage 223 220 167 43 10
ADN
non amp
lifi
a
bl
e
ou % de cellules
tumorales inférieur
au seuil de
sensibilité
46 18 41 118
mutations PDGFRA DHPLC puis
séquençage 223 220 167 43 10
ADN
non
amplifiable
ou % de cellules
tumorales inférieur
46 18 41 118
test MSI Pentaplex 325 310 55 247 8
A
DN non ampli
f
iable
ou % de cellules
tumorales inférieur
87 47 174 17
mutations BRAF PCR-Tacqman 52 49 38 10 1
A
DN non amplifiable
ou % de cellules
17 6 27 2
méthylation MLH1
1 Si deux techni
q
ues distinctes
2
Préciser les localisations tumorales et le biomarqueur (créer autant de lignes que nécessaire)
.
3
Préciser les localisations tumorales.
Niveau
interré
g
ional
Nombre de
patients avec
mutation ou
autre anomalie
Nombre de
patients sans
mutation ou
autre anomalie
Nombre de
patients avec un
résultat non
interprétable
Motif le plus
fréquent ayant
conduit aux
résultats non
interprétables
CH Ets privés
Activité pour des
établissements
dépendant d'une
autre plateforme
cancer du poumon mutations EGFR Séquençage direct 1514 1501 174 1235 92 ADN non amplifiable 460 347 697 10
cancer du sein amplification HER2 par ISH FISH 440 421 86 318 17
Sans hybridation ou
% de cellules
tumorales
inférieures au seuil
167 71 153 49
cancer du col utérin détection HPV de haut risque ( hors
indications ASCUS
)
PCR 176 173 33 3 12 128
FISH 464 455 64 17 40 343
PCR 45 45 14 14 17 0 1 1 43
Nombre de
prescriptions
établissements
de la plateforme
Nombre de prescriptions établissements hors
plateforme
Niveau régional
Résultats des tests (il n'est pas nécessaire de compléter les cases
grisées)
Résultats des tests
Nombre de
prescriptions
établissements
de la plateforme
Nombre de prescriptions établissements hors
plateforme
Nombre
d’examens
effectués en
2011
Nombre de
patients
tumeurs du spectre HNPCC
Pathologie Test : Gène/marqueur
Pathologie Test : Gène/marqueur Technique
utilisée1
Niveau régional
GIST
sarcomes
Technique
utilisée1
Nombre
d’examens
effectués en
2011
Nombre de
patients
translocations diverses
lifi ti MDM2/CDK4

FISH 460 458 199 193 66
S
ans
h
y
b
r
id
at
i
on ou
% de cellules
tumorales
77 17 61 305
mutation BRAF HRM puis
séquençage si
a
n
o
rm
a
l317 317 118 189 10 243 22 46 6
mutation BRAF à visée diagnostique PCR-Tacqman
V
600E 7725 1 6
déséquilibres génomiques Chr 6 et Chr
11 FISH 123 121 53 55 13 Fixateur inadéquat 8 4 18 93
gliome LOH 1p et 19q FISH 34 17 1 14 2 34 0 0 0
méthylation de MGMT methyl-specific PCR 232 232 83 149 193 7 32
Mutation de IDH1 et IDH2
neuroblastome amplification de Nmyc
1 Si deux techniques distinctes
2 Préciser les localisations tumorales et le biomarqueur (créer autant de lignes que nécessaire).
3 Préciser les localisations tumorales.
Niveau
interré
g
ional
CH Ets privés
Activité pour des
établissements
dépendant d'une
autre plateforme
CGH array3FISH 58 46 10 1 7 40
mutations UGT1A1 Pyroséquençage
mode S
QA
22 22 20 1 1
mutations TPMT RFLP 5 5 5
1 Si deux techni
q
ues distinctes
2
Préciser les localisations tumorales et le biomarqueur (créer autant de lignes que nécessaire)
.
3
Préciser les localisations tumorales.
Pharmacogénétique
constitutionnelle
Nombre de
patients
Mélanomes
glioblastome
Test : Gène/marqueur rechercé Technique
utilisée1
Nombre
d’examens
effectués en
2011
Nombre de
prescriptions
établissements
de la plateforme
Nombre de prescriptions établissements hors
plateforme
Niveau régional
amp
lifi
ca
ti
on
MDM2/CDK4
 6
6
 7
7
1
/
7
100%