Détection des transferts horizontaux de gènes

Détection des transferts horizontaux de gènes
12 avril 2016
Alix Boc
Université du Québec à Montréal

❑Phylogénie
❑Problématique
❑Détection des transferts horizontaux de gènes complets
❑Détection des transferts horizontaux de gènes partiels
❑Biolinguistique
❑Conclusion
SOMMAIRE
BIF7002 - Séminaire de bioinformatique
Alix Boc

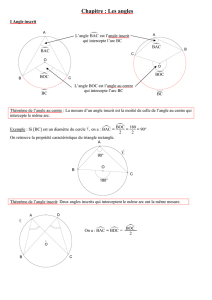
TRANSFERTS HORIZONTAUX DU GÈNE RPL12E
Pyrobaculum aerophilum
Aeropyrum pernix
Sulfolobus solfataricus
Pyrococcus furiosus
Pyrococcus abyssi
Pyrococcus horikoshii
Methanococcus jannaschii
Methanobacterium thermoautotrophicum
Archaeoglobus fulgidus
Methanosarcina barkeri
Haloarcula marismortui
Halobacterium sp.
Thermoplasma acidophilum
Ferroplasma acidarinanus
Pyrobaculum aerophilum
Aeropyrum pernix
Sulfolobus solfataricus
Pyrococcus furiosus
Pyrococcus abyssi
Pyrococcus horikoshii
Methanococcus jannaschii
Methanobacterium thermoautotrophicum
Archaeoglobus fulgidus
Methanosarcina barkeri
Haloarcula marismortui
Halobacterium sp.
Thermoplasma acidophilum
Ferroplasma acidarinanus
arbre d’espèces arbre du gène rpl12e
BIF7002 - Séminaire de bioinformatique
Alix Boc

Phylogénie
BIF7002 - Séminaire de bioinformatique
Alix Boc

ancêtres virtuels
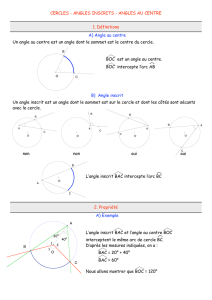
LA PHYLOGÉNIE
racine
branches
feuilles (espèces)
La phylogénie (ou phylogénèse) étudie la parenté entre différents êtres vivants
en vue de comprendre leur évolution.
BIF7002 - Séminaire de bioinformatique
Alix Boc
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
1
/
31
100%