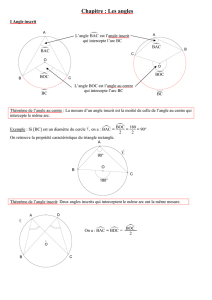
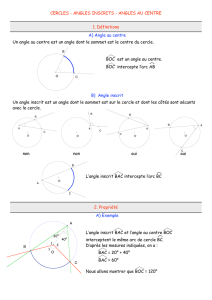
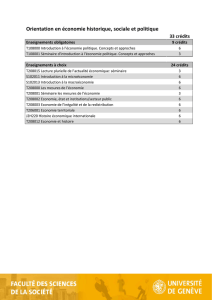
Détection des transferts horizontaux de gènes - LabUnix

Détection des transferts horizontaux de gènes :
modèles et algorithmes appliqués à l’évolution des
espèces et des langues
Alix Boc
Université du Québec à Montréal
Bif7002 –Séminaire de Bioinformatique
BIF7002 –Séminaire de Bioinformatique Alix Boc

BIF7002 –Séminaire de Bioinformatique 2 Alix Boc
Sommaire
Reconstruction des arbres phylogénétiques
Transferts horizontaux de gènes
Détection des transferts de gènes complets
Détection des transferts de gènes partiels
Application en biolinguistique
Travaux futurs

Inf7212 - Introduction aux systèmes informatiques
Bif7002 - Séminaire Bioinformatique Alix Boc
Reconstruction des arbres phylogénétiques

Bif7002 - Séminaire Bioinformatique 4 Alix Boc
L’arbre phylogénétique
Inf7212 - Introduction aux systèmes informatiques
racine
ancêtres virtuels
espèces
branches

Reconstruction d’un arbre phylogénétique
AAATGATCTGCGTCAATATTATAA
GCCTGATCCTCACTACTGTCATCTTAA
ATAGGGCCCGTATTTACCCTATAG
AACTGGTCCACCCTTATACTAAAAGACGCCTCACTAGGAAGCTAA
AACTGATCTGCTTCAATAATTTAA
AAATGATCTGCGTCAATATTA---------------------TAA
GCCTGATCCTCACTA------------------CTGTCATCTTAA
ATA---------------------GGGCCCGTATTTACCCTATAG
AACTGGTCCACCCTTATACTAAAAGACGCCTCACTAGGAAGCTAA
AACTGATCTGCTTCAATAATT---------------------TAA
Alignement de séquences
•ClustalW (Higgins et al., 1994)
•DiAlign (Morgenstern, 1999)
• ….
•Distances
•Maximum de parcimonie
•Maximum de vraisemblance
•Approche Bayesienne
Bif7002 - Séminaire Bioinformatique 5 Alix Boc
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
1
/
35
100%