gfmom td 1 levure

TD##GFMOM#Levure#1# # 14/03/12#
Levure comme organisme modèle :
Cycle cellulaire ; cytokinèse ; recombinaison ; réplication ; réparation ; architecture
nucléaire ; chromatine ; transcription ; métabolisme énergétique ; trafic ; traduction ; génome ;
stabilité ; évolution ; ect
Les#prions#de#levures#qui#sont#très#nombreux#(20),#utilisation#de#composer#pour#lutter#
contre# eux#:# protéine# infectieuse,# agent# responsable# de# la# vache# folle=# tremblante# du#
mouton.#Protéine#de#l’organisme#PRP,#prend#différent#état#:#normal#ou#prion.#Quand#il#y#
a#rencontre#entre#une#protéine#normale#et#une#protéine#prion,#la#protéine#prion#oblige#la#
protéine# normale# à# changer# de# conformation,# accumulation# protéine# prion,# c’est# à#
l’origine#de# la#maladie.# Il#n’y# a#pas# d’acide#nucléique,# c’est#uniquement# les#interactions#
entre#les#protéines.#La#levure#est#utilisé#en#organisme#modèle,#délétion#rapide,#sélection#
de#mutant#et#trouver#de#nouveau#partenaire#dans#un#organisme.#
#
!"#"$%&'() *%+(,$)-) ,./00%&'() 1# on# s’intéresse# à# la# réparation# ou# à# la# stabilité# des#
génomes,# recherche# de# mutant# par# mutagénèse,# identifier# de# nouveaux# gènes# pour#
complexifier#le#schéma.#On#part#d’une#voie,#d’un#phénomène# qui#nous#intéresse#et#l’on#
recherche# de# nouveaux# mutants# pour# identifier# de# nouveaux# acteurs.# Sélection# par#
crible.#
#
!"#"$%&'()%#2(+0()1#gène#qui#nous#intéresse#et#l’on#veut#savoir#dans#quoi#intervient#la#
protéine#codée#par#ce#gène.#Dans#quel#mécanisme#la#protéine#est#impliquée,#cela#passe#
par# la# délétion# du# gène,# et# ensuite# tester# une# batterie# de# phénotype# pour# voir# ou# va#
intervenir#cette#protéine.#
#
3/4.5*%4.56%5#$%&'()1# la# levure# est# capable# de# se# multiplier# à# l’état# haploïde,#
conjugaison#V>#obtention#diploïde.#Multiplication,#obtenir#des#clones#à#l’état#diploïde#et#
par#la#suite#il#y#a#la#méiose,#la#sporulation#qui#permet#de#revenir#à#l’état#haploïde.##
#
#
789:;<);)
#
="$(+>%#(?) ,5>6%(#) *() 0%$(0) 05#$) >%0) (#) @(') */#0) ,A/,'#) *(0) ,/+/,$B+(0)
,5#0%*"+"0C)<#).(0)4+(#/#$)*('D)E)*('DF)"$/6.%00(?)./),/+$()G"#"$%&'()*(),(0)0%$(0C)
)
a#et# α#sont# les#types#sexuels.#On#ne#peux#jamais#avoir#de#diploïde#homozygote#(a/a#ou#
α/α)##
#
Il#faut#déjà#monter#chacun#des#caractères,#qui#sont#contrôlés#par#un#seul#site.#
On#prend#les#caractères#un#par#un.#
#
Hypothèse#1#:#Dans#ce#croisement,#la#prototrophie#pour#le#tryptophane#est#contrôlé#par#
un#site#ou#un#gène#appelé#TRP1#avec#deux#allèles#trp1V1#et#TRP1.#
#
On#ne#précise#pas#si#le#site#ou#le#gène#était#situé#sur#un#autosome#ou#un#chromosome#X#car#
cela# n’existe# pas# dans# la# levure.# Pas# de# chromosome# sexuel# dans# la# levure# mais# un# locus#
sexuel# qui# est# sur# un# chromosome# mais# l’autre# partenaire# a# le# même,# il# y# a# juste# une#
différence#au#niveau#du#locus#sexuel,#il#n’y#a#pas#de#chromosome#sexuel#comme#on#l’entend#
chez#les#animaux.#

TD##GFMOM#Levure#1# # 14/03/12#
#
B# trp#1V1### [TRPV]######X# C# TRP1# # [TRP+]#
#
#
#
2n# trp#1V1###########[#TRP+]#
############################################################################TRP1#
#
TRP1#>#Trp#1V1#
Le# phénotype# associé# à# l’allèle# TRP1# est# dominant# sur# le# phénotype# associé# à# l’allèle#
trp1V1.#
#
#
50%#trp1V1###[TRPV]#
50%#TRP1#####[TRP#+]#
#
Analyse#sur#1000#spores#
#
Effectif#théorique#
Effectif#observé#
[TRP#+#]#
500#
501#
[TRP#V#]#
500#
499#
#
On# constate# que# les# effectifs# ne# sont# pas# significativement# différents,# hypothèse# de# un#
site#est#donc#accepté.#
#
Hypothèse#2#:#Dans#ce#croisement,#la#prototrophie#pour#la# méthionine#est#contrôlé#par#
un#site#ou#un#gène#appelé#MET3#avec#deux#allèles#met3V1#et#MET3.#
#
#
#
B# MET3### [MET+]### #####X# C# met#3V1# # [METV]#
#
#
#
2n# met#3V1###########[MET+]#
##########################################################################MET3#
#
MET3#>#met#3V1#
Le#phénotype#associé#à#l’allèle#MET3#est#dominant#sur#le#phénotype#associé#à#l’allèle#met#
3V1.#
#
#
50%#met#3V1###[METV]#
50%#MET3#####[MET+]#
#
#
#
Sporulation#
Sporulation#

TD##GFMOM#Levure#1# # 14/03/12#
#
1000#spores#
Effectif#théorique#
Effectif#observé#
[MET#+]#
500#
497#
[MET#V]#
500#
503#
#
#
On# constate# que# les# effectifs# ne# sont# pas# significativement# différents,# hypothèse# de# un#
site#est#donc#accepté.#
#
Hypothèse#3#:#Idem#pour#la#thermo#sensibilité,#on#conclut#à#un#seul#site.#
#
Etude#de#liaison#:#indépendant#ou#liés# # # #
Il#y#a#3#façons#de#faire#:#
#
1/#Test#de#l’indépendance#
#
trp#1V1# MET3############X## TRP1# # met#3V1#
#
#####
#
trp#1V1# # MET#3#
# # # # TRP1# # # met#3V1#
#
#
Trp#1V1# MET3# # [TRP#V##MET#+]#
TRP#1# # met#3V1# [TRP#+#MET#V]#
Trp#1V1# met#3V1# [TRP#–#MET#V]#
TRP1# # MET3# # [TRP+#MET+]#
#
Si#il#y#a#indépendance#alors#on#s’attend#à#25%#de#chacune#des#catégories.#
1000#spores#
Effectif#théorique#
Effectif#observé#
TRPV######MET+#
250#
401#
TRP+#####METV#
250#
405#
TRPV######METV#
250#
98#
TRP+#####MET+#
250#
96#
#
Les#résultats#observés#et#théoriques#sont#significativement#différents#donc#les#sites#sont#
liés.#On#peut#calculer#une#distance.#
#
Distance#=######Nombre#de#recombinés#x#100##=## (98+96)#x#100##=##19,4#cM#
### # ######Nombre#total# # # #######1000#
#
#
#
PARENTAUX#
RECOMBINES#
#

TD##GFMOM#Levure#1# # 14/03/12#
2/#Calcul#de#fréquence#
#
Frec#=##Nombre#de#recombinant###=##96+98##=###0,194#
### ##########Nombre#total# # ###1000##
#
La#fréquence#est#comprise#entre#0#et#1.#
Si#fréquence#de#recombinaison#est#d’environ#0,50#alors#il#y#a#indépendance.#
Si# fréquence# de# recombinaison# est# inférieur# à# 0,5,# les# gènes# sont# liés,# on# calcul# la#
distance.#
On#multiplie#par#100#pour#obtenir#des#cM.#
Ici#les#sites#sont#liés#0,194#<#0,5.#
#
3/#Ici#on#a#Parentaux#>#Recombinés##(401+405#>#96+98)#
Les#2#sites#sont#liés,#on#peut#calculer#la#distance.#
#
#
#
Distance#TRP1#–#TS1#=#[(75+65+102)#x#100]#/#1000#=#14#cM#
Distance#MET3#V#TS1#=#[(1+2+31+26)#x#100]#/#1000#=#6#cM#
#
#
Carte#génétique#
#
#
#
19,4#est#différent#de#6+14,#on#a#négligé#les#doubles#recombinants.#Deuxième#et#troisième#
ligne#du#tableau.#
#
Taille#du#génome#de#Saccharomyces#cerevisiae#=#12/13#Mb,#6#600#gènes.#
1#cM#=#2#kb#dans#la#levure.##
#
#
#
#
#

TD##GFMOM#Levure#1# # 14/03/12#
#
789:;<);;)
#
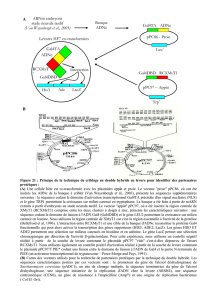
8).H/%*()*H'#)0,A">/F)*",+%2(?).()5').(0)"2"#(>(#$I0J)45'2/#$)K$+()E).H5+%G%#()*()
,($$()05',A()$+/#0L5+>"(C)
#
On#part#du#gène#et#on#essaye#de#déterminer#le#rôle#de#la#protéine#codée#par#le#gène.#
#
met3+#=#MET3+#
#
Transformation,#on#veut#obtenir#la#délétion#de#t.#Délétion#de#la#partie#codante.#
Pour#faire#une#recombinaison#dans#la#levure,#il#faut#30#pb#pour#que#la#levure#soit#capable#
de#faire# de#la#recombinaison.#Puis#on#réalise#une#PCR,#le# produit#contient# le#marqueur#
LEU2#complet#plus#de#chaque#côté#les#régions.#(bleu#et#vert#sur#le#schéma).#Il#y#a#donc#
possibilité# de# recombinaison.# On# obtient# une# souche# délétée# pour# t.# On# a# laissé# LEU2#
sous#son#propre#promoteur#et#non#sous#le#promoteur#de#t#car#on#ne#sait#pas#quand#il#est#
exprimé.# Or# on# veut# que# de# l’enzyme# LEU2# soit# toujours# produite# pour# obtenir# une#
souche# prototrophe# pour# la# leucine.# Avec# toujours# les# régions# homologues.# Obtention#
d’un#hétérozygote#LEU2#et#leu2V1,#on#obtient#une#souche#[LEU+].#
#
#
#
Autres#types#de#transformant#:#[LEU+]#
#
V recombinaison#homologue#au#locus#LEU2#
#
#
#
Efficacité# de# la# recombinaison# dépend# de# la# taille# des# régions# homologues,# ici# régions#
beaucoup#plus#importante#donc#on#s’attend#à#avoir#une#efficacité#plus#importante.#
#
V recombinaison#non#homologue#
Recombinaison#n’importe#ou#dans#le#génome.##
#
#
PCR#sur#le#fragment#contenant#t#est#une#bonne#méthode#mais#on#n’utilise#pas#de#grand#
fragment.#Le#Southern#Blot#est#là#pour#vérifier#l’amplification#par#sonde.##
#
Pourquoi# des# diploïdes#?# Car# l’on# peut# déleter# un# gène# essentiel# à# la# survie#
contrairement# aux# haploïdes# ou# l’on# obtiendrais# que# des# recombinaisons# non#
homologues.#Attention#à#la#délétion#des#gènes.#
#
 6
6
 7
7
 8
8
1
/
8
100%