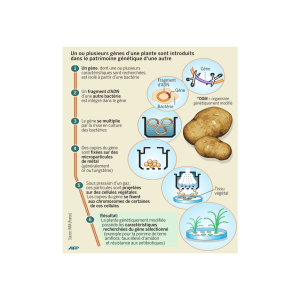
Figure 21 : Principe de la technique de criblage en double hybride

Figure 21 : Principe de la technique de criblage en double hybride en levure pour identifier des partenaires
protéiques :
(A) Une cellule hôte est co-transformée avec les plasmides appât et proie. Le vecteur "proie" pPC86, où ont été
insérés les ADNc de la banque à cribler (Van Wayenbergh et al., 2003), présente les séquences supplémentaires
suivantes : la séquence codant le domaine d'activation transcriptionnel Gal4TA précédée d'un signal nucléaire (NLS)
et le gène TRP1 permettant la croissance sur milieu carencé en tryptophane. La banque a été faite à partir de mARN
extraits à partir d'embryons au stade neurula tardif. Le vecteur "appât" pPC97, où a été insérée la région centrale de
XMyT1 (RCXMyT1) comprise entre les deux clusters à doigts à zinc, présente les caractéristiques suivantes : une
séquence codant le domaine de liaison à l'ADN Gal4 (Gal4DBD) et le gène LEU2 permettant la croissance sur milieu
carencé en leucine. Nous utilisons la région centrale de XMyT1 car c'est la région essentielle à l'activité de la protéine
(Bellefroid et al, 1996). L'interaction entre RCXMyT1 et une cible de la banque d'ADNc reconstitue la protéine Gal4
fonctionnelle qui peut alors activer la transcription des gènes rapporteurs (HIS3, ADE2, LacZ). Les gènes HIS3 ET
ADE2 permettent une sélection sur milieux carencés en histidine et en adénine. Le gène LacZ permet une sélection
chromogénique par détection de l'activité -galactosidase. Pour cette expérience, nous utilisons un contrôle négatif
réalisé à partir de la souche de levure contenant le plasmide pPC97 "vide" c'est-à-dire dépourvu de l'insert
RCXMyT1. Nous utilisons également un contrôle positif d'activation réalisé à partir de la souche de levure contenant
le plasmide pPC97/T PIE* codant une fusion entre le domaine de liaison à l'ADN de Gal4 et la partie N-terminale de
PIE8 (un activateur transcriptionnel de trypanosome – Perez-Morga and Pays, 1991).
(B) Cartes des vecteurs utilisés pour la recherche de partenaires protéiques par la technique du double hybride. Les
séquences caractéristiques portées par ces vecteurs sont : le promoteur du gène de l'alcool déshydrogénase de
Saccharomyces cerevisiae (pADC1), un site de clonage multiple, la séquence terminatrice du gène de l'alcool
déshydrogénase, une séquence initiatrice de la réplication d'ADN chez la levure (ARSH4), une séquence
centromérique (CEN6), un gène de résistance à l'ampicilline (AmpR) et une origine de réplication bactérienne
( Co1E1 Ori).
Leu+
pPC86 - Proie
ADNc
Gal4TA
Banque
ADNc
ARNm embryons
stade neurula tardif
(Van Wayenbergh et al., 2003)
Interaction
His3
Ade
LacZ
Gal4DBD
Gal4TA
RCXMyT
1
ADNc
Levures HF7 co-transformées
pPC97 – Appât
Gal4DBD
Trp+
RCXMyT1
A
B
1
/
1
100%