VOCABULAIRE

Correspondances en Onco-Théranostic - Vol. III - n° 1 - janvier-février-mars 2014
9
dossier thématique
NGS
VOCABULAIREVOCABULAIRE
INTRODUCTION
D
ix ans après le séquençage complet du génome
humain, les progrès technologiques des
méthodes de séquençage et des outils bio-
informatiques d’analyse permettent le décryptage
des altérations génomiques survenant au cours du
développement tumoral. Aujourd’hui, le séquençage
du génome tumoral, ciblé sur certaines régions, élargi
à l’ensemble des séquences codantes, voire à la totalité
du génome, est devenu réalité. Toutes les formes de
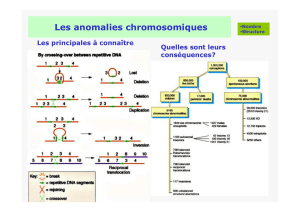
modification de l’ADN peuvent être identifiées : chan-
gements de séquence (mutations), anomalies de struc-
tures (translocations), modifications de nombre (gains,
délétions, amplifications). De plus, le séquençage de
l’ARN est une nouvelle et puissante méthode d’ana-
lyse du transcriptome qui est en train de remplacer les
hybridations sur puces de l’ARN.
De grandes initiatives nationales (The Cancer Genome
Atlas [TCGA], cancergenome.nih.gov) et internatio-
nales (International Cancer Genome Consortium [ICGC],
icgc.org) ont mutualisé les efforts de la communauté
scientifique pour constituer des collections de tumeurs,
permettre leur analyse et rendre les résultats acces-
sibles. Les principales études publiées au cours des
3 dernières années ont porté sur les cancers les plus
fréquents (cancers du sein, du côlon, du poumon, etc.),
les cancers consécutifs à des expositions spécifiques
(cancer du foie, etc.) ou des cancers “modèles” pour
l’étude de l’oncogenèse (cancers du rein, tumeurs héma-
tologiques, etc.) [figure, p. 10].
Ces études dressent le portrait complexe des altéra-
tions génomiques des tumeurs. Elles permettent de
reconstituer l’histoire de la tumeur et de mettre en
évidence l’existence d’une hétérogénéité moléculaire
intratumorale. Elles montrent que, dans chaque tumeur,
il existe potentiellement des mutations fondatrices,
nécessaires mais non suffisantes à la transformation
de la cellule, quelques gènes porteurs de mutations
Le séquençage massif
parallèle ou l’exploration
du panorama génomique
des tumeurs : une “kataegis1”
d’informations !
Anne Vincent-Salomon*, Karen Leroy**
* Institut Curie, département de biopathologie, Paris.
** Inserm U955, département de pathologie, hôpital Henri-Mondor, Créteil.
1 Pluie forte et soudaine,
en grec.
“driver”, ou “motrices”, ayant un impact fonctionnel
sur le développement de la tumeur, et, éventuelle-
ment, des mutations “accélératrices” intervenant dans
la progression tumorale, accompagnées par de très
nombreuses anomalies génomiques (mutations pas-
sagères), dont la signification n’est pas connue et qui
sont probablement uniquement observées dans cette
tumeur. La description progressive des mécanismes de
la résistance aux traitements cytotoxiques classiques
ainsi qu’aux thérapies ciblées constitue un autre aspect
très intéressant des analyses de séquençage massif : par
exemple, la mise en évidence de mutations du gène
ESR1 après traitement par antiaromatase des cancers
du sein métastatiques (2).
La comparaison des altérations génomiques observées
dans les différents types tumoraux met en évidence des
altérations récurrentes et élargit la compréhension des
mécanismes de la carcinogenèse. Ainsi, des altérations
génomiques survenant dans des gènes impliqués dans
des processus biologiques jusque-là peu associés à la
tumorigenèse sont identifiées, par exemple celles sur-
venant dans des gènes régulant l’épissage des ARN, le
métabolisme ou le remodelage de la chromatine.
Comparer la fréquence des altérations génomiques dans
les différents types de cancers permet de dresser des
signatures mutationnelles spécifiques à chaque organe
ou de décrire les associations et exclusions mutuelles
de mutations dans certains gènes (3).
La comparaison de la nature des gènes mutés et
des types de mutations somatiques survenant dans
les tumeurs permet aussi de décrypter les causes
éventuelles de ces anomalies génomiques, comme
la signature attribuée aux APOBEC (apolipoprotein B
mRNA editing enzyme, catalytic polypeptide-like), des
cytidine désaminases dans les cancers du sein (4).
D’autres signatures mutationnelles sont dues à des
carcinogènes connus (rayons ultraviolets, tabac, etc.)

Correspondances en Onco-Théranostic - Vol. III - n° 1 - janvier-février-mars 2014
10
dossier thématique
NGS
Figure. Nombres médians de mutations non silencieuses (non synonymes) observées dans les types tumoraux des organes
principaux. Schéma adapté de B. Vogelstein et al. (1).
Colorectal (MSI)
Poumon (CBPC)
Poumon (CBNPC)
Mélanome
Carcinome épidermoïde de l’œsophage
Lymphome non hodgkinien
Colorectal (MSS)
Tête et cou
Adénocarcinome de l’œsophage
Gastrique
Carcinome endométrioïde de l’endomètre
Adénocarcinome du pancréas
Ovarien (séreux de haut grade)
Prostate
Hépatocellulaire
Glioblastome
Sein
Endomètre (séreux)
Poumon (CBNPC du non-fumeur)
Leucémie lymphoïde chronique
Leucémie myéloïde aiguë
Glioblastome
Neuroblastome
Leucémie lymphoblastique aiguë
Médulloblastome
Tumeur rhabdoïde
1 500
1 000
500
250
225
200
175
150
125
100
75
50
25
0
Nombre de mutations non synonymes par tumeur
(médiane ± 1 quartile)
Cancer des voies aérodigestives
supérieures (66)
Cancer du poumon
(non à petites cellules) 147
Cancer du poumon
(à petites cellules) 163
Adénocarcinome de l’œsophage (57)
Carcinome épidermoïde
de l’œsophage (79)
Cancer gastrique (53)
Cancer colorectal (66)
Cancer de l’endomètre (49)
Cancer de la prostate (41)
Mélanome (135)
Leucémie lymphoïde
aiguë (8)
Leucémie
lymphoblastique
aiguë (11)
Leucémie
chronique myéloïde (12)
Cancer du
pancréas(45)
Cancer
hépatocellulaire (89)
Cancer du sein (33)
Neuroblastome (12)
Tumeur
rhabdoïde (4)
Lymphome non
hodgkinien (74)
Glioblastome (35)
Glioblastome (14)
Médulloblastome (8)
Cancer de l’ovaire (42)

Correspondances en Onco-Théranostic - Vol. III - n° 1 - janvier-février-mars 2014
11
Le séquençage massif parallèle ou l’exploration du panorama génomique des tumeurs :
une “kataegis1” d’informations !
ou à des anomalies de la maintenance du génome.
Néanmoins, la majorité reste sans explication.
Au total, à l’heure actuelle, il semble que les mutations
des séquences codantes d’environ 200 gènes contri-
buent au développement des cancers (driver genes).
Leur sont probablement associées des anomalies de
l’expression de certains gènes consécutives à des ano-
malies des mécanismes de régulation épigénétique
(“épigènes”), ces mécanismes étant plus délicats à
identifier.
ALT: allongement alternatif des télomères. Processus de mainte-
nance de la longueur des télomères indépendant de la télomérase
(enzyme responsable de l’allongement des télomères).
Amplification : altération génomique correspondant à l’augmen-
tation du nombre de copies d’une courte région du génome (moins
de 1mégabase).
Délétion homozygote : délétion des 2 copies d’un gène, héritées
l’une de la mère, l’autre du père.
Driver gene : gène qui contient une mutation driver gene (cf. infra,
driver gene mutation) ou qui est exprimé d’une façon aberrante,
conférant un avantage en croissance (epi-driver gene).
Driver gene mutation : mutation qui confère, directement ou
indirectement, un avantage sélectif en croissance à la cellule dans
laquelle elle survient.
Epi-driver gene : gène qui est exprimé d’une façon aberrante,
conférant un avantage en croissance.
Épigénétique : changement dans l’expression des gènes ou dans
le phénotype cellulaire, causé par des mécanismes autres que des
changements de séquence de l’ADN.
Exome : ensemble des exons dans le génome humain. L’“exome
sequencing” concerne cet ensemble, qui représente l’ensemble des
séquences codant pour des protéines.
Gatekeeper : gène “garde-fou” qui, quand il est inactivé par mutation,
initie la tumorigenèse. Par exemple : le gène Rb est inactivé par muta-
tion. Cette mutation inactivatrice est à l’origine du développement
du rétinoblastome ; celle de VHL, à l’origine de tumeurs du rein.
Gène suppresseur de tumeur : gène qui, quand il est inactivé
par mutation dans une cellule, lui confère un avantage sélectif en
croissance.
Indel : mutation due à l’insertion ou à la délétion d’un petit nombre
de nucléotides.
Méthylation : addition covalente d’un groupement méthyle à une
protéine ou à une base de l’ADN.
Mutation clonale : mutation présente dans la majorité des cellules
néoplasiques d’une même tumeur.
Mutation faux-sens : substitution d’un seul nucléotide qui aboutit
à la substitution d’un acide aminé.
Mutation germinale : mutation du génome d’un individu héritée de
l’un des 2 parents et présente dans toutes les cellules de l’organisme.
Mutation non-sens : substitution d’un seul nucléotide qui aboutit
à la production d’un codon stop.
Mutation non synonyme : mutation qui altère la séquence codant
pour les acides aminés d’une protéine. Cela inclut les mutations faux-
sens ou non-sens, les sites d’épissage, les séquences des sites d’ini-
tiation de la transcription ou des codons stop, et les mutations indel.
Mutation somatique : mutation qui se produit dans n’importe
quelle cellule du corps après la fécondation, comme celles survenant
au cours du déclenchement de la tumorigenèse.
Mutation sous-clonale : mutation qui n’est présente que dans un
sous-ensemble des cellules de la tumeur.
Oncogène : gène qui, quand il est activé par mutation, confère un
avantage sélectif en croissance à la cellule dans laquelle ce gène
est exprimé.
Passenger mutation : mutation qui n’a pas d’impact sur l’avantage
en croissance de la cellule dans laquelle elle se produit.
Promoteur : région qui régule l’initiation de la transcription (l’expres-
sion) d’un gène.
Réarrangement : mutation qui juxtapose des séquences nucléo-
tidiques normalement séparées (sur 2 chromosomes différents ou
sur le même chromosome).
Substitution d’une seule base : modification d’un seul nucléotide
par rapport à la séquence de référence.
Site d’épissage : courte séquence nucléotidique juxtaposée aux
exons et qui dirige, orchestre, l’épissage des exons.
Translocation : réarrangement spécifique joignant 2 segments de
chromosomes non homologues.
Variants germinaux : variations d’une séquence observées entre
différents individus. Deux individus choisis au hasard présentent
environ 20 000différences génétiques, réparties sur l’ensemble
du génome.
Lexique pour faciliter la compréhension du dossier
et de l’univers du séquençage parallèle massif
(la formulation anglaise, indiquée en italique, a été conservée dans certains cas)
A. Vincent-Salomon
déclare ne pas avoir
deliens d’intérêts
concernant le sujet
decette introduction.
K. Leroy déclare ne pas
avoir de liens d’intérêts.
En conclusion, les objectifs du séquençage parallèle
massif sont bien d’améliorer la connaissance de la bio-
logie des cancers, mais aussi, à terme, d’imaginer de
nouvelles stratégies de prévention, de diagnostic et de
traitement. Il apparaît aussi clairement que certaines
régions du génome sont le siège d’une accumulation
majeure d’altérations génomiques, que décrit bien le
terme de kataegis. Reste à comprendre la signification
biologique de l’ensemble de ces altérations ! C’est l’enjeu
des années à venir, qui s’annoncent passionnantes. ■
1.
Vogelstein B, Papadopoulos
N, Velculescu VE, Zhou S, Diaz
LA Jr, Kinzler KW. Cancer
genome landscapes. Science
2013;339(6127):1546-58.
2.
Toy W1, Shen Y, Won H et al.
ESR1 ligand-binding domain
mutations in hormone-resis-
tant breast cancer. Nat Genet
2013;45(12):1439-45.
3. Watson IR, Takahashi K,
Futreal PA, Chin L. Emerging
patterns of somatic muta-
tions in cancer. Nat Rev Genet
2013;14(10):703-18.
4. Nik-Zainal S, Alexandrov LB,
Wedge DC et al. Mutational
processes molding the
genomes of 21 breast cancers.
Cell 2012;149(5):979-93.
Références
1
/
3
100%