Eléments de correction du DS n°4 2016 2017

1
BCPST 1 Lundi 31janvier 2017
DS n°4 : éléments de correction
Partie 1 : Géologie
Exercice 1. Étude de la carte d'Aurignac 1/50 000
1. Coupe
Penser à compléter la légende pour indiquer les figurés correspondant à la coupe.
Attention à :
- L’épaisseur des couches : en général constante
- Respecter l’orientation des couches en plaçant les figurés
- Choisir des figurés pertinents (les croix sont en général utilisées pour des roches plutoniques)
- Bien repérer et représenter les formations en discordance sur les terrains sous-jacents
- (inutile de représenter les alluvions actuelles)
2. Frise chronologique
Géodynamique interne
11. érosion
10. épandage torrentiel en domaine continental : dépôts P
9. érosion
OROGENESE chaîne de montagnes 8. épandage torrentiel en domaine continental : dépôts m3
7. régression émersion et érosion
6. sédimentation m2 : retour d’un domaine marin ou continental
5. régression émersion et érosion
4. sédimentation e2-1 : sédiments + grossiers début régression
3. failles
TECTONIQUE 2. plissement
1. sédimentation (c8a eII-III) en domaine marin
Histoire géologique de la région d' Aurignac
En gras, ce qui est attendu. La reconstitution des grands traits de l'histoire géologique de la région nécessite de
regarder toute la carte et pas seulement les terrains situés sous le trait de coupe (de même pour réaliser
correctement la coupe).
Penser à :
- mettre un titre,
- séparer les processus relevant de la géodynamique externe de ceux relevant de la géodynamique interne,
- regrouper les couches ayant sédimenté dans une continuité au lieu de les énumérer une à une,
- indiquer l'abréviation de la couche (ex : e2-1)
Justification non demandée (cf question posée) :
Tous les terrains du c8a (Cénomanien) au eII-III (Lutétien inf et Yprésien) sont plissés : il y a d'abord eu sédimentation
(principe de superposition) puis plissement (principe de recoupement). La faille recoupe le plissement donc elle lui
est postérieure (principe de recoupement).
Les sédiments m² recouvrent des roches d'âges différents toutes plissées, sans continuité avec les structures pré-
existantes : il y a eu émersion et érosion puis dépôt de m² dans un domaine marin ou continental, en discordance sur
les sédiments e2-1 (Eocène).
Les dépôts m3 sont discordants sur m² : leur mise en place est précédée d’une nouvelle émersion et érosion. Les
dépôts m3 et P correspondent à un « épandage de nature torrentiel » : ce sont des dépôts laissés par des torrents, on
est donc en domaine continental.
Les dépôts P sont discordants sur m3 : il y a eu érosion entre ces deux dépôts.
N.B. Avec les indications données (c : Crétacé, e : Eocène, m : Miocène) il manque l'Oligocène. Il était donc possible
de faire figurer une lacune au sein de cette série. En réalité, les terrains de c8a à eII-III constituent un continuum
Crétacé, Oligocène, Miocène.
Géodynamique externe

2
Exercice 2. Établir une chronologie relative à l'échelle de l'affleurement
Attention à l’utilisation des principes de stratigraphie aux roches magmatiques (ex : principe de superposition non
applicable pour des roches plutoniques).
Observations et principes
Interprétation
Des filons de basalte dans la dolérite et le gabbro
principe de recoupement
Les filons de basalte sont postérieurs au gabbro et à la dolérite
Une petite faille décale le contact gabbro-dolérite
principe de recoupement
La faille est postérieure à la mise en place du gabbro et de la
dolérite
Une petite faille décale les filons de basalte dans le gabbro
principe de recoupement
La faille est postérieure aux filons de basalte
Les minéraux du gabbro sont allongés
principe de déformation continue
Le gabbro a été déformé après sa mise en place
Des minéraux allongés du gabbro sont coupés au contact avec
la dolérite
principe de recoupement
La mise en place de la dolérite est postérieure à celle du
gabbro
Bilan : 1. mise en place du gabbro puis déformation du gabbro
2. mise en place de la dolérite
3. mise en place des filons de basalte
4. faille
Exercice 3. Datation isotopique d'un granite du Massif Central
1. Justifiez l’utilisation de ces éléments chimiques comme géochronomètre, et indiquez quelles sont les
conditions et limites d’utilisation des méthodes de datation absolue.
87Rb, isotope instable du rubidium, se désintègre en 87Sr, isotope stable du strontium. Les quantités de ces isotopes
variant au cours du temps indépendamment des conditions thermodynamiques, on peut les utiliser comme
géochronomètre.
La quantité d’élément père varie selon l’équation : P = P0 e -λt
avec :
P : quantité d’élément père au temps t et P0 : quantité d’élément père à t=0
t temps écoulé depuis la cristallisation de la roche (= fermeture du système)
λ constante de désintégration spécifique du couple considéré = 1,42.10-11a-1
L’équation reliant la quantité d’élément père et la quantité d’élément fils au cours du temps est la suivante :
F = F0 + (P0 – P) = F0 + P (e λt -1)
Les conditions et limites d’utilisation sont les suivantes :
L’élément doit être contenu dans la roche.
La période du couple doit être compatible avec l'évènement à dater (âge à déterminer compris entre T/100 et 10T).
On date la fermeture du système c'est à dire le dernier événement ayant affecté la roche ou le minéral.
La méthode s'applique bien aux roches magmatiques, avec précaution aux roches métamorphiques, mais pas aux
roches sédimentaires qui sont des systèmes ouverts.
2. Etablissez la relation entre ces rapports isotopiques et l’âge d’un échantillon.
Soit l’équation générale : F = F0 + P (e λt -1)
Dans le cas du couple Rb/Sr, F0 est inconnu. On transforme alors la relation en :
87Sr/86Sr = [87Sr/86Sr]0 + 87Rb/86Sr (eλt -1)
86Sr0 est également inconnu, mais on montre sur des roches actuelles que le rapport [87Sr/86Sr]0 est le même pour
tous les minéraux d’une même roche : ce terme est donc une constante, appelée « b ».
87Sr/86Sr est mesuré, c’est la valeur de l’ordonnée, donc le terme « Y » de l’équation.
87Rb/86Sr est mesuré, c’est la valeur de l’abcisse, donc le terme « X » de l’équation.
L’équation devient alors : Y = b + Xa, c’est une équation de droite, avec a = (eλt -1) pente de la droite.
Le temps t qui correspond à l’âge de la roche pourra donc être déterminé graphiquement, à partir de la pente de la
droite.

3
3. Tracer l’isochrone à partir des données fournies sur l’encart de papier millimétré.
Indiquer axes – titre – pente sur le graphe.
4. Calculer l’âge de la roche étudiée.
a = λt d'où t = a / λ
Graphiquement la pente est la tangente de l'angle a, tangente a = Δy/ Δx soit a = 0,004
267 Ma (avec a = 0,004 et λ = 1,5)
333 Ma (avec a = 0,005 et λ = 1,5)
un âge de l’ordre de 300 Ma est attendu.
Partie 2 : Biologie
Thème 1 : Régulation de l’expression de l’information génétique chez les Eubactéries : l’opéron lactose
Cette partie propose de dégager quelques aspects du contrôle de l’expression génétique à partir de résultats
expérimentaux ; l’étude expérimentale a été réalisée chez Escherichia coli.
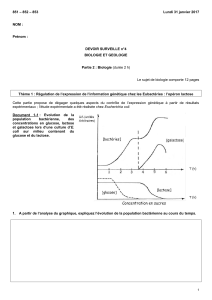
1. A partir de l’analyse du graphique, expliquez l’évolution de la population bactérienne au cours du temps.
Aucune modification n’est observée avant 1 h. De 1 à 3 h, la population bactérienne croît de manière exponentielle
puis on constate un palier. Dans le même temps, la concentration en glucose diminue (elle varie de manière
symétrique avec l’effectif de bactéries, et l’interruption de l’augmentation du nombre de bactéries coïncide avec la
disparition du glucose.
On peut en déduire que les bactéries consomment le glucose, ce qui leur permet de se multiplier.
A partir de 4 h la population bactérienne croît de nouveau, ce qui coïncide avec la diminution de la concentration en
lactose et l’apparition de galactose. La population bactérienne atteint un second palier qui coïncide avec la disparition
du lactose et la diminution de la concentration en galactose. Or le lactose est constitué de l’association d’une
molécule de glucose et d’une molécule de galactose.
On peut donc proposer qu’après l’épuisement du glucose, la population de bactérie devient capable – après un
temps de latence qui correspond au 1er palier – d’utiliser le lactose. Les bactéries hydrolysent le lactose, ce qui libère
le galactose dont la concentration augmente, et le glucose qu’elles utilisent pour se multiplier. La disparition du
galactose suggère que les bactéries le transforment.
Cette utilisation successive de deux substrats par les bactéries est qualifiée de diauxie.

4
2. D’après vos connaissances, indiquez ce qui conditionne l’utilisation du lactose par la population
bactérienne
Les bactéries deviennent capables d’utiliser le lactose à condition :
- que celui-ci soit présent dans le milieu : la présence de lactose lève l’inhibition de l’expression des gènes codant les
enzymes permettant son utilisation (l’opéron lactose),
- que le glucose soit absent : son absence entraîne un signal de carence alimentaire sous la forme d’une
augmentation de la concentration en AMPc qui, lié au complexe protéique CAP, augmente la transcription des gènes
de l’opéron lactose.
3. Présentez les objectifs des expériences puis déduisez de leur analyse la nature du contrôle exercé par le
lactose.
Objectifs : mettre en évidence l’effet du lactose sur le contrôle de la synthèse de la β-galactosidase et préciser l’étape
de la synthèse sur laquelle ce contrôle s’exerce.
Analyse et interprétation :
Dans les expériences A et B les bactéries sont successivement cultivées dans un milieu contenant du glucose puis
dans un milieu contenant du lactose.
- lorsque la méthionine marquée est ajoutée dans le milieu en même temps que le glucose, mais absente du milieu
avec le lactose, la β-galactosidase synthétisée n’est pas marquée.
- en revanche, lorsque la méthionine marquée est présente dans le milieu en même temps que le lactose, la β-
galactosidase synthétisée est marquée, elle a donc incorporé la méthionine marquée.
On peut en déduire que la synthèse de β-galactosidase a lieu lorsque le lactose est présent dans le milieu et non le
glucose : le lactose exerce un contrôle sur la synthèse de β-galactosidase.
Dans l’expérience C, les bactéries sont cultivées dans un milieu contenant du lactose et de la méthionine marquée,
mais aussi de la rifampicine qui inhibe la transcription. On constate alors qu’il n’y a pas de synthèse de β-
galactosidase malgré la présence de lactose.
En bloquant la transcription, la rifampicine empêche l’effet du lactose. On peut donc en déduire que le lactose exerce
son contrôle sur l’étape de transcription : il active l’expression du gène codant la β-galactosidase (et donc de l’opéron
lactose).
4. A l’aide de vos connaissances, schématisez la situation de la portion du génome concernée par le
contrôle dans le cas des bactéries à T= 4h30 (document 1).

5
Thème 2 : Anthocyanes et pigmentation des pièces florales
Extrait du sujet de l’épreuve B du concours Agro-Véto 2016
Cette partie étudie quelques mécanismes à l’origine de la pigmentation des pièces florales. Les fleurs du pétunia
(Petunia, Solanacée) constituent un des modèles utilisés car il existe de nombreux mutants de pigmentation des
pièces florales connus ou générés par les techniques de génie génétique. On s’intéresse aux rôles du gène
anthocyanin 1 dans la pigmentation des pièces florales.
1. Dégagez succinctement les principales tendances reliant la pigmentation et la valeur du pH.
Les corolles les plus pigmentées (clichés A, L, S, T) présentent un pH de l’ordre de 5.4.
Les corolles blanches (clichés C, E, N, P) présentent un pH de 6.0 à 6.1.
Les barres d’erreur ne se chevauchent pas : les différences entre pH des corolles pigmentées et non pigmentées
sont significatives.
En conclusion, ces deux paramètres semblent liés : plus la corolle est pigmentée, plus le pH est acide.
2. Formulez deux hypothèses concernant le rôle du gène anthocyanin 1 (an1).
La pigmentation de la corolle et le pH varient avec les allèles que possèdent les différents plants pour le gène an1.
On peut alors proposer deux hypothèses concernant le rôle de ce gène :
- les produits de l’expression de ce gène ont un effet sur le pH,
- les produits de l’expression de ce gène interviennent dans la synthèse des pigments.
3. A quelle famille de protéines peut appartenir la protéine AN1 ? Connaissez-vous d'autres domaines
appartenant à cette famille de protéines ?
Possédant un domaine de fixation à l’ADN de type bHBH, cette protéine pourrait appartenir à la famille des facteurs
de transcription, protéines dont la fixation à l’ADN module l’expression de gènes.
Ces protéines peuvent présenter d’autres domaines de fixation : domaine à fermeture à glissière de leucine, domaine
à doigts de zinc.
4. Analysez les résultats obtenus (document 2-2) et formulez une hypothèse permettant d'expliquer la
diversité des phénotypes O, Q, R et S (document 2-1) correspondant respectivement aux mutants W211,
W211R3, W211R2 et W211R1.
La comparaison des séquences protéiques du domaine HLH codé par l’allèle sauvage (R27wt) et des allèles mutés
montre des différences plus ou moins importantes :
- le domaine HBH est tronqué après la première hélice, qui se termine par 6 acides aminés modifiés par rapport à
l’allèle sauvage pour W211.
- Il y a trois acides aminés différents entre la protéine codée par W211R3 et celle codée par l’allèle sauvage au
niveau de la fin de la première hélice.
- il y a deux (un) acides aminés différents entre la protéine codée par W211R2 (par W211R1) et celle codée par
l’allèle sauvage, toujours dans la région terminale de la première hélice.
Le Western-blot (panneau 1) révèle la quantité de protéines AN1 produite par chaque type de mutant étudié, en
comparaison avec le plant sauvage. L’absence de tâche pour W211 pourrait s’expliquer par une différence de
migration importante de la protéine dont la séquence est raccourcie. On n’observe pas de différence significative
entre les différents mutants : tous produisent bien cette protéine (la tâche est légèrement moins foncée que pour le
plant sauvage, mais en l’absence de témoin de charge, on ne peut être sûr que la différence soit significative).
Le Northern-blot (panneau 2) met en évidence la quantité d’ARNm résultant de la transcription du gène dfr pour le
plant sauvage et les différents mutants. Les ARNm de gapdh nous servent de témoin de charge : ils permettent de
vérifier que les conditions expérimentales mises en œuvre fournissent des résultats exploitables, c’est bien le cas ici.
On constate qu’il n’y a aucun ARNm produit pour le mutant W211, or c’est celui pour lequel la séquence de la
protéine AN1 était la plus modifiée.
Pour les autres mutants, il y a moins d’ARNm produits que chez le plant sauvage, et on observe que la quantité
d’ARNm produit, proportionnelle à l’intensité de la tâche, semble corrélée au nombre de différences entre la
séquence de la protéine AN1 du mutant considéré par rapport au sauvage : plus les différences sont nombreuses,
moins il y a d’ARNm de dfr.
Or on sait que la protéine AN1 peut se lier à l’ADN et que les différences repérées portent justement sur les
domaines mis en jeu dans cette liaison à l’ADN.
On peut donc proposer que la protéine AN1 joue le rôle de facteur de transcription et, en se liant à l’ADN, contrôle
l’expression du gène dfr.
Le gène dfr est impliqué dans la voie de biosynthèse des anthocyanes (comme le montre le document annexe), dès
lors, cela explique les phénotypes des mutants étudiés : de O à S, les corolles sont de plus en plus pigmentées, ce
qui correspond à une expression plus importante de dfr, contrôlée par la protéine AN1.
 6
6
 7
7
1
/
7
100%