Projet programmation PYTHON 2

Projet programmation
PYTHON 2
Objectif : Lecture d’un fichier de la base de données PDB et
analyses diverses.
Trinôme : Année :
KUNEGEL Marion GB4
MERLET Benjamin 2011/2012
MERLET BILLON Maryvonne

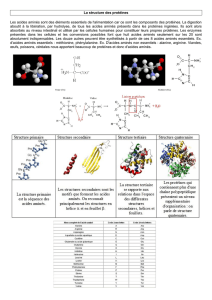
I. Stratégie et remarques générales :
Pour réaliser ce projet nous avons été amenés à définir 18 fonctions indépendantes.
La récupération du fichier sur internet a nécessité l’importation d’un module spécifique.
Dans le programme principal, les choix sont proposés à l’utilisateur par des chaines de
caractères affichées à l’écran par l’outil print. Le dialogue avec l’utilisateur se fait grâce aux
structures de contrôle « input » (variable dont la valeur est définie par l’utilisateur), if et
elif/else. Le retour au menu principal est réalisé par une boucle while, de manière à ce que
tant que le choix de l’utilisateur est différent de 6 (choix pour quitter le programme), le
programme retourne au menu principal en attente d’une nouvelle requête de l’utilisateur.
La même stratégie a été utilisée au niveau des sous-menus. Pour créer ce programme, de
nombreuses variables ont été utilisées, et distinguer les variables locales des variables
globales a été une des principales difficultés. Nous avons également passé beaucoup de
temps sur la gestion des exceptions afin de construire le programme le plus robuste
possible. Ces exceptions ont été gérées à l’aide des outils try et except ou par des boucles
while. Voici la liste des erreurs possibles qui ont été traitées afin de ne pas entrainer le
plantage du programme :
L’utilisateur n’est pas connecté à internet et la fonction du module ne peut pas
récupérer le fichier PDB (en format bytes).
L’utilisateur est connecté à internet mais entre le nom d’un fichier PDB qui n’existe
pas (l’url fonctionne mais elle ouvre une page internet d’erreur).
L’utilisateur doit obligatoirement répondre par « oui » ou par « non » quand on lui
propose un nom de fichier.
Dans le cas où l’utilisateur souhaite lui-même nommer le fichier, le nom choisit doit
être valide.
Pour tout enregistrement le programme vérifie que le fichier n’existe pas déjà dans le
répertoire. Si le fichier existe, il est demandé à l’utilisateur s’il souhaite l’écraser.
L’utilisateur doit obligatoirement entrer un numéro de choix existant dans le menu.
Pour l’analyse personnalisée, les choix indiqués doivent appartenir à la liste donnée à
l’utilisateur.
Pour le calcul de la distance entre 2 acides aminés, les positions et les chaines
protéiques indiquées par l’utilisateur doivent être présentes dans la séquence.
II. Présentation des différentes fonctions du programme :
A. Récupération visualisation et enregistrement du fichier :
load_PDB : Cette fonction prend en entrée le code PDB qui est demandé à l’utilisateur par
un input. Après avoir créé une url à partir de ce code, la fonction urlopen du module

urllib.request permet d’ouvrir le fichier mais en format bytes. Ligne par ligne les bytes sont
décodés par la fonction bytes.decode pour obtenir un fichier texte de type string. Chaque
ligne est incorporée dans une chaine de caractère stockée dans la variable fichier et
incorporée dans une liste ou chaque élément est lui-même une liste constituée des chaines
de caractères de la ligne. Ainsi la fonction retourne à la fois une chaine de caractères
destinée à l’utilisateur (fichier) et une liste de listes (data) qui sera utilisée pour la réalisation
des autres fonctions. L’échec du téléchargement est géré par la variable erreur.
Testfile : Cette fonction permet de savoir si le nom du fichier existe déjà. Elle utilise la
fonction isfile du module os.path. et prend en entrée un nom de fichier (string). La fonction
isfile renvoie True si le fichier existe déjà et False s’il est inconnu. Dans le cas ou le nom
existe déjà (True), on demande à l’utilisateur si celui-ci veut écraser le fichier. La fonction
testfile renvoie False lorsque la question de l’écrasement du fichier à été gérée avec
l’utilisateur.
Save : Cette fonction permet d’enregistrer un fichier. Elle prend en entrée une chaine de
caractère. Dans le programme principal on propose à l’utilisateur un nom de fichier. Une
boucle while impose à l’utilisateur de répondre par « oui » ou par « non ». S’il répond
« non », l’utilisateur peut alors proposer un nom de fichier. Afin de s’assurer de la validité du
nom donné par l’utilisateur, on utilise une exception try et une boucle while.
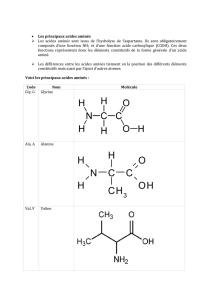
B. Récupération de la séquence dans les différents formats :
Dans cette partie nous avons choisi de prendre en compte le cas des fiches PDB avec
plusieurs modèles : nous avons choisi de n’afficher les données que pour le premier modèle.
Cette stratégie est aussi appliquée aux fiches présentant plusieurs sous-unités. Bien que ces
dernières soient utilisées pour le calcul de distance des acides aminés, dans cette partie ainsi
que pour l’analyse, nous ne tiendrons compte que de la première chaîne.
SeqAA: Cette fonction permet d’extraire la liste des acides aminés au carbone alpha à partir
de la liste de listes data. Ceci nous permet de réduire le temps d’exécution des fonctions
suivantes. Grâce à une boucle while nous créons une liste temporaire temp où nous
enregistrons toutes les lignes du fichier PDB commençant par « ATOM » et contenant « CA »
grâce à la fonction .append(). Puis une autre boucle while nous permet de créer la liste seq
contenant les acides aminés. Afin de traiter le problème des fichiers PDB contenant plusieurs
modèles d’une même protéine, nous avons choisi comme condition d’extraction la
numérotation des acides aminés : ces derniers ne sont ajoutés à la liste que si le numéro qui
leur est associé est supérieur à celui de l’acide aminé précédent. C’est pour cette raison
qu’avant la boucle while le troisième élément de la première sous-liste temp est ajouté à la
liste seq. Ensuite à l’intérieur de la boucle le troisième élément de chaque sous-liste de temp
qui rempli la condition d’exécution est ajouté à la fin de la liste seq.

AA3toAA1 : Cette fonction prend en entrée la liste crée par SeqAA (nommée seq ). Un
dictionnaire (dicoAA) faisant la correspondance entre le code 1 lettre et le code 3 lettres des
acides aminé est préalablement créé. La fonction parcourt la liste et pour chaque élément si
celui-ci est présent parmi les champs du dictionnaire (code 3 lettres) elle récupère sa valeur
(code 1 lettre) et l’ajoute dans une nouvelle chaine de caractère. Cette fonction renvoie
donc une chaine de caractère de la séquence d’acides aminés au format 1 lettre nommée
res.
Fasta : Elle prend en entrée le nom du fichier, la première ligne du fichier PDB qui est le
premier élément de la liste data (le caractère « header » a été exclu puisqu’il n’a pas
d’utilité) et la chaine de caractère res correspondant à la séquence au format 1 lettre. La
fonction écrit alors dans un fichier une première ligne caractéristique du format fasta en
concaténant 3 chaines de caractères : « > » + la première sous-liste transformée en chaines
de caractères mais sans le caractère « header » + « \n » pour un retour à la ligne. La fonction
parcourt ensuite la chaine de caractère res et l’écrit à la suite de la ligne précédente dans le
même fichier. Dès que la longueur de la ligne atteint 80 caractères, on ajoute un retour à la
ligne sauf pour la dernière ligne (conditions du format fasta).
C. Analyse générale de la séquence
Récupération des Titres d’intérêts, la liste : Nous avons créé à la main une liste (listetitre)
comportant les titres des parties descriptives du fichier PDB comme TITLE, EXPDTA, ...
indépendamment du fichier PDB en cours d’analyse.
Verif_titre : Cette fonction vérifie si les titres présents dans listetitre le sont aussi dans le
fichier PDB et crée une liste de titres (titreverif), les titres n’y sont présents qu’une fois grâce
à une boucle if/elif qui vérifie à la fois si le titre du fichier PDB est déjà présent dans titreverif
et s’il est présent dans listetitre.
Fonction recuptitre : Cette fonction, qui n’est pas utilisée dans le programme final, devait
créer la liste de titre à partir du fichier PDB en récupérant les titres d’intérêts. Le programme
n’aurait alors pas eu besoin de vérifier si les titres étaient présents dans le fichier PDB. Nous
n’avons pas pu l’utiliser à cause d’un trop grand nombre d’exceptions à gérer.
Titre : Cette fonction prend deux arguments en entrée : la liste de listes data en cours
d’analyse et le mot-clé correspondant à l’information que l’utilisateur souhaite afficher
keyword. Cette fonction retourne toutes les lignes du fichier PDB qui commencent par ce
keyword, exception faite si le keyword est EXPDTA : si la réponse est X-RAY DIFFRACTION,
alors la fonction va aussi chercher la résolution dans la partie REMARK du fichier PDB.

Ces deux fonctions nous permettent ainsi d’afficher les informations générales que veux
l’utilisateur.
Calcul du poids moléculaire, le dictionnaire : Nous avons créé à la main un dictionnaire
contenant comme clé le code trois lettres de tous les acides aminés, et comme valeur le
poids moléculaire (en g/mol) de chaque acide aminé.
MW : Cette fonction nous permet de calculer le poids moléculaire de la protéine et prend
comme argument la liste créée par la fonction seqAA. Par une boucle while la fonction
parcourt la liste et ajoute à la variable mw la valeur du dictionnaire dico qui correspond à la
clé. Comme nous avons adapté la notation des clés du dictionnaire au code 3 lettres des
acides aminés, nous pouvons demander à la fonction d’agir comme si seqaa[i] était
réellement une clé du dictionnaire.
Frequence : Cette fonction permet de calculer la fréquence des acides aminés présents dans
la séquence protéique en prenant comme argument la liste crée avec la fonction seqAA.
Nous créons un dictionnaire dicoaa qui nous servira de variable de résultat. Par une boucle
while la liste seqaa est parcourue : Si la valeur correspondant à l’indice i n’est pas dans le
dictionnaire dicoaa, ce dernier est mis à jour en ajoutant en clé la valeur de l’indice i et en
valeur 1. Une variable aatot nous permet de comptabiliser le nombre d’acides aminés.
Puisque nous avons travaillé avec un dictionnaire, nous avons choisi une liste comme
variable de résultat : grâce à une boucle for nous recalculons en valeur du dictionnaire la
fréquence correspondant à la clé (qui est un acide aminé). Nous mettons ensuite à jour une
liste (liste), contenant comme premier élément le caractère spécial « \n », en ajoutant
successivement par .append() la clé du dictionnaire, la valeur ainsi que le caractère spécial
« \n » qui correspond à un retour chariot (ceci pour une commodité d’affichage par la suite).
D. Analyse de la distance entre 2 acides aminés :
Pour cette partie (choix 5 dans le menu principal), nous avons décidé de différencier les
protéines comportant plusieurs chaines (ex : 2ght) à celles qui n’en ont qu’une (ex : 1crn).
Notre programme peut ainsi calculer la distance entre deux acides aminés de deux chaines
différentes (si leurs coordonnées sont dans le fichier PDB).
Pour effectuer les calculs de distance, il faut tout d’abord importer le module math pour
utiliser la fonction racine carré math.sqrt().
Liste_Ca : Cette fonction récupère de la liste de listes data toutes les lignes du fichier PDB
commençant par « ATOM » et comportant « CA » comme 3ième élément. La variable
retournée (listCa ) est donc une liste de listes comprenant toutes les informations de
l’ensemble des carbones alpha de la protéine.
 6
6
1
/
6
100%