Projet USI11- Python 2 – GB4 2013

Projet USI11
-
Python 2
–
GB4
2013
Vous devrez rendre une archive (.gz, .zip, …) contenant le/les fichiers sources
python (.py), les documents associés en n’oubliant pas de mettre des
commentaires sur les principales lignes de votre programme.
Un petit document (6 pages maximum) expliquant votre stratégie, vos remarques
sur ce projet ou sur les résultats, etc… est fortement souhaité. Travail en
trinôme.
Cette archive devra être rendue pour le vendredi 29 novembre en n’oubliant pas
d’indiquer les noms des auteurs (nom de l’archive et dans les fichiers sources). Un oral
de 5 minutes par groupe sera organisé le jeudi 5 décembre.
Objectif du programme : Lecture d’un fichier de la base de données PDB et analyses
diverses. Le format du fichier PDB est décrit à l’adresse suivante :
http://www.wwpdb.org/documentation/format33/v3.3.html
Description succincte (Vous êtes libre de rajouter des fonctionnalités ou des
analyses qui vous sembleraient pertinentes) :
Votre programme devra:
Récupérer sur Internet le contenu d’un fichier PDB après que l’utilisateur ait
saisi le code PDB correspondant.
Visualiser ou enregistrer le fichier PDB.
Récupérer la séquence protéique dans différent format et proposer de la
visualiser ou de l’enregistrer dans un fichier.
Faire différentes analyses sur le fichier et proposer soit d’afficher les
résultats à l’écran soit de les enregistrer dans un fichier.
Faire un calcul de distance entre 2 résidus du fichier (sur les Carbones Alpha)
Calculer le profil d’hydrophobicité pour chaque fenêtre glissante de 9 acides
aminés et l’enregistrer dans un fichier.
Ces différentes fonctionnalités devront être proposées sous forme d’un menu avec un
choix demandé à l’utilisateur.
Récupération de la séquence protéique dans différents formats :
On va proposer deux formats : le format fasta (séquence sur 80 colonnes avec une
ligne de commentaire avec le code PDB et la méthode expérimentale) et le format aa3
avec la séquence en code 3L espacée par un tiret. L’utilisateur pourra proposer ou non
un nom de fichier de sortie pour ces différents formats.
Analyse diverses :
On vous demande de récupérer dans le fichier PDB le titre de la structure (champs
TITLE) ainsi que la méthode expérimentale utilisée (S’il s’agit de X-RAY, il faudra
récupérer la résolution de la structure).
On vous demande de calculer le poids moléculaire de la structure, les fréquences des
différents acides aminés ainsi que le pourcentage de résidus hydrophobes, le

Projet USI11
-
Python 2
–
GB4
2013
pourcentage de résidus chargés et la charge net (sommes des charges)
Ces analyses pourront être affichées ou écrites dans un fichier dont l’utilisateur
pourra avoir la maitrise du nom. Par exemple :
Profil d’hydrophobicité de la protéine :
L’hydrophobicité moyenne pour chaque fenêtre glissante de 9 acides aminés est
calculé de la manière suivante :
Avec N le nombre de résidu par fenêtre et H
n
l’hydrophobicité du résidu.
On utilisera pour cela l’échelle d’hydrophobicité de Fauchere et Pliska (Eur. J. Med.
Chem. 18:369-375(1983))
Ala: 0.310 Arg: -1.010 Asn: -0.600 Asp: -0.770 Cys: 1.540 Gln: -0.220 Glu: -0.640 Gly:
0.000 His: 0.130 Ile: 1.800 Leu: 1.700 Lys: -0.990 Met: 1.230 Phe: 1.790 Pro: 0.720
Ser: -0.040 Thr: 0.260 Trp: 2.250 Tyr: 0.960 Val: 1.220
Ces valeurs devront être enregistrées dans un fichier dont le format sera compatible
avec l’utilisation d’un tableur (Excel, OpenOffice Calc, …) pour tracer le profil.
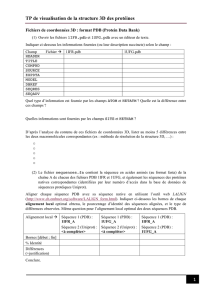
Calcul de distance :
Votre programme devra demander à l’utilisateur deux numéros de résidus (qui devront
être compatibles avec le fichier PDB). Vous calculerez ensuite la distance euclidienne
entre les 2 carbones Alpha de ces résidus grâce aux coordonnées de ces atomes
récupérés dans le fichier PDB, par exemple :
Votre programme devra être robuste vis à vis de l'utilisateur, des choix
demandés, des résultats obtenus et devra gérer au mieux les erreurs possibles
sans entrainer de plantage du programme.
1
/
2
100%