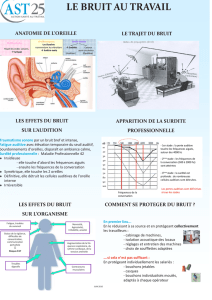
La sélection naturelle: son étude par la génétique des populations

La sélection naturelle: son étude par la génétique
des populations et son importance
comme « force évolutive »
Patrice PICCINO
Equipe Evolution et Génétique des Populations Marines
Station Biologique de Roscoff
DEA d’Océanographie Paris 6
Jeudi 6 décembre 2001

Plan du cours
1-Introduction
1-1-Apparition du concept de sélection naturelle
1-2-Lien entre Biologie, Biologie évolutive, Génétique des populations et Sélection…
1-3-Notion de valeur sélective
2-Les modèles de bases en Génétique des Populations
2-1-Hypothèses implicites
2-2-Calcul des fréquences alléliques avant et après sélection
2-3-Sélections directionnelle, balancée et disruptive et paramétrisation de la valeur
sélective
2-4-Sélection directionnelle (« General dominance »)
- « purifying selection »
- « progressive selection »
2-5-Sélection balancée par avantage de l’hétérozygote
2-6-Sélection disruptive par avantage des homozygotes
P. Piccino

3-Modèles plus complexes
3-1-Influence des paramètres environnementaux (« Ecological genetics »)
3-2-Variation spatiale de ces paramètres: modèle de Levene (1953)
3-3-Autres modèles de sélection
3-4-Test permettant de détecter de la sélection
4-Limites de l’influence de la sélection naturelle
4-1-Niveaux de sélection
4-2-Limites de la sélection
4-3-Evolution moléculaire et sélection
5-Exemples d’études de la sélection naturelle en milieu marin
5-1-Micro-environnements, sélection et calcul de valeur sélective
5-2-Survie différentielle, environnement hétérogène et modèle de Levene
5-3-Comparaison entre marqueurs « neutres » et marqueurs allozymiques: sélection
balancée?
5-4-Evolution moléculaire et sélection positive
5-5-Marqueurs allozymiques, différenciation génétique en fonction de l’habitat
(température,…) et propriétés biochimiques différentielles d’allozymes en fonction de la
température P. Piccino

Bibliographie
Livres
•Philip W. Hedrick. Genetics of populations
. Second Edition.
Jones and Bartlett Publishers. 1999
•Francine Brondex. Évolution Synthèse des faits et théories. Dunod. 1999
Exemples
•Strong natural selection causes microscale allozyme variation in a marine snail
Johannesson et al, Proc. Natl. Acad. Sci. USA, 92, 1995, 2602-2606
•Adaptative maintenance of genetic polymorphism in an interdital Barnacle: Habitat-
and Life-stage-specific survivorship of MPI genotypes
Paul S. Schmidt and David M. Rand, Evolution, 55(7), 2001, 1336-1384
•Balancing Selection at Allozyme loci in Oysters: Implications from Nuclear RFLPs
Stephen A. Karl and John C. Avise. Science, 256, 1992, 100-102
•Antartic fish hemoglobins: Evidence for adaptative evolution at subzero temperature
Barcelloni, L., Marcato, S. and Patarnello, T., Proc. Natl. Acad. Sci. USA, 95, 1998,
8670-8675
P. Piccino

1-Introduction
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
1
/
45
100%