TP3 Évolution et fonction des protéines Les protéines sont des

TP3
Évolution et fonction des protéines
Les protéines sont des éléments fonctionnels de la cellule qui jouent des rôles spécifiques. Leur
fonction dépend entre autres de la nature et de la position des acides aminés qui les composent
puisque ceux-ci déterminent la structure tridimensionnelle de ces protéines. Nous pouvons
supposer qu’il y a une pression évolutive pour la conservation des protéines qui ont des rôles
similaires et/ou pour une adaptation de leur fonction selon les besoins des cellules-organismes
dans lesquelles elles agissent. Dans ce TP, nous allons comparer des familles de protéines à
travers les espèces.
Dans ce TP, à la manière des expériences de laboratoire, vous devrez suivre une approche
scientifique. Vous devrez
1) Faire l’expérience demandée
2) Décrire les résultats obtenus
3) Formuler une hypothèse scientifique qui permet d’expliquer les observations. Tenir
compte que le TP3 porte sur la structure des protéines et de leur conservation/adaptation
lors de l’évolution.
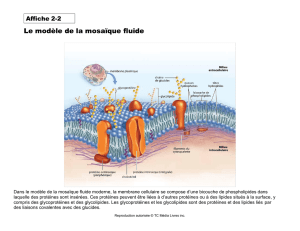
Partie 1 : Histone H4.
Les histones sont des protéines qui, chez les eucaryotes, permettent de structurer et
d’empaqueter l’ADN dans des unités structurales appelées « nucléosomes ». Elles lient donc
d’ADN et sont un important constituant de la chromatine.
Pour étudier l’évolution-adaptation de l’histone H4 dans différentes espèces éloignées, nous
utiliserons l’algorithme du site http://www.uniprot.org.
Dans "UniProtKB", entrez le terme "histone H4"
Dans la page de Résultats, cocher l'entrée P62801, P62805, P62806, P09322,Q41811 et P84040
(espèces Gallus chicken, Homo sapiens, Mus musculus, Schizosaccharomyces pombe, Zea mays
(mais), et Drosophila melanogaster, respectivement)
1- Alignez les séquences des 6 protéines. En haut de la page, cliquez sur "Align". Uniprot va
effectuer un alignement de séquences.
Question 1.1a. Qu’observez-vous?
Question 1.1b.Comment expliquez-vous le résultat du point de vue de la fonction de ces
protéines (formulez une hypothèse scientifique)?
2- Comparez l’identité des séquences de protéine pour l’histone H4 de l’humain (homo) et du
maïs (zea). Comparez maintenant les séquences des ARNm codant pour l’histone H4 de
l’humain (homo.fasta) et du maïs (zea.fasta). Choisissez « Somewhat similar sequences » dans
le panneau « Program Selection ».

Question 1.2a. Quel est le pourcentage d’identité au niveau protéique?
Question 1.2b. Quel est le pourcentage d’identité au niveau de l’ARNm?
Question 1.2c. Comment expliquez-vous le résultat du point de vue moléculaire (formulez une
hypothèse scientifique)?
3- Analysez les propriétés de la protéine. En cochant aux endroits appropriés dans la section
"Annotation" et/ou "Amino acids properties" située à gauche dans la figure d'alignement,
répondez aux questions suivantes.
Question 1.3a. Quelle est la séquence en acides aminés du motif de liaison à l'ADN prédite par
Uniprot ? Quelle propriété possèdent ces résidus? Est-ce consistent avec ce qui a été vu en
cours concernant l’association entre l’histone et l’ADN ?
Question 1.3b- Quelle est la charge nette de la protéine ? Formulez une hypothèse scientifique.
Partie 2 : Kinase PKR
La kinase PKR est une kinase qui est activée par sa liaison à l’ARN double-brin.
Nous allons répéter le même type d’expérience sur le site http://www.uniprot.org/
En bas à gauche de la page, sélectionnez « Align ».
Dans le carré « Protein sequences (FASTA) or UniProt identifiers », inscrivez P19525 et
B7TCB0 (un numéro par ligne). Ce sont les numéros d’accession des protéines PKR chez
l’humain et le poisson, respectivement.
1- Cliquez sur "RUN Align". Uniprot va effectuer un alignement de séquences.
Question 2.1a. Qu’observez-vous?
Question 2.1b.Comment expliquez-vous le résultat du point de vue de la fonction de ces
protéines (formulez une hypothèse scientifique)?
2- Pour analyser plus en détail l’évolution de cette protéine, sélectionnez « domain » dans la
section Annotation à gauche.
Question 2.2a. Qu’observez-vous?
Question 2.2b- A quels domaines fonctionnels correspondent les régions prédites par le
programme Uniprot (en utilisant la séquence de la protéine humaine)?
Question 2.2c.Y a-t-il concordance entre la position des domaines fonctionnels et les résultats
de la comparaison de séquence?
Question 2.2d. Comment expliquez-vous le résultat du point de vue de la fonction de ces
protéines (formulez une hypothèse scientifique)?
Question 2.3- Donnez une fonction de la protéine PKR chez l’humain.

Partie 3 : Isomérases
Accédez à nouveau au site www.uniprot.org
Sous "ALIGN", entrez les numéros d’accession Q6GIL6, P24299 et P0A2W8 (un numéro par
ligne). Ces numéros correspondent aux protéines "Triosephosphate isomerase Staphylococcus
aureus strain MRSA252", " Xylose isomerase Streptomyces albus G" et "Alanine racemase
Streptococcus pneumoniae", respectivement.
1- Cliquez sur "Run Align".
Question 3.1a. Qu’observez-vous?
Question 3.2b.Comment expliquez-vous le résultat du point de vue de la fonction de ces
protéines (formulez une hypothèse scientifique)?
2- Nous allons maintenant nous pencher sur la structure de ces trois protéines.
Démarrer l'application PyMOL.
Observez la structure des protéines 3M9Y_A.pdb, 3S46_A.pdb et 6XIA_A.pdb. Pour vous
faciliter la tâche, vous pouvez utiliser la représentation « cartoon » et colorer les hélices et
feuillets.
Question 3.2a. Qu’observez-vous?
Question 3.2b.Comment expliquez-vous le résultat du point de vue de la fonction de ces
protéines (formulez une hypothèse scientifique)?
Partie 4. Conclusions
Question 4.
En analysant l’ensemble des résultats obtenus dans les 3 sections, formulez une conclusion
quant au lien entre la séquence des protéines, leur fonction et leur conservation durant
l’évolution.

Homo.fasta
1 ggagtcagaa tgggtttaca tttaattctt aacccagttc ctcaaagcct gtagctctgt
61 taagaaaata aaggccattg gtcaagcctg cttggtccca ccctcatctc cccaccctcc
121 cccaatcgct gctcccgcca tttcctgggg cttggaggag gggttaaagg agcggactgt
181 aggcgtcaca tttcccgcct gcgcgctttt cagtctcagt gtccgctgga ggtgggggca
241 ggggtaacgt agatatataa agatcggttt cctattctct cacttgctct tggttcactt
301 cttgggaagt catgtctgga cgtggtaagg gcgggaaggg tttgggtaag gggggtgcca
361 agcgccaccg caaggtgttg cgtgacaaca tccagggcat caccaagccg gccatccggc
421 gtctggcccg gcgtggcggt gtgaagcgga tctctggtct gatctacgag gagactcgcg
481 gggtgctcaa ggtgtttttg gagaacgtga tccgtgacgc tgtcacctat acggagcacg
541 ccaagcgcaa gacagtcact gccatggacg tggtctacgc gcttaagcgc cagggacgca
601 ccctttatgg ctttggcggt taaggttgct gatttctcca cagcttgcat ttctgaacca
661 aaggcccttt tcagggccgc ccaactaaac aaaagaagag ctgtatccat taagtcaaga
721 agctcaatgt gtaattaaga tgaatgatac tgagctgaca tcctaaaaag gaaagattag
781 gggaactcca agtttgccct ccactcacta catatgggta ggggagcaac gatattccaa
841 ctctgaagaa agagtggaaa aaaagtagtg ttaaaaattt gtattagttt ccaagggaca
901 aagaagcgct gcccaatcaa tgagggccat
Zea.fasta
1 aacaacaaca acaacccgca caacggcacg attctcctcc tccgtccgcg tagctctcgg
61 tttcggcgat gtcggggcgc ggcaagggcg gcaaggggct gggcaagggc ggcgccaagc
121 gccaccgcaa ggtgctgcgc gacaacatcc agggcatcac gaagccggcg atccgtaggc
181 tggccaggag gggcggcgtc aagcgcattt ctgggctcat ctacgaggag acccgcggcg
241 tgctcaagat cttcctcgag aacgtcatcc gcgacgccgt cacctacacc gagcacgctc
301 gccgcaagac cgtcaccgcc atggatgtcg tctacgcgct caagcgccag ggccgcacgc
361 tctacggctt cggcggctag gccgcctccc cttctgatgc tttgatatga cggccgtggt
421 cggttgtgtg tctagttggc cggtctactg tagtttgttc tgtatcttcg tttcgaggtg
481 ttccagtttc tagcccattt ggtgtaatat tttcacagct attatcaatg gaaagccctc
541 ggttctatcc tcattgttct ggatcaagat aatccgtttg atatacaaaa aaaaaaaaaa
601 aaaaaa
1
/
4
100%