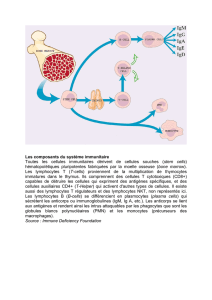
Thèse électronique Habiba ELATMANI

1
Université Victor Segalen Bordeaux 2
Année 2009
Thèse n°_4830
THÈSE
pour le
DOCTORAT DE L’UNIVERSITÉ BORDEAUX 2
Mention : Biologie-santé
Option : Biologie cellulaire et physiopathologie
Présentée et soutenue publiquement
Le 17 Décembre 2009
Par Habiba ELATMANI
Née le 18 Septembre 1981 à Vernon (27)
Caractérisation du rôle d’Unr, une protéine de liaison à l’ARN,
dans les cellules souches embryonnaires murines
Membres du Jury
M CULLIN Christophe ........................................................................ Président
Professeur de l’Université Bordeaux II
Mme MORELLO Dominique .............................................................. Rapporteur
Directeur de recherche CNRS, Université Toulouse III
M AVNER Philippe ............................................................................... Rapporteur
Directeur de recherche CNRS, Institut Pasteur
M LE MENUET Damien ...................................................................... Examinateur
Chargé de recherche INSERM, Université Paris XI
Mme JACQUEMIN-SABLON Hélène ................................................ Directeur de thèse
Chargé de recherche INSERM, Université Bordeaux II

2
Abréviations
APAF-1 : Apopstic Protease-Activating Factor 1
ARE : AU-rich elements
ARE-BPs : ARE Binding Proteins
ARN : Acide RiboNucléique
ARNm (s) : ARN messager (s)
ARNt : ARN de transfert
CSD : Cold Shock Domain
CSPs : Cold Shock Proteins
DCC : Dosage Compensation Complex
Dcp1/2/S : Decapping protein 1/2/S
E : jour Embryonnaire
eEF1A : eukaryotic Elongation Factor A
eIF2 : eukaryotic Inition Factor 2
eIFs : eukaryotic Initiation Factors
Epi : Epiblaste
Epr : Endoderm primitif
ES : Embryonic Stem
IRES : Internal Ribosome Entry Site
ITAFs : IRES Trans Acting Factors
JAK : Janus Kinase
Kb : Kilobase
KDa : KiloDalton
LIF: Leukemia Inhibitory Factor
MCI : Masse Cellulaire Interne
miARN : microARN
mCRD : major Coding Region of Determinant of instability
miRNP (s) : miARN RiboNucleoProteic complex (s)
Msl-2 : Male specific lethal-2
PABP : PolyA Binding Protein
PAIBP1 : PABP Interacting Protein 1
PABPII : PolyA Binding Protein II

3
PAP : PolyA Polymérase
PARN : PolyA-specific RiboNuclease
pb: paire de base
pré-ARNm (s) : ARN pré-messager (s)
Pré-miARN (s) : précurseur miARN (s)
Pri-miARN (s) : miARN (s) primaire (s)
PTB: Polypyrimidine Tract Binding protein
PTH: Parathyroid Hormon
RBP: RNA Binding Protein
RISC: RNA Induced Silencing Complex
RRM: RNA Recognition Motif
SMG: Smaug
Sxl: Sex lethal
TE: Trophectoderme
unr: Upstream of N-ras (gène)
3’NT: Region 3’ Non Traduite
5’NT: Region 5’ Non Traduite

4
Sommaire
Introduction………………………………………………………………………………….6
I) Unr, une protéine de liaison à l’ARN impliquée dans la régulation post-transcriptionnelle
de l’expression des gènes. ...................................................................................................... 7
1) Le gène unr code une protéine de liaison à l’ARN conservée au cours de l’évolution. 7
A) Découverte du gène unr: upstream of N-ras. ............................................................ 7
a) Historique. .............................................................................................................. 7
b) Le gène unr code une protéine à cinq domaines « cold- shock ». ......................... 8
B) Les protéines à domaine cold shock chez les procaryotes et les eucaryotes. ............ 8
a) Structure du domaine cold shock. .......................................................................... 8
b) Les protéines à domaine cold- shock chez les procaryotes. ................................... 8
c) Les protéines à domaine cold-shock chez les eucaryotes. ..................................... 8
C) Unr, une protéine de liaison à l’ARN ubiquitaire et conservée au cours de
l’évolution. ................................................................................................................... 10
a) Rôle Fonctionnel et localisation subcellulaire de la protéine Unr. ...................... 10
b) Conservation d’unr au cours de l’évolution. ........................................................ 10
c) L’expression d’unr est ubiquitaire. ...................................................................... 10
2)
Unr participe à la régulation post-transcriptionnelle de l’expression des gènes. ..... 11
A)
Introduction aux régulations post-transcriptionnelles de l’expression des gènes
chez les eucaryotes. ...................................................................................................... 11
a) Vue générale des différents niveaux de régulation de l’expression des gènes. ... 11
b) Les régulations post transcriptionnelles de l’expression des gènes ..................... 12
B)
Le devenir des ARNms : traduction et dégradation. .......................................... 13
a) Modifications post-transcriptionnelles du pré- ARNm : ajout de la coiffe 5’ et de
la queue polyA. ........................................................................................................ 13
b) La traduction des ARNms. ................................................................................... 13
b1) L’initiation de la traduction coiffe dépendante. ................................................. 14
b2) La traduction dépendante d’IRES. ..................................................................... 15
c) Dégradation des ARNms. ..................................................................................... 15
c1) Enlèvement de la queue polyA. ......................................................................... 15
c2) Enlèvement de la coiffe 5’. ................................................................................ 16
C) Modes de régulation de la traduction et de la stabilité des ARNms. ...................... 17
a) Régulations post-transcriptionnelles par les microARNs. ................................... 17
b) Régulation post-transcriptionnelle par les éléments AU-rich .............................. 20
D) UNR régule la stabilité et la traduction d’ARNms. ................................................ 21
a) Régulation de la stabilité des ARNms. ................................................................ 21
b) Régulation traduction dépendante d’IRES. .......................................................... 23

5
c) Régulation de la traduction coiffe dépendante. .................................................... 23
E) Régulations post-transcriptionnelles et développement : Unr essentielle pour le
développement de la souris .......................................................................................... 25
a) Détermination des axes de l’embryon de drosophile par une succession
d’inhibition de traduction. ........................................................................................ 25
b) La protéine Unr est essentielle pour le développement embryonnaire de la
drosophile et de la souris. ......................................................................................... 26
b1) Unr et dosage génique compensatoire du chromosome X chez la drosophile. .. 26
b2) Le gène unr est essentiel pour le développement de la souris. .......................... 27
II) Développement embryonnaire précoce de la souris et cellules souches embryonnaires
murines. ................................................................................................................................ 30
1) Le développement embryonnaire précoce chez la souris. ........................................... 30
A) De la fécondation à la formation du blastocyste ..................................................... 30
a) Clivage de l’embryon, compaction et formation du blastocyste. ......................... 30
b) Spécification des premiers lignages cellulaires. .................................................. 32
b1) Ségrégation du trophectoderme et de la masse cellulaire interne ...................... 33
b2) Ségrégation de l’endoderme primitif et de l’épiblaste. ...................................... 34
B) De l’implantation à la gastrulation. ......................................................................... 36
a) Implantation et formation de l’œuf cylindre ........................................................ 36
b) La gastrulation: différenciation de l’épiblaste, formation des feuillets
primordiaux .............................................................................................................. 37
2)
Les cellules souches embryonnaires murines. .......................................................... 39
A) Description des cellules souches embryonnaires. ................................................... 39
a) Propriétés des cellules souches embryonnaires. ................................................... 39
b) Culture des cellules ES murines. ......................................................................... 41
B) Etablissement, maintien et régulation de la pluripotence. ....................................... 41
a) Identification de gènes clés de pluripotence: oct4, sox2 et nanog. ...................... 41
a1) Oct4 .................................................................................................................... 41
a2) Sox2 .................................................................................................................... 42
a3) Nanog. ................................................................................................................ 42
b) Circuit transcriptionnel de régulation de la pluripotence. .................................... 45
c) MicroARN et cellules ES ..................................................................................... 46
Objectifs……………………………………………………………………………………...48
Résultats……………………………………………………………………………………...49
Discussion…………………………………………………………………………………….76
Perspectives…………………………………………………………………………………..80
Bibliographie………………………………………………………………………………...82
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
 53
53
 54
54
 55
55
 56
56
 57
57
 58
58
 59
59
 60
60
 61
61
 62
62
 63
63
 64
64
 65
65
 66
66
 67
67
 68
68
 69
69
 70
70
 71
71
 72
72
 73
73
 74
74
 75
75
 76
76
 77
77
 78
78
 79
79
 80
80
 81
81
 82
82
 83
83
 84
84
 85
85
 86
86
 87
87
 88
88
 89
89
 90
90
1
/
90
100%



![Poster CIMNA journée CHOISIR [PPT - 8 Mo ]](http://s1.studylibfr.com/store/data/003496163_1-211ccc570e9e2c72f5d6b6c5d46b9530-300x300.png)