Le compartiment cytosolique

1
Le cytoplasme est divisé en 2 compartiments
Les organites
Le cytosol
Le noyau a une enveloppe qui fait partie du système endomembranaire et qui
communique avec le cytosol par l’intermédiaire des pores nucléaires (cf cours sur
le traffic nucléocytoplasmique).
Le compartiment cytosolique
-1-Généralités
De viscosité et de richesse variable d’un point à l’autre de la ¢
Riche en eau, ions (K+, peu de Na+, peu de chlore)
-Formes de stockage : (visibles en microscopie électroniques).
- Inclusions lipidiques,
- Sucres (particules de glycogène)
-x mol.: protéines, polysaccharides, lipides, issues du catabolisme de la ¢
-Complexes prot macromol dont le protéasome (cf plus loin)
-Ribosomes dissociés (2 sous unités détachées) ou actifs, libres dans le cytosol (≠
des ribosomes du RER)
-Composition et structure
-Cytosquelette
-2-1- Carrefour métabolique : voies anaboliques et cataboliques.
-Certaines voies
-sont ∩-dépendantes (carrefour)
- commencent ds le cytosol et se terminent ds d’autres compartiments
Ex :
-Σde mol exportées (hormones stéroïdiennes)
-attachées à la membrane
-Σdes constituants -des mitoch.
-Mb
-des peroxysomes
-
-2-Fonct°

2
2-2-1- La glycolyse
Anaérobie = ne consomme pas d’O2
2-2-2- La voie des pentoses
2-2-3- La Glycogénogénèse
.
2-2- Métabolisme du glucose
Le cytosol est l’1 des 2 sites de product° de l’énergie
Glucose Glucose-6-P Glyceraldehyde-P Pyruvate
NADPH
Nucléotides
Glucose-1-P
Glycogène
Glycogénolyse FOIE
Acide lactique
MUSCLE
O2
ATP
ATP
Ribose-5-P
Synthèse
des AG
Anaérobie
isomérisation
ATP
NADH
Stockage:
glycogénogénèse
polymérisation
-glycolyse anaérobie
-voie des pentoses
-
glycolyseaérobie
2-3-1- Σet Adressage des prot.
2-3- Métabolisme des prot
Les prot sont Σà partir des ARNm dans les ribosomes : traduction.
La Σdes prot. débute tjrs ds le cytosol.
Les prot destinées à être
sécrétées
lysosomales
transmb
Les prot nouvellement Σ Эds séquences reconnues / des facteurs cytosoliques
spécifiques, les orienter vers tel ou tel compartiment ou organite: c’est l’adressage
Ex :
Le signal NLS (Nuclear Localisation Signal) adresse les prot au noyau.
Séquences d’adressage connues pour
-RER,
- Noyau,
- Mitochondries ,
- Lysosomes et
-Peroxysomes.
finissent d’être Σds le RER
Les autres prot. finissent d’être S ds le cytosol.

3
RER
golgi
Protéines sécrétées
ribosomes
Protéines
membranaires
endosome
peroxisomes
mitoch
noyau Protéines
pour lysosomes
Protéines
Transmembr. Protéines
pour sécrétion
Ribosomes
cytosol.
Protéines
extracellulaires
protéasome
lysosomes
2-3-2- Modificat° ds le cytosol des prot pendant et après la traduct°
Acquisit° de la conformat° active des prot (structure II et III)
Favorisées / mol chaperonnes. (= prot nécessaire pour l'assemblage ou le
pliage approprié d'une prot mais qui n'y reste pas associée.
-Phosphorylat° (/ kinases)
-Déphosphorylat° ( / phosphatases)
-Méthylat° ou carboxylat° ds AA
-Liaisons des prot à des AG (farnésyl, ..etc..;)
Définit°
= complexe enzymatique multi-prot cylindrique présent ds le cytoplasme et le
noyau de ttes les ¢ eucaryotes.
2-3-3- Le protéasome
Structure complète du protéasome 26S
-2 unités 19S aux extrémités
-1 unité centrale 20S catalytique

4
2-3-3- Le protéasome
µe du proteasome d’une bactérie.
100 nm
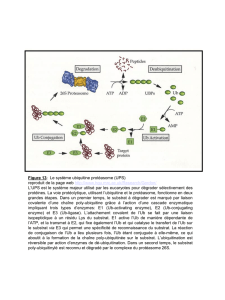
Ubiquitine (Ub) et l'ubiquitinat° (ub°)
L’ activité du protéasome est régulée /l’ubiquitine
L’ Ub. “marque” spécifiquement les prot à dégrader
L’ub° est spécifique et régulée
2-3-2- Le protéasome
L'ub. ЭSéquences hautement conservée au cours de l'évolut° chez ts les
eucaryotes (Indique son importance fondamentale).
L'ub°: régulée / X étapes:
-1- Activat° de l’ub, déclenchée / l’enzyme E1 (ATP-dépendant)
-2- Ub activée est transférée vers une enzyme de conjugaison E2 (> 50
enzymes E2 ≠).
-3-L’enzyme ub.ligase E3 (X centaines d’enzymes E3 spécifiques +++++ ds prot
à dégrader) recrute la protéine à dégrader et
-4- ce complexe se lie au couple “ub.-E2” nouveau complexe E2-E3-protéine
-5-la protéine est liée à l’ub. Plusieurs molécules d’ub. sont ainsi attachées à
la protéine à dégrader.(poly-ubiquitination)
-5- L'ub est reconnue par la partie 19S la protéine attachée est alors "avalée'
dans le canal du protéasome et dégradée , les ubiquitines sont désactivées pui
recyclée
2-3-3- Le protéasome

5
.
Les enzymes du protéasome sont une cible
thérapeutique dans certains cancers: les
inhibiteurs du protéasome induisent
l’apoptose de cellules cancéreuses) : utilisé
actuelleme,t dans une maladie liée à une
prolifération cancéreuse des lymphocytes B
activés : le myelome
☺En 1980:1 chaîne polypeptidique
thermostable, ( facteur protéolytique–1
ATP dépendant), pouvait être conjuguée
aux protéines qui étaient alors désignées
pour la dégradation.
☺Puis, ce facteur est assimilé à une
petite protéine de 76 AA ( identifiée par
Goldstein et coll = d'ubiquitine)
☺En 1983, La cascade enzymatique de
conjugaison entre l'ub et une protéine a
été élucidée
Prix Nobel 2004 CHIMIE Avram Hershko et Aaron Ciechanover
Irwin Rose
 6
6
 7
7
 8
8
 9
9
 10
10
1
/
10
100%