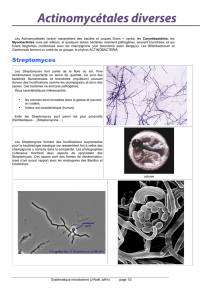
Les actinomycètes

RÉPUBLIQUE ALGÉRIENNE DÉMOCRATIQUE ET POPULAIRE
MINISTÈRE DE L’ENSEIGNEMENT SUPÉRIEUR ET DE LA RECHERCHE SCIENTIFIQUE
N° d'ordre : Année 2014
Université Aboubakr Belkaïd-Tlemcen
Faculté des Sciences de la Nature et de la Vie, des Sciences de la Terre et de l’Univers
Département de Biologie
Laboratoire des Produits Naturels (LAPRONA)
Laboratoire de l’Unité Molécules de Communication et Adaptation des Micro-organismes
MNHN (Paris)
THÈSE
Présentée
par
Mr. BELYAGOUBI Larbi
Pour l’obtention d’un Doctorat en Biologie
Option : Substances Naturelles, Activités Biologiques et Synthèse
*********************
Antibiotiques produits par des bactéries
(actinomycètes et bactéries lactiques) issus de
différents écosystèmes naturels Algériens.
*********************
Soutenue le : Jeudi 26 Juin 2014
Devant le jury composé de :
Année universitaire : 2013-2014
Président : M
r
Chabane Sari D. Professeur Université de Tlemcen
Examinateurs : M
r
Kihal M. Professeur Université d’Oran
M
me
Zirah S.
Maître de conférences
MNHN, Paris
M
me
Bekhechi C.
Maître de conférences
Université de Tlemcen
Directeur de thèse : M
r
Abdelouahid D. E.
Professeur Université de Tlemcen
Co-directeur de thèse :
M
me
Rebuffat S. Professeur MNHN, Paris

i
Remerciements
Avant toute chose, je tiens à remercier «Allah» qui m’a donné la force et la volonté
pour terminer ce modeste travail.
Les travaux présentés dans cette thèse ont été effectués au Laboratoire des Produits
Naturels (LAPRONA) du département de Biologie, de la Faculté des Sciences de la
Nature et de la Vie, des Sciences de la Terre et de l’Univers (SNV/STU), de l’Université
Abou Bakr Belkaïd de Tlemcen. La grande partie de cette étude a été réalisée au sein de
l’unité Molécules de Communication et Adaptation des Micro-organismes, UMR 7245
CNRS/MNHN (anciennement Chimie et Biochimie des Substances naturelles, UMR
5154) dirigée par Madame le professeur Sylvie REBUFFAT. Je tiens à lui exprimer mes
sincères remerciements pour m’avoir permis d’effectuer ces travaux dans cette unité.
L’encadrement scientifique de ce travail a été assuré par Monsieur Djamel Eddine
ABDELOUAHID, Professeur de microbiologie à la Faculté des SNV/STU de Tlemcen.
Je tiens vivement à lui exprimer ma profonde reconnaissance et gratitude pour sa
disponibilité, sa patience, sa compréhension, ses qualités humaines et ses intérêts portés
pour mon sujet de recherche. Je le remercie de m’avoir fait confiance et d’avoir été présent
aussi souvent que possible malgré ses tâches pédagogiques. Son soutien permanent et son
dynamisme m’ont permis d’avancer plus loin dans mes recherches.
Je remercie très chaleureusement ma co-directrice de thèse, Madame Sylvie
REBUFFAT, professeur au MNHN, de l’unité Molécules de Communication et
Adaptation des Micro-organismes, pour avoir accepté de m’accueillir au sein de son
laboratoire pour réaliser des expériences dans ma thèse dans d’excellentes conditions, pour
son soutien et nos conversations constructives.
Je souhaite remercier particulièrement, Madame Séverine ZIRAH, maître de
conférences au MNHN, pour sa grande contribution à ces travaux de recherche, pour son
aide précieuse, pour ses compétences scientifiques, pour sa confiance et ses conseils
scientifiques précieux, pour son soutien et sa gentillesse, pour nos nombreuses discussions
scientifiques, pour ses encouragements et sa bonne humeur.
Je tiens à remercier Monsieur le Professeur Daoudi CHABANE SARI, Directeur
du laboratoire des « Produits Naturels », pour m’avoir fait l’honneur d’accepter la
présidence du jury. Qu’il trouve ici mes sincères impressions de gratitude et de respect.
Mes remerciements vont aussi à Monsieur Mebrouk KIHAL, Professeur à
l’Université d’Oran, d’avoir ménagé son temps pour juger et critiquer ce travail. Je suis
particulièrement reconnaissant et honoré par sa participation au jury de cette thèse.

ii
Que Madame Séverine ZIRAH, maître de conférences au MNHN, trouve ici
l’expression de ma profonde reconnaissance et mes sincères remerciements, de m’avoir fait
l’honneur d’être examinatrice et de participer au jury de ce travail .
Il m’est aussi agréable de remercier Madame Chahrazed
BEKHECHI, maître de
conférences à l’Université de Tlemcen, qu’elle trouve ici l’expression de ma gratitude pour
l’attention qu’elle a bien voulu porter à ce travail, en acceptant de le juger en qualité de
membre de jury.
J’exprime mes vifs remerciements également à tous les membres du Laboratoire des
Travaux Publics de l’Ouest (LTPO) à Tlemcen pour leurs aides dans la réalisation des
analyses pédologiques.
Je tiens à remercier très vivement tous les membres de l’unité « Molécules de
Communication et Adaptation des Microorganismes », particulièrement Madame Manon
VANDERVENNET pour son aide absolument indispensable en microbiologie. Mes
remerciements vont également aux personnes avec qui j’ai pu collaboré notamment,
mademoiselle Carine LOMBARD, Monsieur Jean PEDUZZI, Monsieur Christophe
GOULARD et Monsieur Gérard GASTINE. Je tiens à remercier aussi Madame Yanyan
LI pour sa compétence, son dynamisme et son encadrement dans la partie de biologie
moléculaire. Je remercie sincèrement monsieur Idres MASSINISSA pour sa grande
contribution à l’identification des bactéries lactiques.
Mes remerciements vont aussi à l’égard de Monsieur Hamza BOUABDALLAH
Maître assistant à l’Université de Tlemcen, pour m’avoir donnée les coordonnées
géographiques de nos stations d’échantillonnage.
Je remercie mes collègues et amis pour leur soutien et plus précisément Monsieur
GOUZI Hicham et Monsieur MEBITIL Khalil pour leur aide et son amitié.
J’adresse encore mes remerciements à tous les membres du laboratoire de produits
naturels qui contribuent par leur bonne humeur à créer un cadre de travail agréable.
Pour terminer, je dédie ce manuscrit à ma famille, tout particulièrement mes parents
pour leur soutien, leur amour et leur sagesse qui m’ont permis d’aboutir au grade de
Docteur en biologie et de devenir la personne que je suis. Mes pensées vont à mes frères,
mes sœurs, mes oncles pour leur présence de tous les instants, leur sympathie et leur
encouragement qu’ils m’ont apporté.
Un grand merci à ma femme pour sa sympathie chaleureuse, son appui inestimable
et le sourire dans les moments difficiles et à mes fils pour la joie qu’ils nous procurent.

iii
ـــــــــ
!"#$%&'
()*+. () *+,%%-. // -0 10 23)
4 5-623)02.
728 , 9:/;<.2=>?@ AB)(C2 -D=$%#
().
<# &+% E!3 2=F,G/ )%H !"( @6IJ,KLM(2%,
5 , NOF )PB P5 P(O & /.( Q%@ RN ,% NHE S=F TG :@2U J
V2,(-WStreptomyces P 7< ' ..
-0 8H)/XNX12 -()5># 3+" ),"P (+P 5. )( P
:L %" 2%-E! 1+, RN2 QQ,() )(, %2
AB C2 .RN,%Y 3= (API50 CHL) *+" (ARNr 16S) P 822)
5H% $% $X -2 - G/ Z2 :(65%) Enterococcus P (21%) Leuconostoc P
Lactobacillus
),G/(P Lactococcus )G/ (Weissella )G/(.
?@ (-F 7 LX () NH2 $% AB )( 2=F TG/ C2 4
) ("X [U\" )Staphylococcus aureusP Micrococcus luteusP Bacillus cereus
(Listeria monocytogenes
)/ \"[U )Pseudomonas aeruginosa
Escherichia
(coli
C<=L(Candida albicans) /( F$%HE]YN/N <[.
RN,%9 -WQ%@7D=/TTG/8N,Streptomyces sp.
- 6 72A. 2/)^G6/XT5= "2- AB)( 7K_ C2
&+%$X ().
Q)H) )2=)( ABC2( ' Q3. *+" () K =12. 7
)] `2 3+" $% %2 %" .#J23), 5)J- (HE.
ا تا : (P 5F"2 #P P2=FAB)(C2P !"NH2P
().

iv
Résumé
RésuméRésumé
Résumé
La résistance bactérienne aux antibiotiques et l’émergence de nouvelles maladies
infectieuses justifient l’urgence de disposer de nouvelles molécules antimicrobiennes. Les
peptides antimicrobiens sont des molécules clés dans la défense des organismes vivants face
aux agressions extérieures et aux compétiteurs.
L’objectif de ce travail s’inscrit dans le cadre de la recherche et le criblage des
actinomycètes et des bactéries lactiques capables de synthétiser des substances
antimicrobiennes.
Pour cela, cent huit souches d’actinomycètes ont été isolées à partir d’échantillons de sol
prélevés de différents sites de Tlemcen et Laghouat (forêts, oueds, oasis, montagnes
et barrage). L’identification moléculaire basée sur le séquençage du gène codant l’ARNr 16S
des quarante-sept souches les plus actives de différentes origines, a permis de les rattacher au
genre Streptomyces, souches réputées être la source la plus importante d’antibiotiques.
Aussi dans le cadre de l’exploitation de la diversité microbienne de préparations laitières
traditionnelles algériennes (Jben, Zebda, Dhan et klila), un total de 8 échantillons provenant
de différentes fermes et régions ont fait l’objet d’une analyse microbiologique pour
déterminer leur diversité en bactéries lactiques. Par des méthodes biochimiques (API50 CHL)
et moléculaires (ARNr 16S), on a pu identifier 20 souches qui appartient aux genres
Enterococcus (65%), Leuconostoc (21%), Lactobacillus (2 souches), Lactococcus (1 souche)
et Weissella (1 souche).
Après un criblage initial, le test d’activité antimicrobienne a été effectué sur des souches
d’actinomycètes et de bactéries lactiques contre des bactéries à coloration de Gram positive
(Staphylococcus aureus, Micrococcus luteus, Bacillus cereus et Listeria monocytogenes), des
bactéries à coloration de Gram négative (Pseudomonas aeruginosa, Escherichia coli) et une
levure (Candida albicans) par des méthodes de diffusion sur un agar.
Les souches actives ont montré une forte activité inhibitrice vis-à-vis des
microorganismes pathogènes et les diamètres des zones d’inhibition varient entre 06 et 29
mm pour les actinomycètes et entre 03 et 37 mm pour les bactéries lactiques.
La rechercher par PCR des systèmes génétiques de biosynthèse des peptides lasso par des
Streptomyces sp. n’a pas donné d’amplification d’ADN. Ainsi que des essais d’extraction de
bactériocines produites par des bactéries lactiques n’ont pas abouti à l’isolement de peptides
antimicrobiens.
Les bactéries (actinomycètes et bactéries lactiques) sont une source prodigieuse de
molécules antimicrobiennes d’une grande diversité. Les écosystèmes Algériens abritent une
variété intéressante de microorganismes qui peuvent avoir des activités biologiques.
Mots clés : Sol, Produis laitiers traditionnels, Actinomycètes, Bactéries lactiques, Activité
antimicrobienne, Peptides antimicrobiens.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
 53
53
 54
54
 55
55
 56
56
 57
57
 58
58
 59
59
 60
60
 61
61
 62
62
 63
63
 64
64
 65
65
 66
66
 67
67
 68
68
 69
69
 70
70
 71
71
 72
72
 73
73
 74
74
 75
75
 76
76
 77
77
 78
78
 79
79
 80
80
 81
81
 82
82
 83
83
 84
84
 85
85
 86
86
 87
87
 88
88
 89
89
 90
90
 91
91
 92
92
 93
93
 94
94
 95
95
 96
96
 97
97
 98
98
 99
99
 100
100
 101
101
 102
102
 103
103
 104
104
 105
105
 106
106
 107
107
 108
108
 109
109
 110
110
 111
111
 112
112
 113
113
 114
114
 115
115
 116
116
 117
117
 118
118
 119
119
 120
120
 121
121
 122
122
 123
123
 124
124
 125
125
 126
126
 127
127
 128
128
 129
129
 130
130
 131
131
 132
132
 133
133
 134
134
 135
135
 136
136
 137
137
 138
138
 139
139
 140
140
 141
141
 142
142
 143
143
 144
144
 145
145
 146
146
 147
147
 148
148
 149
149
 150
150
 151
151
 152
152
 153
153
 154
154
 155
155
 156
156
 157
157
 158
158
 159
159
 160
160
 161
161
 162
162
 163
163
 164
164
 165
165
 166
166
 167
167
 168
168
 169
169
 170
170
 171
171
 172
172
 173
173
 174
174
 175
175
 176
176
 177
177
 178
178
 179
179
 180
180
 181
181
 182
182
 183
183
 184
184
 185
185
 186
186
 187
187
 188
188
 189
189
 190
190
 191
191
 192
192
 193
193
 194
194
 195
195
 196
196
 197
197
 198
198
 199
199
 200
200
 201
201
 202
202
 203
203
 204
204
 205
205
 206
206
 207
207
 208
208
 209
209
1
/
209
100%