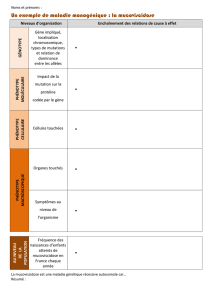
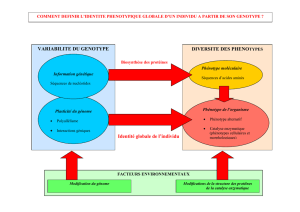
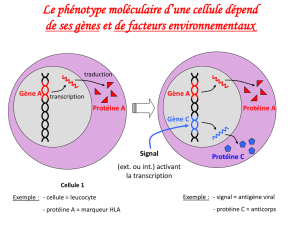
Diversité phénotypique et adaptation chez Escherichia Coli étudiées

Diversit´e ph´enotypique et adaptation chez Escherichia
Coli ´etudi´ees en millifluidique digitale
Denis Cottinet
To cite this version:
Denis Cottinet. Diversit´e ph´enotypique et adaptation chez Escherichia Coli ´etudi´ees en mil-
lifluidique digitale. Biophysique. Universit´e Pierre et Marie Curie - Paris VI, 2013. Fran¸cais.
<pastel-00981358>
HAL Id: pastel-00981358
https://pastel.archives-ouvertes.fr/pastel-00981358
Submitted on 22 Apr 2014
HAL is a multi-disciplinary open access
archive for the deposit and dissemination of sci-
entific research documents, whether they are pub-
lished or not. The documents may come from
teaching and research institutions in France or
abroad, or from public or private research centers.
L’archive ouverte pluridisciplinaire HAL, est
destin´ee au d´epˆot et `a la diffusion de documents
scientifiques de niveau recherche, publi´es ou non,
´emanant des ´etablissements d’enseignement et de
recherche fran¸cais ou ´etrangers, des laboratoires
publics ou priv´es.

THÈSE DE DOCTORAT
DE
L’UNIVERSITÉ PIERRE ET MARIE CURIE
École doctorale de chimie physique et chimie analytique de Paris Centre
Présentée par
Denis COTTINET
Pour obtenir le grade de
DOCTEUR de l’UNIVERSITÉ PIERRE ET MARIE CURIE
Sujet de thèse :
Diversité phénotypique et adaptation chez Escherichia coli
étudiées en millifluidique digitale
Soutenue le 11 décembre 2013 devant le jury composé de :
Mme Nicole COTTE-PATTAT Rapportrice
M. Erick DENAMUR Rapporteur
M. Jean-François JOANNY Président du jury
M. Paul RAINEY Examinateur
M. Arjan DE VISSER Examinateur
M. Matthieu JULES Examinateur
M. Jérôme BIBETTE Directeur de thèse
M. Jean BAUDRY Co-Directeur de thèse


Remerciements
Cette thèse est terminée je suis ravi du chemin parcouru. Ce bonheur tiens nécessairement à un peu
d’autosatisfaction, avouons le, mais surtout à tout ce que j’ai reçu des personnes qui m’ont
entourées et que j’ai rencontrées pendant ces trois ans. Alors je voudrais essayer de toutes les
nommer et remercier.
En premier lieu, je remercie Jérôme Bibette pour m’avoir accueilli au LCMD en stage de master dès
février 2010 puis pour les trois ans de doctorats. En tant que directeur de thèse ces conseils et sa
vision du sujet ont évidemment été très importants. Mais la confiance et l’autonomie qu’il accorde
volontiers m’ont fait progresser sur le plan personnel, et les succès obtenus ou observés au
laboratoire m’ont rendu plus optimiste et plus ambitieux.
Autant que mon directeur de thèse, Jean Baudry a joué un rôle important dans ma formation durant
les quatre années passées au laboratoire. Sa curiosité scientifique extrêmement vaste et sa passion
pour les méthodes de mesures pertinentes et innovantes sont contagieuses et je lui suis
reconnaissant de me les avoir transmis au moins en partie. Dans le cadre de ma mission doctorale
d’enseignement, je le remercie de m’avoir fait confiance pour les TP de microscopie en Master 2 car
c’est une expérience d’enseignement très enrichissante.
Je remercie Florence Condamine, technicienne au LCMD, pour son travail d’organisation et de
maintenance au laboratoire qui nous permet de travailler dans des conditions exceptionnelles. Mais
je remercie surtout Florence pour les expériences qu’elle a réalisée pour ma thèse et l’enthousiasme
qu’elle a manifestée. Certaines discussions et certaines de ces exigences méthodologiques m’ont fait
grandement progresser.
Je remercie également Nicolas Bremond qui a suivi mes travaux de loin mais qui à chaque fois que je
l’ai sollicité sur des questions scientifiques ou professionnelles a toujours été disponible et a apporté
un avis extérieur très précieux.
Mon jury de thèse a également contribué à ma thèse en portant un regard critique et constructif sur
ce travail de physico-chimiste en microbiologie et je l’en remercie : Jean-François Joanny pour
m’avoir fait l’honneur de présider ce jury, Erick Denamur et Nicole Cotte-Pattat pour avoir accepté le

travail de rapporteur avec curiosité, Matthieu Jules pour sa disponibilité et son intérêt au moment de
constituer ce jury, Arjan de Visser et Paul Rainey pour les raisons détaillées ci-après.
Tout d’abord, je remercie Arjan pour ses conseils et son soutien lorsque j’ai commencé sur ce sujet
sans rien connaître en microbiologie. Il a joué un rôle de « phare », distant mais indispensable pour
ce repéré et garder le cap. Il est pour moi un encadrant à part entière et sa présence le jour de ma
soutenance m’a fait très plaisir. Je garde le souvenir d’une semaine formidable passée à Wageningen,
qui sans pouvoir désigner précisément comment m’a véritablement permis de me saisir de mon
sujet. Je remercie Merijn Salverda pour son accueil à cette occasion.
Je remercie également Paul Rainey pour l’honneur qu’il m’a fait en prenant part à mon jury et pour
les discussions dans le dernier mois de ma thèse.
Au cours de ma thèse j’ai bénéficié des conseils avisés et de l’aide précieuse de nombreuses
personnes pour apprendre la biologie et utiliser ses techniques. Je remercie Joelle Vinh, Yann Verdier
et Iman Haddad du SMPB à l’ESPCI pour leur soutien et leur patience pour la caractérisation sur le
gène ag43 et le plaisir que j’ai eu à travailler avec eux. J’ai rencontré Christophe Beloin de l’Institut
Pasteur par hasard, mais j’aurais pu et dû le sollicité plutôt au sujet du gène ag43. Son intérêt et ses
conseils sur ce thème m’ont indéniablement permis de franchir un des caps qui font l’avancement
d’une thèse. Clément Nizak, invité au laboratoire de Biochimie (LBC) de l’ESPCI, m’a lui fait profiter
de son expérience et de sa connaissance de la communauté scientifique nationale et internationale
au sein de laquelle ce travail de thèse s’inscrit. Je remercie Andrew Griffith pour le regard extérieur
positif qu’il a porté régulièrement sur mon travail. Enfin je suis content de signaler le rôle particulier
de Pierre Sivadon et Régis Grimaud de l’IPREM à Pau. Ils sont les premiers avoir suggéré l’implication
de variations de phases comme celle d’ag43 dans nos résultats sur la diversité phénotypique, je les
remercie pour le temps qu’il nous ont fait gagner à ce moment là et pour le plaisir que j’ai eu à
travailler en marge de ma thèse sur leur problématique.
Je remercie toutes les personnes qui ont fait de la millifluidique au laboratoire car nous avons
partagé et partageons nos problématiques expérimentales ce qui est très agréable et fait souvent
gagner un temps précieux. Fabien et Larysa ont été les pionniers de la millifluidique au LCMD. Wang
et Laurent ont considérablement intégré le dispositif et le programme de pilotage. Je remercie
Laurent également pour m’avoir accueilli en stage de master et m’avoir parfaitement préparer à ce
moment là aux trois années de thèse qui ont suivi, mais aussi pour le meneur et animateur qu’il est
au travail, auprès duquel on ne peut que cultiver l’optimisme et l’ambition.Shima, Jairo et Lianmei
ont aborder des thèmes différents avec la technologie millifluidique et les échanges sur nos
expériences respectives ont été source d’inspiration et de motivation dans une ambiance très
agréables.
J’ai beaucoup apprécié de travailler avec les stagiaires qui sont passés pour quelques mois au
laboratoire sur des sujets variés en marge de ma thèse car on ne peut pas faire tout le temps la
même chose : Sarah Abidh, Ernest Mordret, Christoph Kleinschmidt, François Mazuel, Alexis
Mazurier, Mélanie Jacquet et Joshua Ricouvier.
Les rôles de Lise, pour l’administratif et Jean-Yves pour la maintenance et l’entretien sont
évidemment indispensables. Ils les remplissent d’une manière qui force l’admiration mais surtout je
retiens qu’en plus ce sont des personnes agréables qui ont et vous donne chaque fois le sourire.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
 53
53
 54
54
 55
55
 56
56
 57
57
 58
58
 59
59
 60
60
 61
61
 62
62
 63
63
 64
64
 65
65
 66
66
 67
67
 68
68
 69
69
 70
70
 71
71
 72
72
 73
73
 74
74
 75
75
 76
76
 77
77
 78
78
 79
79
 80
80
 81
81
 82
82
 83
83
 84
84
 85
85
 86
86
 87
87
 88
88
 89
89
 90
90
 91
91
 92
92
 93
93
 94
94
 95
95
 96
96
 97
97
 98
98
 99
99
 100
100
 101
101
 102
102
 103
103
 104
104
 105
105
 106
106
 107
107
 108
108
 109
109
 110
110
 111
111
 112
112
 113
113
 114
114
 115
115
 116
116
 117
117
 118
118
 119
119
 120
120
 121
121
 122
122
 123
123
 124
124
 125
125
 126
126
 127
127
 128
128
 129
129
 130
130
 131
131
 132
132
 133
133
 134
134
 135
135
 136
136
 137
137
 138
138
 139
139
 140
140
 141
141
 142
142
 143
143
 144
144
 145
145
 146
146
 147
147
 148
148
 149
149
 150
150
 151
151
 152
152
 153
153
 154
154
 155
155
 156
156
 157
157
 158
158
 159
159
 160
160
 161
161
 162
162
 163
163
 164
164
 165
165
1
/
165
100%