L2 de la Licence Sciences de la vie

Travaux Dirigés de Biochimie (réactions cellulaires) - BI301
TD 1 - Cinétique enzymatique
Exercice A
Une enzyme catalyse la réaction suivante sur un substrat : S + H2O P et la quantité de produit formé (en
µmoles de produit par tube) est mesurée en fonction du temps et pour différentes concentrations de substrat.
Les résultats obtenus sont résumés dans le tableau ci-dessous.
Temps (min) 2,5 5 10 15 20
S]o (mM)
20 1,8 3,6 6,2 8 9
60 4 7,8 13,5 17 19
200 6,4 12,8 25 33,8 38
300 7,2 14 29 37
1) Cinétique d’apparition de P
a) Tracez les cinétiques de réaction de cette enzyme et expliquez l’allure des tracés obtenus.
b) Déterminez les vitesses initiales de réaction pour chaque concentration de S
2) Courbe de Michaelis
a) Tracez la représentation de la vitesse initiale en fonction de la concentration de S. Commentez cette courbe.
b) Déterminez les paramètres cinétiques.
3) Tracé de Lineweaver-Burk
a) Déterminez à partir de cette représentation les paramètres cinétiques.
b) Comparez les valeurs obtenues avec les précédentes et commentez ces résultats.
4) Déterminez l’activité spécifique de l’enzyme ainsi que la constante catalytique. La concentration d’enzyme
initiale est de 60 µM, le volume réactionnel 3 ml et le volume d’enzyme utilisé 100 µl. La masse molaire de
l’enzyme est 100.000 g / mole.

Exercice B
L'hexokinase catalyse la fixation de phosphate sur le glucose. Pour suivre la purification de cette enzyme, on
mesure la fixation de phosphate radioactif provenant l'ATP marqué au 32P sur le glucose :
glucose + ATP glucose-6-phosphate + ADP
Le mélange réactionnel (1 ml) renferme toutes les substances nécessaires à la réaction. Le volume d'extrait
enzymatique utilisé est de 0,1 ml. L'activité spécifique de l'ATP est de 2000 coups par minute par nmol (CPM /
nmol). Les résultats obtenus sont présentés dans le tableau suivant :
Temps (minutes) 5 10 15 20 25 30 40
___________________________________________________________________________
CPM / 40 µl de
mélange réactionnel 1700 3400 5100 6700 7600 7800 7800
1) Calculer l'activité enzymatique de l'extrait en unité par ml. 1 UE est la quantité d'enzyme catalysant la
fixation de 1 nmol de P / minute dans les conditions expérimentales.
2) Calculer l'activité spécifique sachant que la quantité de protéine utilisée dans l'expérience est de 60 µg.
3) Le KM de l'enzyme vis-à-vis du substrat est de 8.10-6 M. Démontrer que le glucose est en quantité suffisante
pour déterminer l'activité enzymatique (on admettra que, dans une cinétique enzymatique, Vmax est atteinte
quand la concentration en substrat est au moins égale à 10 KM).

TD 2 - Métabolisme
Problème 1 : Fermentation alcoolique des levures
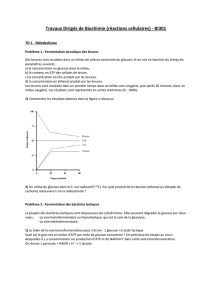
Des levures sont incubées dans un milieu de culture contenant du glucose, et on suit en fonction du temps les
paramètres suivants :
a) la concentration en glucose dans le milieu.
b) le contenu en ATP des cellules de levure.
c) la concentration en CO2 produit par les levures.
d) la concentration en éthanol produit par les levures.
Les levures sont incubées dans un premier temps dans un milieu sans oxygène, puis après 20 minutes, dans un
milieu oxygéné. Les résultats sont représentés en unités arbitraires (0 - 100%).
1) Commentez les résultats obtenus dans la figure ci-dessous :
2) On utilise du glucose dont le C1 est radioactif (14C). Sur quel produit de la réaction (éthanol ou dioxyde de
carbone) retrouvera-t-on la radioactivité ?
Problème 2 : Fermentation des bactéries lactiques
La plupart des bactéries lactiques sont dépourvues de cytochromes. Elles peuvent dégrader le glucose par deux
voies : - La voie homofermentaire ou homolactique, qui est la voie de la glycolyse.
- La voie hétérofermentaire.
1) Le bilan de la voie homofermentative peut s’écrire : 1 glucose->2 acide lactique
Quel est le gain net en moles d’ATP par mole de glucose consommé ? On précisera les étapes au cours
desquelles il y a consommation ou production d’ATP et de NADH+H+ dans cette voie homofermentative.
On donne 1 pyruvate + NADH + H+ -> 1 lactate.

2) La voie hétérofermentative d’une bactérie lactique comporte la séquence de réactions précisée sur le
schéma ci-dessous :
Le 3-phosphoglycéraldéhyde formé est transformé en lactate par une séquence de réactions commune avec
celle de la voie homofermentaire.
Quel est le gain net en moles d’ATP par mole de glucose consommé par la voie hétérofermentaire, lorsque
cette voie donne les produits de fermentation figurant dans le bilan suivant :
1 glucose -> 1 CO2 + 1 acide lactique + 1 éthanol
Etablir le bilan en NADH + H+ participant aux réactions de cette voie.

TD 3 - Métabolisme et couplages énergétiques
Ce TD concerne 2 voies métaboliques vues dans les cours :
A) la voie de synthèse des ARN.
B) la voie de fermentation alcoolique.
En plus de ces voies métaboliques, il faut connaitre l'existence et les réactions de trois autres enzymes :
* La Transhydrogénase qui catalyse la réaction : NADH + NADP+ ↔ NAD+ + NADPH
* La glycérol kinase qui catalyse la réaction : Glycérol + ATP ↔ Glycérol 3 P+ ADP
* La glycérol 3 phosphate déshydrogénase qui catalyse la réaction :
Glycérol 3 P + NAD+ ↔ Dihydroxy acétone phosphate + NADH
1) Quel est le bilan métabolique de la synthèse d'un petit ARN de séquence pppApGpCpUpCpU à partir de
glucose, et des bases Uracile, Adénine et Guanine ?
2) Quel est le bilan métabolique de la conversion de glucose en glycérol ?
3) Comparer ce bilan avec les bilans des conversions de glucose en Ethanol et de glucose en acétaldéhyde. Que
peut-on en déduire ?
4) En vous appuyant sur les bilans métaboliques précédemment obtenus, quels pourraient les ’autres’ produits
synthétisés par une cellule de levure poussant sur un milieu contenant du glucose, les bases nucléotidiques U,
A, et G qui fabrique le petit ARN ‘AGCUCU’ ?
5) En déduire les transferts possibles entre les voies anaboliques et les voies cataboliques ?
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
1
/
15
100%