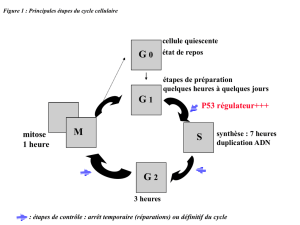
Télomérase et Cancer


Télomérase et Cancer
Télomères
Télomérase
Régulation Télomérase
Régulation Télomères
Télomères

Quelques dates
Notion d’extrémités des chromosomes protégées WATSON
Hypothèse d’OLOVNIKOV (1971)
Perte progressive d’ADN au cours
des réplications
Mort des cellules par perte de
gènes vitaux
Depuis 1996
Modèles
Protozoaire : Tetrahymenia thermophila (in vivo)
Levures : • Saccharomyces cerevisiae (in vivo)
• Schizosaccharomyces pombe (in vivo)
Souris : TR limitant (in vivo, souris invalidées)
Homme : TERT limitante (in vitro)
Télomères et Télomérases sont très étudiés
Protozoaire : Tetrahymenia thermophila
Mise en évidence d’un complexe Ribonucléoprotéique
Constitué - Reverse Transcriptase (TERT)
- ARN matriciel (TR)
Greider et
Blackburn
1985
Point de départ des travaux sur télomères
Télomères Télomérase et Cancer

Télomères Télomérase et Cancer
Télomères
Pour échapper aux systèmes de
réparation des ADN endommagés
et à la fusion inter-chromosomique
Nécessité pour la cellule de différentier
les extrémités de ses chromosomes des cassures dans l’ADN
Système NHEJ
Arrêt du cycle, Apoptose

Organismes Uni¢aires
Ciliés (Euplotes…) TTTTGGGG
Levure (S.Cerevisiae) TG
Flagellés(Tripanosome, Leishmania) TTAGGG
Algues Chlamydomonas … TTTTAGGG
Organismes Multi¢aires
Vertébrés -homme TTAGGG
-souris TTAGGG
-tous les autres TTAGGG
Truite TTAGGG
Plantes Arabidopsis thaliana… TTTAGGG
Insectes (lepidoptères) TTAGG
Champignons (Neurospora) TTAGGG
Nématodes (Ascaris,Caenorhabditis Elegans) TTAGGC
Télomères Télomérase et Cancer
Séquences ADN - situées aux extrémités des chromosomes
- non codantes
- longueur variable selon les espèces
-constituées essentiellement d’un motif répété
Télomères ?
< 100 pb
< 500 pb
< 100 pb
< 500 pb
> 10 kpb
> 50 kpb
> 20 kpb
3 kpb
> 10 kpb
?
?
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
1
/
45
100%