Stage - DLST

EFS Santé
2012
Département Oncogenèse
et Biotechnologie
Immunobiologie et Immunothérapie des
cancers
Pierre-François Meyer

Laboratoire R&D-EFS Santé
Le laboratoire dans lequel j’ai effectué mon stage d’excellence au cours du mois de
juillet 2012 se spécialise dans le domaine de la biologie cellulaire et plus particulièrement en
immunologie. L’intitulé de la recherche au sein de ce laboratoire est « Immunobiologie et
Immunothérapie des cancers ».
L’équipe étudie les cellules immunitaires impliquées lors du développement tumorale
dans l’objectif de développer de nouvelles stratégies thérapeutiques. L’étude est portée
principalement sur les cellules dendritiques, qui sont à la base de la réponse immunitaire, et
sur les lymphocytes T cytotoxiques. La particularité de leur recherche est qu’ils travaillent sur
des cellules dendritiques plasmocytoïdes (PDC) qui, comme ils ont pu le montrer auparavant,
jouent un rôle majeur dans la réponse anti-tumorale en sécrétant de nombreux intermédiaires
et en stimulant les lymphocytes T cytotoxiques spécifiques.
Le but de la recherche est de développer un vaccin et ou une thérapie pour le cancer en
se concentrant principalement sur le mélanome. L’objectif est d’utiliser les PDCs de manière
à ce qu’ils engendrent une réaction immunitaire spécifique au cancer. Dans le cas du
mélanome, l’objectif est de rendre les PDCs sensible à la présence de protéines spécifiques
des cellules tumorales afin d’assurer une réponse immunitaire plus rapide et plus forte.
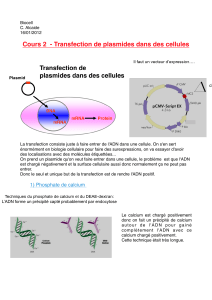
Pour réaliser cela il faut donc effectuer une transduction des PDC avec un gène d’intérêt afin
qu’ils puissent réagir.
Le travail au sein du laboratoire est divisé. J’ai pu travailler spécifiquement sur deux
parties bien distinctes.
Biologie Moléculaire
L’équipe travaille au sein d’un projet Européen et est donc en collaboration avec d’autres
équipes notamment une basée à l’University College London (UCL). L’équipe d’UCL a déjà
mis en place une partie biologie moléculaire visant à la production de nombreux plasmides
portant des gènes d’intérêts qui peuvent ensuite être utilisés pour la transduction de cellules.
Lorsque je suis arrivé, le laboratoire R&D était, à son tour, en train de mettre en place un
travail en biologie moléculaire afin de créer une réserve de différents gènes qu’ils pourront
utiliser par la suite afin d’être autonome.
Biologie cellulaire
Une fois que les plasmides sont produits en quantité suffisante ils peuvent ensuite être utilisés
pour la transduction des PDCs afin de s’inscrire dans la stratégie globale de l’équipe.
L’objectif pour nous était d’incorporer le ou les gènes d’intérêts au sein des PDCs puis de
vérifier s’il y avait ensuite réaction par rapport à des cellules « malades ».

Activités au sein du laboratoire
Biologie Moléculaire
Transformation bactérienne
Dans un premier temps je me suis retrouvé impliqué dans la mise en place d’une
réserve de différents plasmides portant des gènes d’intérêts. A la base l’équipe disposait de
tous ces plasmides mais dans une quantité extrêmement faible. Pour cette raison mon rôle (en
binôme avec une autre stagiaire issue de deuxième année d’IUT de biologie) était de procéder,
en premier lieu, à une transformation bactérienne.
Pour cela, on utilise une lignée cellulaire C2988J qui est particulièrement utile pour ce genre
de manipulation. On note que tous les plasmides que l’on souhaite incorporer dans les
bactéries codent pour un gène de résistance à la carbenicillin.
J’ai ensuite procédé à la manipulation suivante :
1) 25uL de bactéries sont incuber dans la glace dans un eppendorf de 0.5mL.
NB : On utilise une micropipette de 200uL pour faire ce prélèvement car on ne dispose
que de pipettes de 10, 20, 200 et 1000uL
2) 2uL de plasmide sont ajoutés aux bactéries (toujours dans la glace) et une incubation
de 20 à 60 minutes commence.
3) On procède ensuite à un choque thermique. L’objectif de ce choc est de dilater la
membrane bactérienne afin de permettre aux plasmides de rentrer au sein des
bactéries.
On déplace donc l’eppendorf de 0.5mL dans un bain marie à 42°C pendant exactement
25s puis on remet le tout dans la glace au moins 2 minutes.
4) On ajoute ensuite 250uL de milieu de croissance bactérien qui optimise la
transformation des cellules compétentes: le SOC puis on met le milieu sous une
agitation de 200 rpm (Rotations par minutes), à 37°C pendant 40 à 80 minutes.
Cette transformation bactérienne a pour seul et unique but la production en masse du plasmide
incorporé. Cette dernière étape vise donc à commencer le développement bactérien. On
procède ensuite à la distribution du mélange préalablement créer sur un milieu d’agarose dans
des boites de pétri. Ce milieu d’agarose favorise la croissance bactérienne et contient de la
carbenicillin. Ainsi, toutes les bactéries non transformées seront éliminées par l’antibiotique
ce qui permet de faire une première sélection des cellules transformées.

5) On réparti le milieu SOC + bactérie dans une boite de pétri en faisant bien attention à
répartir largement avec une baguette afin d’obtenir un maximum de colonies
bactériennes isolées.
Les miniprep, midiprep et les digestions enzymatiques
Culture de masse
Pour optimiser la production de plasmides, l’idée est de produire en grande quantité
des bactéries ayant incorporées le plasmide d’intérêt. Dans un premier temps on va d’abord
chercher à vérifier que les bactéries ont bel et bien incorporées le plasmide. La croissance sur
le milieu d’agarose contenant de la carbenicillin participe à la sélection. Dans un second
temps, l’objectif va être de vérifier par digestion puis par southern blot que les bactéries
possèdent le plasmide.
Suite à la mise en culture en boite de pétri on va donc prélever une colonie que l’on va
ensuite mettre dans un milieu de culture bactérien en présence de carbenicillin. On fait
d’abord cela en petite quantité. En effet, on ne va pas procéder à une culture bactérienne de
masse avant d’être absolument certain que les bactéries ont répondues correctement à la
transformation.
Afin de maximiser nos chances de trouver, au sein de nos premières cultures, une colonie de
bactéries ayant complètement intégrée le plasmide, il est important de choisir une colonie très
isolée et, si possible, de faire plusieurs cultures avec des colonies différentes. Dans notre cas,
à chaque fois que l’on faisait une nouvelle transformation nous mettions ensuite 4 colonies en
culture.
La marche à suivre pour cette mise en culture est la suivante :
- On utilise une pipette pour mettre 5 mL de milieu LB (milieu de culture bactérien)
dans un tube à essaie de 15 mL à fond rond
- On ajoute 10uL de carbenicillin. Ceci nouveau dans un but de sélection. On s’assure
ainsi qu’aucune bactérie n’ayant pas incorporé le plasmide ne puisse contaminer notre milieu
de culture.
- On prélève une colonie au sein de notre boite de pétri à l’aide d’un cône en plastique
que l’on plonge dans le tube. Ce tube est ensuite fermé mais pas de manière hermétique pour
permettre la respiration bactérienne.
NB : La colonie doit être la plus isolée possible afin de minimiser le risque de contamination
par une autre colonie qui, elle, n’aurait pas été transformée, et inversement.
- Les cultures sont mises sous une agitation de 250 rpm à 37°C pendant 12h à 16h.
Miniprep
On va donc chercher à récupérer l’ADN contenue dans les bactéries. Pour cela, il existe des
kits. Dans notre cas nous utilisions le QIAprep Spin Miniprep kit de Qiagen.

L’objectif est simple : il faut lyser la bactérie et se défaire de l’ADN bactérien pour récupérer
uniquement l’ADN plasmidien.
Les étapes de cette manipulation sont les suivantes :
1) On prélève 1.5mL de la culture bactérienne que l’on centrifuge à 13 000 G pendant 3
minutes afin de faire former un culot par les bactéries.
2) On remet les bactéries en suspension dans 250uL dans un tampon nommé P1 qui est
un tampon de suspension.
3) On ajoute ensuite 250uL d’un tampon de lyse qui va permettre de lysée les bactéries et
de libérer l’ADN. En revanche, il faut faire attention à ne pas faire durer cette étape
plus de 5 minute car cela risque d’endommager l’Adn.
4) Il suffit de rajouter 350uL d’un tampon qui neutralise le tampon de lyse et qui arrête la
réaction.
5) On centrifuge ensuite 10 min à 13000 rpm
6) Le surnageant est ensuite passé à travers une colonne qui va retenir l’ADN plasmidien.
On effectue une première centrifugation de 60s.
7) On nettoie ensuite la colonne avec 750uL d’un quatrième tampon nommé PE. Par la
suite il faut faire deux centrifugations successives d’environ 60s chacune pour nettoyer
puis pour éliminer des résidus de ce tampon.
8) On utilise enfin 50uL de tampon d’élution pour éluer l’ADN dans un eppendorf
NEUF.
NB : Toute cette manipulation se fait avec des eppendorfs de 0.5mL sauf pour l’étape 8 qui se
fait dans un eppendorf de 1.5mL.
Digestion enzymatique
Afin de vérifier que la transformation est efficace il suffit de montrer que l’ADN
plasmidien récupéré au sein de nos bactéries correspond à celui que l’on souhaitait incorporer.
Pour cela on soumet l’ADN extrait à l’activité de plusieurs enzymes différentes :
- EcoR1 et HindIII simultanément
- NhEI seul
- BgL2 et Xho1 simultanément
On n’oublie pas de garder un test qui, lui, n’est soumis à aucune activité enzymatique.
Ainsi, connaissant les sites de restrictions de chacune de ces enzymes on peut ensuite
effectuer une vérification par southern blot.
Lorsque l’on fait la digestion on a utilisé des quantités considérées comme « standard »
d’enzyme en rapport avec la quantité de plasmide.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
1
/
12
100%