Glucose-6-Phosphate

1. Glucose → Glucose-6-Phosphate
Hexokinase
2. Glucose-6-Phosphate → Fructose-6-Phosphate
Phosphoglucose isomérase
3. Fructose-6-Phosphate → Fructose-1,6-DiPhosphate
Phosphofructokinase
4. Fructose-1,6-DiPhosphate → Dihydroxy Acétone Phosphate + Glycéraldéhyde-3-phosphate
Aldolase
5. Dihydroxy Acétone Phosphate → Glycéraldéhyde-3-phosphate
Triose phosphate isomérase
6. Glycéraldéhyde-3-phosphate → 1,3 DiPhosphate Glycérate
3 Phospho glycéraldéhyde déshydrogénase
7. 1,3 DiPhosphate Glycérate → 3 Phosphoglycérate
Phosphoglycérate kinase
8. 3 Phosphoglycérate → 2 Phosphoglycérate
Phosphoglycérate mutase
9. 2 Phosphoglycérate → Phospho-énol-Pyruvate
Enolase
10. Phospho-énol-Pyruvate → Pyruvate
Pyruvate kinase
NADH
6 ATP / Glucose
Etapes 1 à 5 : ACTIVATION
- Consommation de 2 ATP
Etapes 6 à 10 : OXYDATION (Se produisent en double)
- Restitution de 2 ATP
- Formation de 2 ATP
- Formation de 2 NADH
Bilan : 2ATP + 2 NADH (=2ATP/NADH cytoplasmique) = 6 ATP / Glucose
Régulation enzymatique (Toutes IRREVERSIBLES)
1. Hexokinase (G6P ⊖)
3. Phosphofructokinase (ATP ⊖ ; Citrate ⊖ ; AMP ⊕)
10. Pyruvate kinase (ATP ⊖ ; Phosphorylation ⊖)
GLYCOLYSE
(CYTOPLASME CELLULAIRE)
ATP
ATP
ATP
ATP
X 2

Pyruvate → Acétyl-CoA (*)
Pyruvate déshydrogénase
Pyruvate → Oxaloacétate (**)
Pyruvate carboxylase
Pyruvate → Lactate
Lacticodeshydrogénase
Pyruvate → Alanine (***)
Alanine aminotransférase
DEVENIR DU PYRUVATE
NADH + CO2
ATP + HCO3
NAD+
Mitochondriale
Cytoplasmique (anaérobiose)
(**) Co facteurs
- Biotine
(*) Co facteurs
- FAD
- NADH
- Lipoate
- TPP (Thiamine)
- CoA - SH
(***) Co facteurs
- Phosphate de pyridoxal

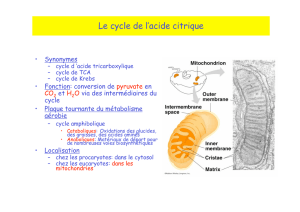
1. Acétyl-CoA + Oxaloacétate → Citrate
Citrate synthétase
2. Citrate → Isocitrate
Aconitase
3. Isocitrate → α-cétoglutarate
Isocitrate déshydrogénase
4. α-cétoglutarate → Succinyl-CoA (*)
α-cétoglutarate déshydrogénase
5. Succinyl-CoA → Succinate
Succinate thiokinase
6. Succinate → Fumarate
Succinate déshydrogénase
7. Fumarate → Malate
Fumarate hydratase (Fumarase)
8. Malate → Oxaloacétate
Malate déshydrogénase
NADH
NADH
GTP
FADH2
NADH
H2O
H2O
H2O
CO2
CO2
12 ATP / Cycles / molécule d’Acétyl-CoA
GTP - - -> ATP
=> Dinucléotide kinase
Régulation enzymatique
(Irréversible)1. Citrate synthétase (ATP ⊖)
(Réversible) 3. Isocitrate déshydrogénase (ATP ⊖ ; NADH ⊖ ; AMP ⊕ ; ADP ⊕)
(Irréversible)4. α-cétoglutarate déshydrogénase (ATP ⊖ ; NADH ⊕)
CYCLE DE KREBS
(MATRICE MITOCHONDRIALE)
(*) Co facteurs
- FAD
- NADH
- Lipoate
- TPP
- CoA - SH

1. Glucose → Glucose-6-Phosphate
Glucokinase
2. Glucose-6-Phosphate → Glucose-1-Phosphate
Phosphoglucomutase
3. Glucose-1-phosphate + UTP → UDP-Glucose + Pyrophosphate (P∼Pi)
UDP-Glucose-Pyrophosphorylase
3 bis. Pyrophosphate (P∼Pi) → 2Pi
o Pyrophosphatase
4. UDP-Glucose → chaîne d’au moins 4 glucoses unis par des liaisons 1 - 4
Glycogène synthétase
4 bis. Reconstitution de l’UTP : UDP → UTP
5. 7 résidus liées en α 1 - 4 → liaison α 1 - 6
Enzyme branchante [AMYLO (1,4 / 1,6) transglucosylas]
Bilan :
Pour chaque molécule de glucose ajouté à la chaine il y a consommation de 2 ATP (Etape 1 ; Etape 4 bis)
GLYCOGENOGENESE
(FOIE ET MUSCLES)
CYTOPLASMIQUE
ATP
UDP
ATP

1. Liaisons α 1 - 4 → Glucose-1-Phosphate (G1P)
Phosphorylase
2. Coupure de 3 résidus glucose de la branche restante, et les transferts au bout d’une
autre branche en 1 – 4 => il ne reste plus qu’un seul glucose branché en 1 - 6
Transférase
3. Liaison α 1 - 6 → Glucose
Enzyme débranchante (α 1,6 glucosidase)
4. Glucose-1-Phosphate (G1P) → Glucose-6-Phosphate (G6P)
Phosphoglucomutase
GLYCOGENOLYSE
(FOIE ET MUSCLES)
Devenir du Glucose-6-Phosphate (G6P)
↳ Incapable de traverser la membrane plasmique
1. Dans le foie
G6P → Glucose + Pi
Glucose 6 phosphatase
2. Dans le muscle
G6P → Pyruvate
Le muscle ne possède pas de glucose 6 phosphatase, le
glucose entame donc le processus centripète de la GLYCOLYSE
aboutissant au pyruvate qui s’orientera dans un catabolisme
aérobie ou anaérobie selon les besoins.
Note : Une molécule de glycogène donne -
- Par liaison 1 – 4 => Une molécule de G1P
- Par liaison 1 – 6 => Une molécule de Glucose
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
1
/
24
100%