Thème 3 : Glycémie et diabète TP 2 : La spécificité d`une enzyme : le

Thème 3 : Glycémie et diabète
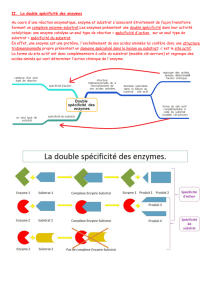
TP 2 : La spécificité d’une enzyme : le complexe enzyme-substrat
On sait qu’une enzyme (E) est capable de catalyser une réaction chimique, en se liant de manière spécifique
à un substrat (S), puis en permettant sa transformation en produit (P).
Un évènement important de l’acte catalytique des enzymes est donc la formation d’un complexe enzyme-
substrat : E + S ES E + P
On veut montrer qu’une enzyme est spécifique d’un substrat et que l’activité catalytique d’une enzyme est
liée à sa conformation c'est-à-dire sa structure spatiale tridimensionnelle.
Q 1) Les caractéristiques de l’enzyme carboxypeptidase
- A partir du logiciel RASTOP, ouvrir dans le fichier « carboxy3D », la molécule carboxypeptidase
intitulée « CPASEUL.pdb ».
- « Molécule/Séquences » puis « Atomes/colorer par chaine » puis
« Ruban/Afficher » pour observer la configuration de la molécule.
Décrire la molécule CPA. - Choisir la représentation « en sphères » la plus judicieuse pour faire
apparaître la « poche » correspondant au site de fixation de l’enzyme à
son substrat.
Appeler le professeur pour vérifier
- Ouvrir l’enzyme liée à son substrat « CPASUB.pdb » et faire apparaître
le substrat en rouge dans l’enzyme. Que constatez-vous ?
- Réaliser un schéma simple de l’enzyme avec son substrat.
Appeler le professeur pour vérifier
Q 2) Le site actif de la CPA
- Fermer les deux molécules et reouvrir la molécule CPA avec son substrat, choisir la représentation en « fil
de fer », en fond blanc (changer le fond dans la palette couleur).
- Faire apparaître en représentation en « sphères » : en rouge le substrat, en vert les acides aminés du site
catalytique et en bleu les acides aminés du site de reconnaissance qui représentent le site actif de l’enzyme.
- Faire le même travail avec la CPA seule.
Appeler le professeur pour vérifier
- Zoomer sur le site actif des deux molécules. Que constatez vous et comment expliquez vous la fonction du
site actif de l’enzyme ?
Q 3) Le site actif de l’enzyme muté
- Ouvrir la molécule « mut_cpasub.pdb », faire apparaître avec le même code couleur le site le site actif de
l’enzyme.
- Comparer la CPA normal et la CPA mutée et proposer une explication à l’inactivité de la CPA mutée.
Q 4) Les allèles du gène de la CPA
A l’aide du logiciel Anagène, ouvrir le fichier « 2carboxypeptidases.edi » et comparer les séquences de la
CPA normale (pris comme référence) et de la CPA mutée.
Appeler le professeur pour vérifier
BILAN : A partir des différentes comparaisons des molécules, répondre à la problématique.

Info carboxypeptidase
Structure de la carboxypeptidase A (ou α) bovine.
Enzyme hydrolase (C-TERMINAL PEPTIDASE), c'est à dire qu'elle hydrolyse les chaînes polypeptidiques à partir de
leur extrémité C-terminale (celle qui porte la fonction acide carboxylique).
La carboxypeptidase est produite par le pancréas sous la forme de procarboxypeptidase, complexée avec le
chymotrypsinogène et la proprotéinase E. Ce complexe est appelé zymogène.
Ce modèle présenté ici comporte l'enzyme avec son substrat : un dipeptide Gly1 Tyr2. L'enzyme a une forme
globuleuse. L'affichage en rubans révèle une structure complexe comprenant des hélices alpha, des feuillets bêta et
des coudes. La structure spatiale est maintenue par des liaisons hydrogène (liaisons faibles) et des ponts disulfure
(liaisons covalentes).
Le site actif possède un hétéro-atome de zinc et il met en jeu 6 résidus : His69, Glu72, His196, Arg145, Tyr248 et
Glu270. Parmi les six résidus qui interviennent dans la catalyse, on distingue :
Trois résidus, His69, Glu72, His196 appartiennent au site catalytique au sens strict,
Trois résidus, Arg145, Tyr248 et Glu270 jouent le rôle de site de fixation du substrat.
Tous les acides aminés du site catalytique de la carboxypeptidase sont linéairement très espacés dans la séquence
primaire de la protéine ( n° d'ordre dans la séquence primaire étant : 69, 72, 145, 196, 248 et 270). Pour autant, tous
ces acides aminés sont disposés à proximité les uns des autres, autour du site catalytique, dans la structure spatiale.
La conformation spatiale de la carboxypeptidase est "adaptée" à la fonction de l'enzyme.
Ces 6 résidus (69, 72, 145, 196, 248 et 270) sont disposés à proximité les uns des autres, autour du site catalytique,
autour du substrat (en rouge) et du zinc (en mauve) dans la structure spatiale.
D’après le document 1, la conformation d’une enzyme est sa structure particulière tridimensionnelle. Cette conformation est due à
des ponts de disulfures soit des liaisons chimiques permettant de relier 2 points de la chaine polypeptidique de la protéine.
On en déduit que c’est grâce cette conformation que chaque enzyme a une spécificité de substrat et qu’elle peut jouer son rôle de
catalyseur.
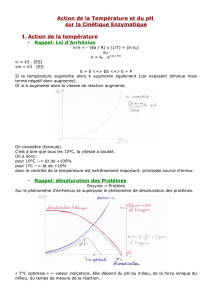
Le document 2 nous montre les effets de la dénaturation de l’enzyme sur son activité. La dénaturation est la modification et la
perte de la conformation de la protéine. On remarque que l’élévation de la température, la modification du PH ou encore
l’utilisation d’acide, rompent certains pont de disulfures. La conformation de la protéine se retrouve modifiée, elle est dénaturée.
On remarque sur le graphique représentant l’impact de la dénaturation d’un enzyme sur son activité que plus il y a de groupe SH
détruit, plus l’activité enzymatique est faible.
La conformation de l’enzyme permet à cette dernière de s’associer à un substrat spécifique pour former un complexe enzyme-
substrat. Plus la dénaturation détruit de groupe SH, plus la conformation de la protéine change. Or lors de la modification de la
conformation de la protéine, le site actif qui a une forme complémentaire aux substrats se retrouvent également modifié et ne peux
plus former un complexe enzyme substrat. L’activité catalytique de l’enzyme se retrouve ainsi moins productive jusqu’à presque
nulle si l’enzyme se retrouve beaucoup trop différente. Par conséquent, l’activité catalytique de l’enzyme est liée à sa conformation.

Matériel disponible et protocole d'utilisation du matériel
Ressource complémentaire :
On travaille sur la structure 3D d’une enzyme, la carboxypeptidase (CPA). La carboxypeptidase est une enzyme digestive pancréatique dont la fonction consiste à hydrolyser certaines liaisons
peptidiques en se fixant préférentiellement sur les acides aminés terminaux présentant une chaîne latérale très volumineuse (ici la tyrosine). Par exemple, avec un substrat dipeptide formé de
seulement deux acides aminés : carboxypeptidase
Glycine-Tyrosine Glycine + Tyrosine
Chez certains individus, la CPA est inactive.
Dans l’état actuel des connaissances on sait que certains acides aminés du site actif sont indispensables au fonctionnement de l’enzyme. Le site actif comprend :
Un site catalytique, site d’action de la CPA impliqué dans l’hydrolyse de la liaison peptidique et situé à proximité de la zone de reconnaissance. Il est formé de deux histidines
(His 196 et His 69), d’un acide glutamique (Glu 72), d’une molécule d’eau et au centre d’un atome de zinc (Zn2+).
Un site de reconnaissance, petite poche située près de la surface, constitué de trois acides aminés, arginine 145 (Arg145), tyrosine 248 (Tyr248) et acide glutamique (Glu270).
Remarque : Pour faire apparaître une molécule, un atome, tapez dans la fenêtre « expression AbC » la nomenclature de l’atome ou de la molécules correspondant.
Exemple : taper « 72 » pour faire apparaître l’acide aminé glutamique dans la molécule.
Matériel
- logiciel de modélisation moléculaire RASTOP (fichier .pdb) et sa fiche technique
- logiciel de traitement de séquences ANAGèNE (fichier .edi) et sa fiche technique
- fichiers de modélisation moléculaire de différents complexes :
- «CPASEUL.pdb» : fichier de l’enzyme carboxypeptidase isolée, seule sans
son substrat.
- «CPASUB.pdb » : fichier de l’enzyme carboxypeptidase liée à son substrat
(un dipeptide Glycine-Tyrosine de nomenclature Gly1 et Tyr2).
- «mut_cpasub.pdb» : fichier de l’enzyme carboxypeptidase mutée avec son
substrat.
- fichier «2carboxypeptidase.edi» : séquences d’acides aminés de deux allèles du gène
de la carboxypeptidase, l’allèle d’une CPA active et l’allèle d’une CPA non
fonctionnelle.
Afin de comprendre la formation d’un complexe « ES » et comment la double
spécificité de l’enzyme ?
avec le logiciel de modélisation moléculaire RASTOP :
- traiter les modèles moléculaires de la CPA seule et liée à son substrat afin de
mettre en valeur :
l’enzyme ;
le substrat ;
les acides aminés du site actif ;
les acides aminés du site de reconnaissance.
avec le logiciel de traitement de séquences ANAGèNE :
- comparer les séquences des chaines d’acides aminés des deux allèles du gène de la
CPA.
1
/
3
100%