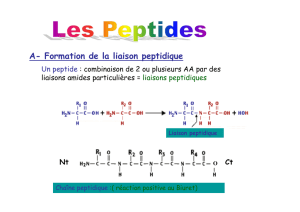
La liaison peptidique

Peptides
I- Définition, importance.
Un peptide démarre lorsquʼon a au moins 2 aa reliés par une liaison peptidique, quand il
est engagé dans une liaison on lʼappelle un résidus. Parmis ces peptides :
- hormones (insuline, glucagon, ACTH...)
- médicaments antibiotiques (valinomycine, gramicidine A)
- médicaments antitumoraux (bléomycine)
La liaison peptidique
On a un peptide qui comporte n aa qui sont reliés par la fonction carboxylique et amine.
!"#$%"%&'(#)*)+%,%-.*#
NH-CH-CO-NH-CH-CO-
R1
R2
D
D
N-terminal
C-terminal
!"#$%&'$(&)*+,-*.*/& 0&
On a donc un N et une fonction carboxylique en bout de chaine.
Convention et sens de lecture :
Convention et Sens de lecture
acide aminé N-terminal
(fonction amine libre)
acide aminé C-terminal
(fonction carboxylique libre)
Sens de lecture
!"#$%&'$(&)*+,-*.*/& 0&
II- Structure primaire
1- Détermination du nombre dʼaa
2- Détermination de la structure chimique
3- Détermination de lʼordre dʼenchainement des aa
Modification de la structure primaire :
Ces mutations de la structure primaires sont déclenchées par des mutations du code
génétique ce qui provoquera la substitution dʼun aa (arret du peptide), ce qui provoquera
une diminution ou une perte de lʼactivité physiologique. (maladies)
Techniques de séparation, qui vont permettre dʼanalyser le peptide :
Applicables aux peptides de faible poids moléculaire, et aux aa.
- Chromatographies

- Electrophorèses
Détermination de la structure primaire :
Les méthodes :
- Hydrolyse acide
- Hydrolyse enzymatique
- Combinaison hydrolyse acide et enzymatique
1- Hydrolyse acide dʼun peptide
On va partir dʼun milieu acide (HCl 6N) à une température élevée (110°C) dans des tubes
scellés pour couper les liaisons peptidiques.
Conséquences :
- Rupture de la liaison peptidique
- Destruction complète de Trp, Cys et Cystine
- Désamidation de Gln => Glu
- Désamidation de Asn => Asp
Après lʼhydrolyse on fait une analyse par chromatographie, ce qui permet de séparer tous
les aa isolés. Ce qui permet dʼavoir une idée de la composition (mais aucun sur la
séquence) du peptide.
Coupure sélective : voie chimique
Le bromure de cyanigène (BrCN), on a une coupure à droite de la méthionine, et à gauche
on a la présence dʼune homosérine lactone.
On peut aussi utiliser lʼacide de 2-nitro-5-thiocyanobenzoique, qui coupe à droite de la
cystéine.
Ou encore lʼhydroxylamine qui coupe les liaisons Asn-Gly.
Détermination de la séquence : (cʼest le code génétique qui impose la séquence)
Quatre méthodes :
- Méthode de Sanger
- Méthode dʼEdman
- Méthodes enzymatiques
- Analyse de lʼADN
2- Méthode de Sanger
Frédérick Sanger a obtenu le Noble en 1958 pour la détermination de la structure de
lʼinsuline et en 1980 pour la séquence dʼun acide nucléique.
Sa méthode :
- Marquage des fragments par lʼaction du 1-fluoro-2,4 dinitrobenzène qui va se fixer sur
lʼacide N-terminal
- Hydrolyse enzymatique de la chaine polypeptidique
- Isolement du dérivé dʼinitrobenzylé et dʼautres fragments par chromatographie
Ce qui permet lʼidentification de lʼacide aminé N-terminal.
Schéma de la méthode de Sanger

Schéma de la méthode de Sanger
DBNF
DBNF
F
NO2
NO2
NH
NO2
NO2
CH
R1
C NH CH C
O
O
R2
DNB-peptide
Peptide
Hydrolyse acide
Chromatographie
Identification
NH
NO2
NO2
CH
R1
C
O
OH
DNBF
!"#$%&'$(&)*+,-*.*/& (0&
3- Méthode dʼEdman
- Marquage des acides aminés N-terminaux
- Enlèvement séquentiel des acides aminés
- Identification des acides aminés marqués
Réactif : Phényl iso-thiocyanate (PiTC)
5pDFWLRQG¶(GPDQ
NH
CS
NH
CH C NH CH C
O
O
R2
R1
NH2CH C
O
R2
N
N
O
S
H
R1
N
CS
Peptide (n-1)
Peptide (n)
Dérivé- PTH PiTC
1° cycle
2° cycle
Hydrolyse non acide
!"#$%&'$(&)*+,-*.*/& (0&
Méthode utilisée pour des peptides petits.
4- Méthodes enzymatiques
- Masse peptidique peu importante
- Très utilisées

Méthodes enzymatiques
Masse peptidique peu importante
Très utilisées
NH2 COOH
Aminopeptidases Carboxypeptidases Exopeptidases
Endopeptidases
!"#$%&'$(&)*+,-*.*/& (0&
Groupes dʼenzymes qui vont venir couper la chaine peptidique.
Sites de coupure :
Sites de coupure
Leucine amino peptidase: toutes liaisons peptidiques
sauf celles engageant la proline
Trypsine: peptide----Lys----peptide
peptide----Arg----peptide
Chymotrypsine: peptide----Tyr-----peptide
peptide----Phe----peptide
peptide----Trp----peptide
peptide----Met----peptide
peptide----Leu----peptide
peptide----Asn----peptide
peptide----Gln----peptide
!"#$%&'$(&)*+,-*.*/& (0&
5- Analyse de lʼADN
Cette technique est lʼanalyse du gène codant pour le peptide recherché. Lʼinconvénient
cʼest la présence dʼintrons ne codant pas pour le peptides recherché.
Rupture des ponts disulfures :
Rupture des ponts disulfures
N N
S
HO
N N
S
H
O
N N
S
HO
N N
S
H
O
S
SC H2C H2O H
C H2C H2O H
N N
S
H
O
C O O -
I
N N
S
H
O
C O O -
H+I
2(SH C H2C H2O H)
+
Deux cystéines
Résidu cystine
+ +
Iodoacétate
Résidu
S-carboxyméthylcystéine
!"#$%&'$(&)*+,-*.*/& 0(&
Les Cystéine vont faire des ponts de sulfure qui va faire une boucle au niveau du peptide.

Le mercaptoéthanol va couper les ponts disulfure, mais les S-S vont refaire un pont
disulfure. On utilise donc lʼiodoacétate qui va se combiner avec le soufre, on aura un
résidu S-carboxyméthylcystéine, ce soufre ne peut plus refaire des ponts disulfures.
Exemple de détermination de la séquence :
- Utilisation du bromure de cyanogène
Exemple de détermination de la séquence
1) Utilisation du bromure de cyanogène
$QDO\VHG¶(GPDQ
!"#$%&'$(&)*+,-*.*/& 00&
On a un dipeptide qui va etre analyser par la méthode dʼEdman.
- Méthodes enzymatiques
Détermination de la séquence
2) Méthodes enzymatiques
!"#$%&'$(&)*+,-*.*/& 01&
A l a -C O O H
A l a -C O O H
A l a -C O O H
A l a -C O O H
-A la - C O O H
-A la - C O O H
 6
6
 7
7
1
/
7
100%