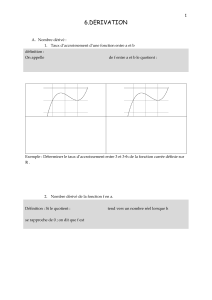
12parenté1

http://lewebpedagogique.com/bouchaud
1
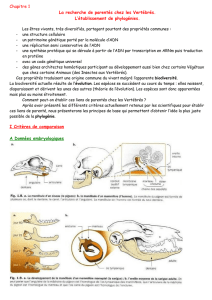
I.2 Parenté entre êtres vivants…
1/3. Etablissement de phylogénie
L’ADN, la réplication, la transcription, la traduction, le code génétique sont des mécanismes communs au sein de la cellule, unité
de base du monde vivant. Cela suppose l’existence d’une filiation et donc d’un ancêtre commun. Ainsi, il existe des relations de
parenté entre les êtres vivants. On cherche ici à établir des relations de parenté à partir de quelques exemples.
Capacités
Activité 1. La notion d’homologie
Comment établir des relations de parenté ? Livre pages 20 et 21. Répondre aux trois questions page 21.
Activité 2. Établissement d’un arbre phylogénétique
Important, il faut
comprendre les notions :
- d’homologie
- d’état primitif (ancestral)
- d’état dérivé
- groupe extérieur
- noeud
- ancêtre commun
hypothétique
- principe de parcimonie
Utiliser un logiciel
(Anagène / Phylogène) :
- prise en main du logiciel
- utilisation des
fonctionnalités du logiciel
- obtention des résultats
demandés
- rangement en fin de
séance
Quelques principes pour la construction d’arbres : lire les documents 1, 2 page 22 et 6 page 23.
Parmi les Vertébrés, on cherche à identifier les liens de parenté entre cinq espèces : pigeon, homme,
grenouille, sardine et chauve-souris (oreillard).
1. Ouvrir le logiciel phylogène puis « collection Vertébrés lycée » puis « construire ».
2. Sélectionner les animaux suivants : sardine, pigeon, oreillard, homme et grenouille.
3. Prendre les caractères : ailes, allaitement, amnios, pièces basales, placenta, squelette et plumes (tous
sont des caractères homologues).
4. Compléter le tableau des caractères à l’écran puis vérifier l’absence d’erreur. Recopier le tableau en
notant l’état des caractères dans l’échantillon : 0 pour l’état primitif et 1 pour l’état dérivé (par
convention).
5. Sélectionner le menu arbre. Construire l’arbre, c’est-à-dire les relations les plus logiques entre ces êtres
vivants. Le meilleur arbre doit posséder le moins d’innovations possibles. Pour construire l’arbre, voir la
fiche technique. Un conseil : sélectionner caractère par caractère pour construire l’arbre (les couleurs jaune
= état dérivé et bleu = état ancestral doivent vous aider). L’arbre doit être raciné par l’espèce possédant le
plus de caractères ancestraux.
6. Recopier l’arbre obtenu et y faire figurer les différentes innovations génétiques (traits + nom).
7. Placer les caractères précédents sur les deux arbres proposés ci-dessous et choisir le meilleur arbre entre
les trois en justifiant (recopier les arbres pour plus de lisibilité).
8. Déterminer si l’ensemble des caractères utilisés permet d’établir des liens de parenté entre les espèces
étudiées. Dans le cas contraire, indiquer le ou les caractères inutile(s).
9. Interpréter la présence simultanée d’ailes chez le pigeon et l’oreillard.
10. Identifier les caractères que devait avoir l’ancêtre commun à la chauve-souris et à l’homme.
Activité 3. Utilisation des caractères moléculaires.
Alors que, pendant des siècles, les relations de parenté entre les êtres vivants n’ont pu être recherchées qu’à partir de données
morphologiques, anatomiques, embryologiques ou paléontologiques, depuis une trentaine d’années, l’étude comparée des molécules
(protéines et ADN) est devenue un outil pour l’étude de l’évolution. On étudie des molécules qualifiées d’homologues.
1. Ouvrir le logiciel phylogène, puis « Vertébrés lycée » et sélectionner Fichier, Ouvrir, Tableau de séquences.
2. Sélectionner Parentés entre organismes, Vertébrés, HBA-vertébrés.aln.
Le logiciel vous présente un tableau des espèces avec les séquences d’acides aminés du polypeptide « chaîne alpha de
l’hémoglobine ». La chaîne alpha est une des chaînes constituant l’hémoglobine (avec la chaîne bêta).
- Les acides aminés sont représentés dans le code à une lettre ;
- Les nombres indiquent la position des acides aminés dans la séquence ;
- La première séquence de la liste, affichée en rouge, sert de référence ;
- Dans les autres séquences, les acides aminés différents sont affichés en noir ;
- Les tirets (-) indiquent l’absence d’un acide aminé dans une séquence.
3. Ne sélectionner que les espèces suivantes : pigeon, homme, chat, triton, thon, rorqual.
Le logiciel détermine lui-même les différences entre les séquences : il les affiche dans un tableau appelé « matrice des distances ».
4. Afficher et recopier ce tableau pour en avoir une trace sur votre feuille.
On cherche ensuite à construire l’arbre qui établit les liens de parenté entre les espèces sélectionnées. Le principe est le suivant : plus
il y a de différences entre les protéines, plus les espèces sont éloignées. Le logiciel construit seul l’arbre.
5. Sélectionner « arbre » et recopier l’arbre proposé (là encore pour en avoir une trace).
6. Faire le même travail avec la molécule de chaîne bêta « HBB-vertébrés.aln ».
7. Comparer les deux arbres construits et expliquer vos résultats.
Synthèse finale. En quelques lignes, expliquer comment on peut établir des relations de parenté entre les êtres vivants.
Mots clés : homologie ; état ancestral, état dérivé ; parenté ; ancêtre commun ; arbres phylogénétiques.
© logiciel Phylogene
(inrp)

http://lewebpedagogique.com/bouchaud
2
Fiche technique d'utilisation de PHYLOGENE
1 – Construire une matrice de
caractères
Exemple de matrice de caractères
3 – Établir des parentés
Déplacer les branches de
l’arbre
- Ouvrir un fichier image.
- Cliquer sur construire.
- Sélectionner les espèces en
cliquant sur chacune d’elles. Un
nouveau clic sur la figure
enlève l’espèce de la sélection.
- Choisir les caractères dans le
menu déroulant.
- Remplir le tableau en cliquant
dans chacune des cases et en
utilisant les informations qui
apparaissent en bas, à droite de
l’écran.
- Vérifier votre tableau.
- Corriger si nécessaire.
2. Polariser et organiser le
tableau
Exemple
- Polariser consiste à distinguer
l'état primitif et le ou les états
dérivés.
- Rechercher un taxon
extragroupe. Ce taxon ne fait
pas partie du groupe, il ne
partage donc aucun caractère
dérivé avec les membres du
groupe étudié.
- Les états primitifs des
caractères (dans le cadre de
l'étude) ont été colorés avec le
style qui a été choisi pour cela
(par défaut cadre et texte en
bleu et fond blanc).
- Choisir de colorer de la même
manière tous les états identiques
(pour un caractère donné).
- Cliquer sur Établir des parentés.
- L'icône de choix des taxons doit
être activée.
- Au départ aucun taxon n'est actif.
Il faut en choisir deux qui possèdent
au moins un état dérivé commun et
les activer en cliquant sur leurs
noms dans le tableau.
- Activer un caractère dont un état
dérivé est partagé par les deux
taxons.
- Relier alors ces deux taxons en
raison de leur état dérivé exclusif
(parmi les taxons sélectionnés)
partagé.
- Lorsque l'on choisit un caractère,
un code couleur apparaît : c'est le
code qui a été choisi à l'étape
polariser. Si cette étape a été omise
un code par défaut est utilisé :
* en bleu : état primitif du caractère
* en blanc : état indéterminé (?)
* en jaune puis orange...état dérivé.
- Puis choisir un autre taxon ayant
un caractère dérivé partagé avec un
des deux autres.
- S'il y a un caractère dérivé partagé
avec un seul des deux premiers
taxons, on branche le nouveau
taxon sur la branche de l'ancien.
Dans le cas contraire, on branche le
nouveau taxon sur le noeud.
Mode d’emploi + icônes pour créer l’arbre
- Cliquer sur la branche à déplacer avec le bouton
gauche de la souris.
- Sans lâcher ce bouton, déplacer la souris de façon à
venir faire le branchement là où on le souhaite (lorsque
le branchement est possible, un symbole apparaît :
croix pour un branchement simple. Doigt pointé vers le
haut pour un branchement multiple).
- Pour inverser l'ordre de deux branche : prendre la
branche à déplacer à la souris et la relâcher après celle
dont elle doit prendre la position.
Annule les opérations
réalisées (une par une).
Cette touche doit être
enfoncée pour permettre le
travail de permutation des
branches et d'affichage du
code couleur.
Permutation des branches
autour d’un nœud. Cliquer
sur cette icône puis se
placer au niveau d’un nœud
et cliquer à nouveau.
- Cliquer sur une case correspondant à un
état dérivé. Toutes les cases identiques
pour le caractère se colorent de la même
manière.
- Opérer de la même manière pour toutes
les colonnes.
- Faire vérifier en cliquant sur le bouton
correspondant.
- S'il y a un deuxième état dérivé (donc
autre que '?'), utiliser une autre couleur.
- Organiser le tableau en rangeant les
colonnes et les lignes.
- Déplacer les colonnes avec la souris en
les prenant au niveau des titres.
- Mettre le plus à gauche les caractères
qui sont à l'état dérivé chez le plus grand
nombre de taxons.
- Respecter aussi les logiques de groupes :
si un ensemble se dessine, continuer avec
les sous catégories avant de passer à un
autre groupe.
- Les lignes ont aussi été classées suivant
le même principe.
- L'extragroupe est ainsi en bas du
tableau.
Rejoint toutes les branches.
1
/
2
100%