Projet de thèse : Implication de la structure et de la

Projet de thèse!:
Implication de la structure et de la dynamique de la chromatine sur
l’organisation fonctionnelle des génomes
Alain Arneodo et Benjamin Audit, Laboratoire de Physique et Laboratoire Joliot-Curie, ENS-Lyon.
{Alain.Arneodo|Benjamin.Audit}@ens-lyon.fr
Objectifs de la thèse
Le projet de thèse vise à proposer une compréhension originale de l’organisation fonctionnelle du génome
basée sur son interaction avec la structure et la dynamique de la chromatine.
De nombreuses observations suggèrent qu’il existe une interaction réciproque entre la dynamique structurelle
de la chromatine et la régulation de l’expression du génome. D'une part, la structuration locale de la
chromatine conditionne l’accessibilité aux régions régulatrices des facteurs de transcription et de remodelage
de la chromatine nécessaire pour l’expression normale des gènes. D'autre part, l’expression du génome peut
conditionner cette structuration physique, dans la mesure où les polymérases peuvent jouer le rôle de
moteurs moléculaires et sont donc tout à fait susceptibles de participer au remodelage et au maintien de la
conformation de la chromatine. Ceci se traduit par le fait que chez les procaryotes comme chez les
eucaryotes, la transcription a lieu dans des régions spécifiques du nucléoïde ou du noyau. Ce couplage entre
d’une part la structuration physique du génome et d’autre part son expression est susceptible d'être recruté
comme mécanisme de régulation. La localisation chromosomique des gènes serait ainsi l’un des facteurs de
contrôle de leur expression.
Cette problématique est le sujet d'une importante activité expérimentale!; toutefois aucun modèle intégratif
définitif n’a encore été avancé [1]. Ce projet propose d’apporter des éléments de réponse à ces questions à la
lumière de l’analyse de données biologiques (séquences génomiques et leurs annotations, profils
d'expression, données sur la structuration du génome [2]). L'utilisation de concepts et de méthodes issus de la
physique développés par l’équipe d’accueil (corrélations à longue portée, transformation en ondelettes)
apportera un point de vue original sur ces données, en permettant de dissocier les différentes échelles
pertinentes pour leur analyse. Le travail de thèse consistera donc à étudier les liens entre séquence d’ADN,
réplication et transcription d’une part, et entre séquence d’ADN et organisation structurelle de la chromatine
d’autre part. La synthèse des différents résultats obtenus devrait enrichir nos connaissances sur les
interactions entre régulation du génome et dynamique structurelle de la chromatine.
Séquence et réplication
L’initiation de la réplication de l’ADN chez les eucaryotes s’effectue en de nombreuses origines réparties le
long du génome. L’activation des différentes origines de réplication est coordonnée au déroulement du cycle
cellulaire et est fonctionnellement reliée à l’état de la chromatine ainsi qu’à la chronologie de la
transcription. La connaissance de ces origines est donc essentielle à la compréhension des liens entre ces
processus. Toutefois chez les eucaryotes supérieurs, seulement une dizaine d’origines a été identifiée
expérimentalement sur les milliers théoriquement nécessaires à chaque cycle et aucun réel consensus de
séquence n’a pu être établi. Récemment, l'équipe d'accueil a montré que, chez les mammifères, la réplication
induit une asymétrie locale de composition nucléotidique entre les deux brins de la double hélice [3]. On
notera que, les mutations à l’origine de cette asymétrie n’étant transmises que si elles ont lieu dans les
lignées germinales, leur étude ne peut a priori pas fournir d’information sur des mécanismes éventuellement
spécifiques des lignées somatiques. Cette asymétrie a permis d'identifier plus de mille nouvelles origines de

réplication candidates, grâce à une stratégie multi-échelle (transformée en ondelettes) d'analyse des profils
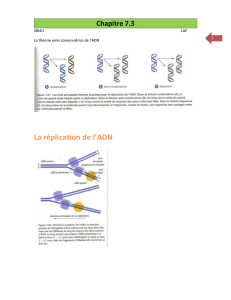
d'asymétrie mesurés le long des chromosomes (Fig.!1). Cette première approche est compatible avec un
modèle de la réplication chez les mammifères où les origines de réplication sont fixes alors que les
terminaisons ont lieu apparemment de façon aléatoire, à des sites différents à chaque cycle cellulaire (Fig.!1).
Ces premiers résultats fournissent une stratégie permettant d’obtenir une image de la dynamique de la
réplication de l'ADN à l'échelle du génome chez les eucaryotes. Se pose alors la question de savoir si
origines et terminaisons sont fixes ou flottantes chez les autres eucaryotes (animaux, plantes, protistes). On
peut s’attendre à découvrir des mécanismes différents dans les différentes branches, comme le suggère les
données de la littérature concernant la drosophile et le xénoppe, qui ne présentent pas de sites d’initiation de
la réplication fixes pendant les premières étapes du développement. L’une des premières étapes de la thèse
consistera donc à analyser l'asymétrie de composition pour l'ensemble des génomes eucaryotes
complètement séquencés afin de déterminer lorsque cela sera possible la distribution spatiale des origines
voir des terminus de réplication. Par ailleurs, les séquences d'ADN proches des loci détectés seront analysées
pour chercher des motifs communs les identifiant, comme c’est le cas avec les séquences ARS
(Autonomously Replicating Sequences) pour les origines de réplication chez S.!cerevisiae. L'existence de tels
motifs permettrait de détecter les origines qui ne sont pas actives dans la lignée germinale.
Figure 1 : Asymétrie de composition S le long d'un fragment du chromosome 20 de l'homme. S=(T-A)/(T+A)+(G-C)/(G+C) est
calculée dans des fenêtres de 1 kpb. Les couleurs rouge, bleu ou noir marquent les gènes sens, les gènes anti-sens et les zones
intergéniques, respectivement. La ligne pleine verticale pointe la position de l'origine de réplication TOP1. Les lignes pointillées
correspondent à deux origines de réplication putatives, prédites par la présence d'un saut ascendant de l'asymétrie. Le profil
linéairement décroissant et l'absence de saut descendant entre deux origines suggèrent que la terminaison de la réplication ne
s'effectue pas au même locus d'un cycle de réplication à l'autre. Noter que les gènes sens (anti-sens) tendent à se placer à droite (à
gauche) des origines.
Séquence, transcription et domaines d’expression
Dans la deuxième étape du projet, nous étudierons la relation entre la distribution des gènes le long des
chromosomes et leur transcription. En particulier nous chercherons s’il existe une règle de localisation
correspondant à des domaines d’expression, en étudiant la distribution spatiale de l’activité des ARN-
polymérases. Cette activité sera estimée par deux approches complémentaires. Tout d’abord, nous
exploiterons le fait que la transcription induit, comme la réplication, une asymétrie de composition entre les
deux brins. En utilisant les résultats de la première partie du projet, on pourra s'affranchir de la contribution
de la réplication à l'asymétrie de composition des gènes. Puis, en utilisant des données de puces à ARN
disponibles dans les bases de données, nous vérifierons dans quelle mesure cette asymétrie reflète bien
l’activité moyenne des ARN-polymérases le long des gènes. Cette approche, une fois mise au point sur le

génome humain, sera généralisable à un grand nombre de génomes, quelle que soit la qualité de l’annotation
ou des données d’expression les concernant. En second lieu, nous utiliserons les données d’expression afin
d’appréhender la dynamique de la transcription, puisqu’elles permettent une estimation directe de l'activité
des ARN-polymérases. On analysera alors les variations temporelles ou inter-tissulaires des profils
d'expression et des éventuels domaines d'expression.
Séquence et domaines structuraux
En s'appuyant sur les travaux précédents de l'équipe d'accueil qui ont montré que les séquences d’ADN
induisent une succession particulière des sites de courbure/flexibilité du polymère d’ADN [4] et que cette
écriture structurale influence très probablement la dynamique de compaction de l'ADN [5], il s'agira de
délimiter des domaines structuraux au sein de la molécule d’ADN. Ce travail sera facilité par le
développement récent d'expérimentations fournissant des informations relatives à la structuration du génome
à l'échelle génomique [2].
Pour l'ensemble des domaines qui auront pu être délimités (réplicons, domaines d'expression, domaines
structuraux), il sera particulièrement informatif d'analyser leur degré de superposition et de savoir s’ils
correspondent à des régions de densité et/ou co-orientation significatives des gènes. Enfin, il s’agira de
comprendre comment la fonction des gènes et leurs interactions se projettent sur le génome en relation avec
les domaines. En effet, le réseau des interactions forme un graphe complexe sans échelle caractéristique.
Cette propriété ne s’accommode pas naturellement avec l’agencement séquentiel des gènes le long des
chromosomes. Par exemple, il sera intéressant de savoir si les gènes les plus importants (les plus connectés
au sein du graphe) ont une distribution particulière. De même, si la coordination de la transcription est
couplée à la structure et à la dynamique de la chromatine alors certaines associations fonctionnelles de gènes
devraient favoriser la localisation de ceux-ci dans des domaines d’expression/structuraux coexistants. Nous
nous emploierons donc à étudier si certaines classes fonctionnelles de gènes possèdent des motifs de
localisation spécifiques.
Environnement
Ce projet de thèse se fera à l'interface des différentes équipes expérimentales du Laboratoire Joliot-Curie qui
par l’intermédiaire d’expériences de biologie moléculaire et de micro-manipulation de molécule unique
(microscopie par force atomique, résonance plasmon, pinces magnétiques, microscopie de fluorescence)
s’intéressent aux problèmes de la structuration de l’ADN en relation avec la réplication et la transcription. Il
sera développé en étroite collaboration avec le groupe de biologistes dirigé par C.!Thermes au Centre de
Génétique Moléculaire à Gif/Yvette.
1 - T. Cremer et C. Cremer. Chromosome territories, nuclear architecture and gene regulation in mammalian cells.
Nature Reviews Genetics 2, 292-301 (2001).
2 - G.-C. Yuan et al. Genome-scale identification of nucleosome positions in S. cerevisiae. Science 309, 626-630
(2005).
3 - M. Touchon et al. Replication-associated strand asymmetries in mammalian genomes: Toward detection of
replication origins. Proceedings of the National Academy of Sciences USA 102, 9836–9841 (2005)
4 - B. Audit et al. Long-range correlations between DNA bending sites: Relation to the structure and dynamics of
nucleosomes. Journal of Molecular Biology 316, 903-918 (2002).
5 - C. Vaillant, B. Audit & A. Arneodo. Thermodynamics of DNA loops with long-range correlated structural disorder.
Physical Review Letters 95, 068101 (2005).
1
/
3
100%