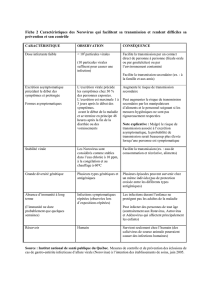
revue - John Libbey Eurotext

revue
Les calicivirus humains,
une cause majeure de gastro-entérite aiguë
E. Kohli
F. Bon
K. Balay
P. Pothier
Laboratoire de virologie, CNR des virus
entériques, Hôpital du Bocage,
21079 Dijon Cedex
Microbiologie médicale et moléculaire,
UFR Médecine et Pharmacie,
21079 Dijon Cedex
Résumé.Les calicivirus humains (HuCV) appartiennent aux genres Norovirus
et Sapovirus de la famille des Caliciviridae. Ils sont responsables de gastro-
entérites aiguës. Ces virus à ARN simple brin présentent une grande variabilité
génétique rendant leur détection complexe. En effet, 7 génogroupes distincts
incluant au moins 34 génotypes humains sont actuellement reconnus à l’intérieur
de ces deux genres. Par ailleurs, des souches recombinantes de norovirus ainsi
que des variants à l’intérieur d’un même génotype ont été récemment décrits,
ajoutant encore à cette diversité. Grâce à l’amélioration des outils diagnostiques,
les norovirus sont maintenant reconnus dans les pays industrialisés comme des
agents majeurs de gastro-entérite aiguë sporadique et épidémique dans toutes les
tranches d’âge, à l’origine d’une morbidité et de dépenses importantes. La
prévalence des HuCV dans les pays en voie de développement reste à détermi-
ner. La biologie de ces virus, jusque-là mal connue du fait de l’impossibilité de
les cultiver et de l’absence de modèle animal, connaît actuellement d’importants
développements dans le domaine des norovirus avec, d’une part la description de
facteurs génétiques de sensibilité et, d’autre part, la découverte d’un norovirus
murin cultivable et pouvant servir de modèle. Ces progrès pourraient aider au
contrôle des infections à HuCV.
Mots clés :Caliciviridae,Norovirus,Sapovirus, gastro-entérite aiguë
Abstract.Human caliciviruses (HuCVs) are grouped into two genera, Norovi-
rus and Sapovirus within the Caliciviridae family. These single-stranded RNA
viruses show a great genetic diversity making their detection difficult. Indeed, 7
genogroups including at least 34 human genotypes are now recognized within
these 2 genera. In addition, norovirus recombinant strains as well as variants
within a genotype have been shown recently to generate further diversity. Recent
advances in molecular diagnostic techniques have allowed to recognize norovi-
ruses as a major cause of sporadic and outbreak gastroenteritis in all age groups,
leading to an important morbidity and high costs in industrialized countries.
Their prevalence in developing countries remains to be determined. Until now,
the biology of these viruses has been difficult to study because of the lack of
either cell culture or animal model systems that support their propagation. New
insights into the biology of noroviruses were recently provided with the descrip-
tion of genetic host susceptibility factors as well as the discovery of a cultivable
murine norovirus which should provide a new model for human noroviruses.
These improvements could help to the control of HuCV infections.
Key words:Caliciviridae,Norovirus,Sapovirus, acute gastroenteritis
Tirés à part : E. Kohli
Virologie 2005, 9 : 93-106
Virologie, Vol. 9, n° 2, mars-avril 2005
93
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 03/06/2017.

Les calicivirus humains (HuCV) appartiennent à la famille
des Caliciviridae. Cette famille comprend quatre genres
dont deux incluent essentiellement des virus pathogènes
pour l’homme, les genres Norovirus (anciennement
Norwalk-like virus)etSapovirus (anciennement Sapporo-
like virus) et deux, les genres Vesivirus et Lagovirus, infec-
tent les animaux. Si les calicivirus animaux sont à l’origine
de syndromes très variés, maladie hémorragique fatale chez
le lapin (rabbit hemorrhagic disease virus ou RHDV),
syndromes respiratoires chez le chat (feline calicivirus ou
FCV) ou encore avortements et lésions vésiculaires chez
l’otarie (San Miguel Sea lion virus ou SMSV), les calici-
virus humains sont, eux, responsables de gastro-entérites
aiguës. Grâce à l’amélioration des outils diagnostiques, des
progrès importants ont été réalisés ces dernières années
dans la connaissance de l’épidémiologie des HuCV, les-
quels sont actuellement reconnus comme des agents étio-
logiques majeurs de gastro-entérite épidémique et sporadi-
que dans toutes les tranches d’âge. Du fait de l’absence de
système de culture in vitro et de modèle animal de labora-
toire, la biologie de ces virus est encore mal connue. Tou-
tefois, d’importants développements sont en cours dans le
domaine de la biologie des norovirus. Ces développements
font suite à la découverte de l’importance des oligosaccha-
rides de la famille des antigènes tissulaires de groupe san-
guin comme facteurs génétiques de sensibilité à l’infection
(cet aspect a fait l’objet d’une revue récente par N. Ruvoën-
Clouet dans Virologie [1]). Par ailleurs, la description d’un
norovirus murin cultivable et pouvant servir de modèle
animal [2] ouvre également d’intéressantes perspectives.
Historique
Le virus Norwalk, prototype des norovirus, a été identifié
pour la première fois en 1972 par immunomicroscopie
électronique par Kapikian et al. après passage chez des
volontaires d’un filtrat de selles provenant d’un patient
infecté lors d’une épidémie survenue dans une école à
Norwalk (Ohio) en 1968 [3]. Ce virus, non enveloppé, d’un
diamètre de 27 nm, est le premier virus de gastro-entérite
identifié. À la fin des années 1970, Madeley et Cosgrove
puis Flewett et Davies et enfin Chiba et al. rapportèrent la
présence, dans des selles d’enfants et de nourrissons, de
calicivirus « typiques » similaires aux calicivirus animaux
caractérisés auparavant. Un tel virus, détecté dans une
institution pour enfants à Sapporo et antigéniquement dis-
tinct du virus Norwalk, a par la suite été désigné
Sapporo/82/Japan [4]. Une classification en deux groupes
fondée sur la morphologie en microscopie électronique a
alors été proposée, incluant d’une part les calicivirus clas-
siques représentés par le virus Sapporo et d’autre part les
SRSV (small round structured viruses) représentés par le
virus Norwalk. Les calicivirus classiques possèdent à leur
surface des dépressions en forme de coupe ou calice (d’où
leur nom) alors que les SRSV ont des dépressions moins
prononcées leur donnant une surface moins typique
(figure 1).
La description du génome du virus Norwalk par Jiang et al.
en 1990 [5] a représenté une étape importante dans la
connaissance de ces virus non cultivables et a ouvert la voie
à leur diagnostic moléculaire. Elle a permis de reconnaître
que le virus Norwalk était le représentant d’un groupe très
varié de virus pouvant être divisés en deux sous-groupes
phylogénétiques majeurs, les génogroupes I (GI, virus
Norwalk) et II (GII, virus Hawaii, Snow Mountain agent).
Par ailleurs, le clonage du virus Sapporo/82/Japan a permis
de reconnaître deux genres différents parmi les HuCV, les
genres Norovirus et Sapovirus, et, ainsi, de valider la clas-
sification en deux groupes fondée sur la morphologie
50nm
50nm
Figure 1. Virus Norwalk (Norovirus, à gauche) et Sapporo (Sapovirus, à droite) observés en microscopie électronique
(www.virology.net/Big_Virology/BVRNAcalici.html, échelle = 50 nm).
revue
Virologie, Vol. 9, n° 2, mars-avril 2005
94
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 03/06/2017.

(figure 2). Depuis, de nombreuses souches ont été décrites à
l’intérieur de ces deux genres, parmi lesquelles des souches
détectées chez les animaux.
Organisation génomique
Le génome des HuCV est constitué d’un ARN simple brin
positif polyadénylé comprenant environ 7 500 bases
(figure 3) [4]. Un motif GU présent à l’extrémité 5’ est aussi
retrouvé à l’intérieur du génome de tous les calicivirus et
correspond probablement au début d’un ARN subgénomi-
que. Un tel ARN a en effet été observé dans les cellules
infectées par le FCV et le RHDV. Le génome des HuCV
présente deux types d’organisation selon que les gènes des
protéines non structurales (parmi lesquelles l’hélicase, la
protéase et la polymérase) et de la protéine majeure de la
capside (VP1) sont dans un ou deux cadres ouverts de
lecture ou ORF (cas des sapovirus et des norovirus respec-
tivement). Dans les deux types d’organisation, un petit
ORF (ORF2 ou ORF3) code une protéine structurale mi-
neure basique désignée VP2. Il est à noter que, pour la
plupart des souches de sapovirus décrites, un troisième
ORF de fonction inconnue, chevauchant la région 5’ du
gène de la capside, a été identifié. L’extrémité 5’ est liée à
une protéineVPg qui pourrait jouer un rôle dans l’induction
de la synthèse protéique.
Structure
La capside du virus Norwalk (norovirus, génogroupe I) est
composée d’une protéine majeure de 58 kDa, VP1 et de
quelques copies d’une seconde protéine basique, VP2. La
structure atomique de particules recombinantes ou VLP
(virus-like particles) du virus Norwalk constituées de la
protéine VP1 exprimée dans le système baculovirus a été
élucidée par Prasad et al. en cryomicroscopie électronique
et par cristallographie aux rayons X à 3,4 Å [6]. La capside
ainsi obtenue est constituée de 90 dimères de la protéine
Calicivirus canins
Calicivirus félins
SMSV - VESV
Parkville
Sapporo
London
Sapovirus
RHDV
EBHSV
Lagovirus
α1
Norovirus
Lordsdale
Toronto
Hawaii
Melksham
Desert Shield Southampton
Norwalk
Vesivirus
Figure 2. Analyse phylogénétique de la capside de quelques souches représentatives des calicivirus (d’après
www.iah.bbsrc.ac.uk/virus/Caliciviridae/trees/Capsid_tree.htm).
SMSV: San Miguel sea lion virus; VESV: vesicular exanthema of swine virus; RHDV: rabbit hemorrhagic disease virus; EBHSV: European
brown hare syndrome virus.
revue
Virologie, Vol. 9, n° 2, mars-avril 2005
95
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 03/06/2017.

VP1 et présente une symétrie icosaédrique (T = 3)
(figure 4). La protéine VP1 s’organise en un domaine in-
terne relativement conservé (shell) comprenant un bras
N-terminal (N) ainsi que le domaine S (domaine intermé-
diaire) et un domaine exposé à la surface de la capside, le
domaine P (protruding). Ce dernier comporte deux sous-
domaines, P1 et P2. Le domaine P2 représente le domaine
le plus variable de la capside des norovirus et son exposi-
tion à la surface est compatible avec son rôle dans l’interac-
tion avec des récepteurs présents à la surface des cellules
épithéliales intestinales [7, 8].
L’organisation en modules S-P1-P2 retrouvée pour
d’autres calicivirus permettrait à la protéine majeure de
capside ses propriétés d’assemblage, d’interaction avec les
récepteurs et d’immunogénicité, malgré une grande varia-
bilité [9].
La protéine VP2, faiblement représentée dans la capside
virale, augmenterait la stabilité de VP1 et ainsi la protége-
rait de la dégradation, notamment par les protéases.
Variabilité génétique et antigénique,
classification
Les norovirus présentent une grande diversité génétique
(tableau 1). Les souches sont classées en génogroupes,
chaque génogroupe étant lui-même subdivisé en génotypes
sur la base de la séquence en acides aminés de la protéine de
capside VP1. La dernière classification proposée par Green
et al. en 2001 [4] est en cours de réactualisation. Actuelle-
ment, les norovirus peuvent être classés en 5 génogroupes
dont 3 comprennent des souches humaines, les génogrou-
pes I, II et IV. Zheng et al. (département des gastro-entérites
virales, CDC, Atlanta, USA) ont proposé, à la seconde
conférence internationale sur les calicivirus, 26 génotypes
humains à l’intérieur de ces 3 génogroupes (GI 1 à 8, GII 1
à 17 et GIV 1) [10]. Toutefois, cette classification n’est pas
officialisée et d’autres génotypes ont été décrits.
Des calicivirus animaux sont également classés dans le
genre Norovirus, parmi lesquels des souches bovines (gé-
nogroupe III, virus Jena et Newbury-agent 2), porcines
(génogroupe II) et une souche murine (génogroupe V).
Par ailleurs, des phénomènes de recombinaison entre sou-
ches présentant une polymérase et une capside dérivées de
souches parents différentes, générant ainsi de nouveaux
virus, ont été décrits [11, 12]. L’une de ces souches recom-
binantes, le variant GGIIb, est particulièrement remarqua-
ble. Il a été isolé pour la première fois au cours d’une
épidémie d’origine hydrique dans le sud de la France en
août 2000 [13]. Ce virus possédant une polymérase (ORF1)
ne permettant pas de le relier à une souche de référence
donnée et une capside (ORF2) appartenant au génotype
Norovirus
Sapovirus
Hel : hélicase
Pro : protéase
Pol : ARN polymérase
5'
5'
VPg
VPg
VPg
Hel
Hel
VPg
VPg
Pro
Pro
Pol
Pol
VP1
VP1
VP1
VP2
VP2
VP2
ORF 1
ORF 1
ORF 2
ORF 2
ORF ?
ORF 3
6950
6950
6855
6852 7349
3'
3'5'
5358
5374
7588
3' poly A
poly A
poly A
ARN subgénomique
7654
Figure 3. Organisation du génome des calicivirus humains. Positions sur le virus Norwalk (Genbank accession n° M87661) pour les
norovirus et Manchester (Genbank accession n° X86560) pour les sapovirus.
revue
Virologie, Vol. 9, n° 2, mars-avril 2005
96
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 03/06/2017.

Hawaii (figure 5) représente en fait un groupe de virus
ayant diffusé de façon explosive en France mais aussi dans
de nombreux pays européens au cours de l’hiver 2000-
2001. Sa diffusion a pu être observée en temps réel grâce à
un réseau de surveillance européen mis en place en 2000,
Foodborne viruses in Europe [14]. Ces virus présentent une
homologie importante au niveau du gène de la polymérase,
lequel peut être associé à 3 gènes de capside différents
(génotypes Hawaii,Mexico et Snow Mountain) [15]. Le
site de recombinaison se situerait au niveau d’une région
hautement conservée à la jonction ORF1-ORF2 [11, 12] au
niveau de laquelle l’ARN polymérase pourrait passer d’un
génome parent à l’autre au cours de la réplication virale en
cas de co-infection. Nous avons identifié trois autres sou-
ches recombinantes, suggérant que ce phénomène n’est pas
rare [16]. Cependant, ces souches ont présenté une diffu-
sion apparemment limitée, à la différence des souches
GGIIb. Il est à noter que, si la recombinaison génère de
nouveaux virus, toutes les souches recombinantes décrites
à ce jour présentent des capsides correspondant à des géno-
types déjà décrits. Ainsi, le mécanisme conférant à certai-
nes souches un avantage sélectif reste à élucider.
Enfin, en 2002, a été rapportée l’émergence dans différents
pays européens d’un « nouveau variant » à l’intérieur du
génotype Bristol-Lordsdale (GII-4), à l’origine d’une aug-
mentation importante des épidémies à norovirus au cours
du printemps et de l’été [17]. Il est probable que le même
phénomène s’est reproduit en 2004 avec une nouvelle sou-
che. Des travaux récents ont montré que ces souches nou-
velles présentaient des mutations majeures dans la protéine
VP1, notamment au niveau de la région P2 [18]. Ces muta-
tions pourraient être à l’origine d’un échappement à la
réponse immunitaire, expliquant ainsi la circulation conti-
nue de ce génotype dans la population, et aussi d’une
modification des propriétés de liaison de ces virus à leurs
récepteurs cellulaires.
Au total, ces phénomènes de recombinaison générant de
nouveaux virus ainsi que l’émergence de nouveaux variants
à l’intérieur d’un génotype donné contribuent à la variabi-
N (10-49) S (50-225) P1 (226-278) P2 (279-405) P1 (406-520)
Domaine P2Domaine P2
Domaine P1
Domaine S
N-terminal
N
N
N
C
CC A
B
C
Domaine interne (shell) Domaine P (protruding)
MonomèreDimère
Figure 4. La protéine de capside VP1 du virus Norwalk : structure d ’une pseudo-particule virale (VLP). A) reconstruction en
cryomicroscopie électronique, représentation de surface (haut) et en coupe (bas). B) structure du monomère et du dimère par
cristallographie aux rayonsXà3,4Å.C) structure primaire de VP1 (position des acides aminés). Adapté de Hutson et al. [43] avec
autorisation.
revue
Virologie, Vol. 9, n° 2, mars-avril 2005
97
Copyright © 2017 John Libbey Eurotext. Téléchargé par un robot venant de 88.99.165.207 le 03/06/2017.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
1
/
14
100%