Le talon d`Achille des bactéries

Reflexions, le site de vulgarisation de l'Université de Liège
© Université de Liège - http://reflexions.ulg.ac.be/ - 19 April 2017
- 1 -
Le talon d'Achille des bactéries
22/01/09
Avec l'apparition d'un nombre sans cesse croissant de souches bactériennes résistantes aux antibiotiques, la
recherche de nouvelles cibles pour en venir à bout est une priorité en termes de santé publique. Au Centre
d'Ingénierie des Protéines de l'ULg, Mohammed Terrak et son équipe ont mis au jour le rôle d'une série de
«motifs protéiques» d'une enzyme essentielle dans la formation de la paroi des bactéries. Cette découverte
ouvre de nouvelles pistes pour la recherche de molécules antibactériennes.
L'utilisation toujours plus large des antibiotiques pour traiter les maladies d'origine bactérienne favorise le
phénomène de résistance de ces micro-organismes. En effet, si toutes les espèces vivantes sont caractérisées
par leur capacité d'adaptation à leur environnement, les bactéries sont particulièrement performantes dans
ce domaine (lire aussi Antibiotiques contre bactéries). Au fil de leur multiplication, les bactéries mutent
et certaines acquièrent les armes génétiques nécessaires pour résister aux produits destinés à les détruire.
Les bactéries mutantes sont alors sélectionnées puisqu'elles sont les seules à survivre au traitement. De
plus, ces «élues» peuvent transmettre leur pouvoir de résistance par transfert de leur matériel génétique à
leurs congénères. Suite à cette dissémination des gènes de résistance, les traitements habituels deviennent
rapidement inefficaces pour soigner les maladies provoquées par ces bactéries. D'où l'intérêt de chercher de
nouveaux «talons d'Achille» chez ces être microscopiques.
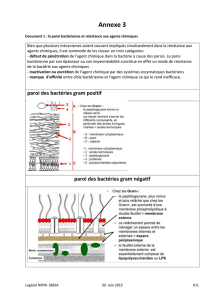
La paroi bactérienne, une armure pas infaillible
Une des particularités de la plupart des bactéries est qu'elles sont pourvues d'une paroi cellulaire
caractéristique que l'on ne retrouve pas chez les Eucaryotes. «Cette différence fondamentale représente une
aubaine en termes de lutte contre ces microbes car en visant la destruction de la paroi des bactéries, les
antibiotiques ne risquent pas de nuire aux patients», indique Mohammed Terrak, chercheur qualifié FNRS
au Centre d'Ingénierie des Protéines de l'ULg. Au vu de cette caractéristique particulière, les scientifiques
se sont donc penchés sur des antibiotiques dont le mode d'action empêcherait la formation de cette fameuse
armure.
La paroi des bactéries est composée de longues chaînes de sucres reliées entre elles par des peptides pour
former un réseau tridimensionnel rigide, tel une sorte de sac qui entoure et protège le micro-organisme. Sans
cette protection, et sous l'effet de la pression osmotique qui règne à l'intérieur de son cytoplasme, la bactérie
éclate.
Une fois synthétisés à l'intérieur de la bactérie, les précurseurs nécessaires à la construction de la paroi
traversent la membrane plasmique. Ils sont ensuite pris en charge par une protéine dont la mission est double :
fabriquer les longues chaînes de sucre et les assembler en réseau, des activités respectivement appelées
glycosyltransférase et transpeptidase.
Une protéine, deux actions, deux cibles
Cette enzyme bifonctionnelle fait partie de la famille des Penicillin-Binding Proteins (PBP). S'il en existe
différentes formes au sein du monde bactérien, toutes présentent une structure fort semblable. Comme

Reflexions, le site de vulgarisation de l'Université de Liège
© Université de Liège - http://reflexions.ulg.ac.be/ - 19 April 2017
- 2 -
l'indique leur nom, les scientifiques connaissent depuis longtemps les effets de la pénicilline sur l'activité de ces
protéines. Cet antibiotique naturel inhibe en effet l'activité transpeptidase des PBP, bloquant ainsi le pontage
des chaînes de sucres entre elles.
Depuis cette découverte, bon nombre d'autres molécules aux propriétés similaires ont été découvertes.
Mais jusqu'ici les scientifiques n'ont encore jamais mis la main sur des antibiotiques capables de s'attaquer à
la fabrication des chaînes de sucres, soit à l'activité glycosyltransférase des PBPs, et qui ne sont pas toxiques
pour l'homme. Une des raisons de cet «insuccès» réside dans le fait que la structure tridimensionnelle de ces
enzymes était jusqu'il y a peu encore inconnue.

Reflexions, le site de vulgarisation de l'Université de Liège
© Université de Liège - http://reflexions.ulg.ac.be/ - 19 April 2017
- 3 -
En 2007 cependant, deux équipes de chercheurs nord américains ont réussi à déterminer la structure 3D
de deux membres de la famille des glycosyltransferases/PBPs grâce à une technique d'analyse basée sur
la diffraction de rayons X par des cristaux de protéines. A partir de ces résultats et grâce à l'aide du Dr.
Eric Sauvage du laboratoire de Cristallographie des Protéines du Centre d'Ingénierie des Protéines de l'ULg,
Mohammed Terrak et son équipe ont pu modéliser par informatique la structure tridimensionnelle de la protéine
1b d'Escherichia coli, un autre membre de la famille des PBPs. «Dans le cadre du projet européen EUR-
INTAFAR, l'objectif de notre recherche est de comprendre le mécanisme catalytique et la fonction de ces
enzymes afin de pouvoir identifier des inhibiteurs de ces protéines», explique Mohammed Terrak.
Cinq motifs protéiques cruciaux
Le modèle informatique en trois dimensions de la protéine 1b a permis aux chercheurs de modifier un par
un les acides aminés conservés dont elle est composée. Une opération dont le but était de découvrir quels
sont les acides aminés cruciaux pour l'activité de l'enzyme. Ce travail consiste à provoquer des mutations
ponctuelles pour chaque acide aminé et à faire ensuite une analyse détaillée de la protéine ainsi modifiée.
«Nous voulions voir comment la protéine, dont un acide aminé avait été altéré, réagissait avec le substrat et si
cela empêchait l'enzyme de mener à bien sa mission de formation de la paroi», précise Mohammed Terrak. Il
a fallu environ deux à trois mois aux chercheurs pour la réalisation et l'analyse de chaque enzyme mutée. On
comprend dès lors pourquoi les résultats de cette étude, publiés récemment dans the Journal of Biological
Chemistry (1), ont mis près de deux ans et demi à voir le jour.
Le travail minutieux et la patience des scientifiques ont fini par payer : cinq motifs protéiques, c'est-à-dire des
séquences d'acides aminés, ont été identifiés comme jouant un rôle crucial pour l'activité de la protéine 1b.
Parmi ces cinq motifs, les trois premiers seraient particulièrement importants pour la fabrication des chaînes
de sucres.

Reflexions, le site de vulgarisation de l'Université de Liège
© Université de Liège - http://reflexions.ulg.ac.be/ - 19 April 2017
- 4 -
Des renforts pour les antibiotiques ?
«D'ici à trouver les molécules qui pourront interférer avec ces motifs pour inhiber l'activité de la protéine, le
processus est encore long», affirme Mohammed Terrak. Mais la recherche en ce sens se poursuit en deux
voies au Centre d'Ingénierie des Protéines.
Maintenant que la structure du site actif de l'enzyme est connue, les chercheurs tentent de modifier les
précurseurs nécessaires à la formation des chaînes de sucres afin que ceux-ci ne soient plus reconnus comme
substrats par la protéine mais qu'ils aient un effet inhibiteur sur cette dernière (dans le cadre d'un projet PAI ).
C'est ainsi que les scientifiques espèrent trouver de nouvelles molécules capables d'enrayer la mécanique
de formation de la paroi chez les bactéries. L'autre voie explorée par Mohammed Terrak et son équipe est
le criblage de librairies de molécules chimiques. Il existe en effet des listes de molécules connues que les
chercheurs peuvent tester in vitro sur la protéine 1b afin de voir si elles bloquent l'activité de cette enzyme. Si
l'une d'entre elles s'avérait être efficace, les chercheurs devraient alors déterminer la structure 3D du complexe
formé par la molécule et la protéine pour voir s'il est possible d'en faire un nouvel antibiotique.
(1) Mohammed Terrak, Eric Sauvage, Adeline Derouaux, Dominique Dehareng, Ahmed Bouhss, Eefjan Breukink, Sylvie Jeanjean and Martine Nguyen-
Distèche, Importance of the conserved residues in the peptidoglycan glycosyltransferase module of the class A penicillin-binding protein 1b of Escherichia
coli, in J. Biol. Chem., Vol. 283, Issue 42, 28464-28470, October 17, 2008.
1
/
4
100%