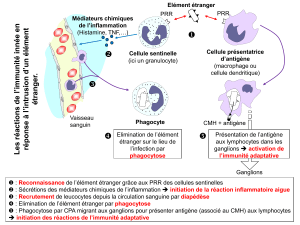
Biologie des récepteurs de l`immunité innée : applications cliniques

REVUE FRANCOPHONE DES LABORATOIRES - JUILLET-AOÛT 2010 - N°424 // 41
IMMUNOLOGIE : ACTUALITÉS 2010
Biologie des récepteurs
del’immunitéinnée:
applicationscliniques et thérapeutiques
a Laboratoire d’immunologie et d’allergologie
Centre hospitalier universitaire d’Angers – Hôpital Larrey
4, rue Larrey
49033 Angers cedex 09
b Université d'Angers – Inserm U892 – Angers
c Université d’Angers – UMR_S 892 – Angers
* Correspondance
article reçu le 21 avril, accepté le 18 mai 2010
© 2010 – Elsevier Masson SAS – Tous droits réservés.
RÉSUMÉ
La reconnaissance des microorganismes par le système immunitaire inné
implique une famille de récepteurs (pattern recognition receptor ou PRR).
Les PRR ont une spécicité génétiquement déterminée et ont pour prin-
cipale fonction de discriminer le soi du non-soi via la reconnaissance de
motifs moléculaires exprimés sélectivement par les microorganismes. Les
PRR sont également capables de discriminer le soi du soi modié, souli-
gnant leur rôle dans la détection des signaux de danger pour le système
immunitaire, qu’ils soient d’origine endogène (cellules mortes) ou d’origine
exogène (microbes). Les PRRs ont été classés en trois familles constituées
par les récepteurs associés aux cellules, impliqués dans la reconnaissance
du soi modié ou du non soi ou dans l’activation des cellules immunes
et les récepteurs solubles. Les PRR jouent un rôle crucial dans l’initiation
des réponses immunitaires adaptatives, généralement protectrices. Le
concept actuel est que le type de PRR recruté par un pathogène donné
va conditionner, du moins en partie, la nature de la réponse immunitaire
générée qui sera adaptée à la nature du microbe rencontré. À l’excep-
tion de certaines molécules, les PRR n’ont pas encore trouvé une place
importante dans l’arsenal des marqueurs utilisés dans le suivi clinique
des patients, que ce soit au cours des affections aiguës ou chroniques.
Ce constat peut résulter du fait que l’étude du système immunitaire inné
constitue une « science » encore jeune, dont le corollaire est l’absence
de sondes spéciques validées et calibrées, ainsi qu’à la biologie des
récepteurs de l’immunité innée eux-mêmes, leur redondance pouvant
masquer des anomalies de certains PRR. De nombreuses études sont
donc encore nécessaires pour élucider la biologie des PRR, leurs rôles
dans l’initiation des réponses immunitaires adaptatives et leur implication
dans des pathologies humaines.
Immunité innée – récepteurs de l’immunité innée –
marqueurs diagnostiques – immunothérapie.
SUMMARY
Biology of innate immune receptors: clinical and
diagnostic applications
The recognition of microbes by the innate immune
system is mediated by a family of receptors called
pattern recognition receptor (PRR). The specicity of
PRR is genetically determined and their main function
is to discriminate self from non self via the recogni-
tion of motifs expressed by microbes. PRR can also
discriminate self from modied self, underlining the
role played by PRR in the detection of endogenous
(dying cells) and exogenous (microbes) danger signals.
PRRs are classied in three families: cell-associated
(involved in the internalization or cell activation) and
soluble receptors, also called opsonins. PRR play a
pivotal in the initiation of protective adaptive immune
responses. The actual concept is that the type of PRR
recruited will determine, at least in part, the nature
of the immune reponse generated, adapted to the
motif encountered. Except some PRR, such as the
soluble PRR belonging to the pentraxin family, PRR
are not used as markers in clinical and diagnostic
studies. This may result from the redundancy of PRR
that may mask the absence of inefcacy of one PRR
that can be compensated by another one. Additional
studies are thus required to understand the biology
of the PRR and their potential use as clinical and/or
biological markers in human diseases.
Innate immunity – PRR – diagnosis – immunitaire
therapy.
Pascale Jeannina,b,c,*, Sébastien Jaillonb,c , Yves Delnestea,b,c
1. Introduction
Le système immunitaire est organisé autour de deux com-
posantes : l’immunité innée et l’immunité adaptative. Le
système immunitaire inné constitue la première ligne de
défense contre les microorganismes. Ce système est très
ancien en termes d’évolution et les molécules impliquées
sont particulièrement bien conservées entre les espèces.
Ainsi, certaines familles de molécules de l’immunité innée
telles que les défensines (peptides antimicrobiens) sont
retrouvées aussi bien chez les plantes que chez les mam-
mifères. Chez les vertébrés, ce système est complété par
l’immunité adaptative ou acquise, plus récente en termes
d’évolution. L’immunité adaptative, qui découle des méca-

42 // REVUE FRANCOPHONE DES LABORATOIRES - JUILLET-AOÛT 2010 - N°424
nismes initiés par le système immunitaire inné, permet la
génération des réponses spéciques d’antigènes, comme
par exemple la génération des anticorps. Elle constitue
le support de la mémoire immunologique et utilise des
récepteurs spéciques d’une très grande variabilité codés
par réarrangements géniques.
Les organismes sont en permanence exposés à des
microorganismes et doivent ainsi reconnaître les patho-
gènes efcacement et rapidement. La reconnaissance des
pathogènes par le système immunitaire inné implique une
famille de récepteurs communément appelés « pattern reco-
gnition receptor » (PRR) [1]. Ces récepteurs reconnaissent
des motifs conservés parmi les familles de microorganis-
mes, tel que le lipopolysaccharide (LPS) et leur expression,
constitutive dans la majorité des cas, n’est pas sujette à
des réorganisations géniques. Les cellules impliquées
dans la réponse immunitaire innée sont essentiellement
les épithéliums (première barrière du système immunitaire
inné contre les microorganismes) et les globules blancs
(polynucléaires neutrophiles, éosinophiles et basophiles ;
les monocytes ; les macrophages ; les cellules dendritiques
et les cellules « natural killer » [NK]), à l’exception notable
des lymphocytes T et des lymphocytes B.
Le système immunitaire inné a connu un net regain d’inté-
rêt ces dernières années grâce à la découverte des bases
moléculaires de reconnaissance des microbes, de l’identi-
cation de nouvelles fonctions des cellules de l’immunité
innée et leur rôle dans l’initiation des réponses immunitaires
adaptatives. Dans cette revue, nous nous focaliserons sur
les récepteurs de l’immunité innée.
La reconnaissance des pathogènes par les cellules immu-
nitaires innées fait intervenir les PRRs. Ces récepteurs ont
une spécicité génétiquement déterminée, et ont pour prin-
cipale fonction de discriminer le soi du non-soi via la recon-
naissance de motifs moléculaires exprimés sélectivement
par les microorganismes. Ces motifs microbiens, appelés
« pathogen-associated molecular pattern » ou PAMPs, sont
des structures hautement conservées durant l’évolution et
ne sont pas exprimées par les cellules de l’hôte.
Il est important de noter que les PRR sont également impli-
qués dans la reconnaissance du soi modié (lipoprotéines
de faible densité oxydées (OxLDL), cellules apoptotiques)
soulignant le rôle des PRR dans la détection des agents
représentant des signaux de danger pour le système immu-
nitaire, qu’ils soient d’origine endogène (cellules mortes)
ou d’origine exogène (microbes).
Les PRRs ont été classés en trois familles constituées
par les récepteurs associés aux cellules et les récepteurs
solubles.
2. Récepteurs de l’immunité
innée associés aux cellules
Les récepteurs associés aux cellules sont divisés en deux
familles sur la base de leurs fonctions : les récepteurs
d’endocytose et les récepteurs de signalisation.
2.1. Les récepteurs d’endocytose
Ces récepteurs membranaires sont spécialisés dans la
reconnaissance et l’internalisation des microorganismes.
Ce groupe est composé principalement des récepteurs
d’épuration et des récepteurs lectiniques de type-C.
2.1.1. Les récepteurs d’épuration
Les récepteurs d’épuration ou « scavenger receptors » (SR)
sont des glycoprotéines de surface exprimées principale-
ment par les cellules endothéliales, les macrophages et les
cellules dendritiques. Leur fonction principale est de xer
et d’internaliser les Ox-LDL mais pas les LDL natives [2].
Exprimés par les macrophages, ils sont impliqués dans
la formation de cellules riches en lipides (« foam cells »),
constituants majeurs de la plaque d’athérome. Comme la
majorité des PRR, les SRs reconnaissent également d’autres
ligands du soi-modié, tels que les cellules apoptotiques
et des composants de la matrice extracellulaire (ex : col-
lagène) ainsi qu’une grande diversité de microorganismes.
La reconnaissance d’un ligand est suivie de son internali-
sation et/ou de sa destruction. Sur la base de leur structure
moléculaire, les SRs sont classés en huit classes (classe A
à H), brièvement décrites ci-dessous (revue [2, 3]).
2.2.1.1. Classe A
Les « scavenger receptors » de classe A comprennent cinq
membres : MARCO (récepteur des macrophages avec un
domaine collagène), SRCL (« scavenger receptor » avec
un domaine lectininique de type-C), class A scavenger
receptor (SR-AI), SR-AII et SR-AIII. SR-AI, SR-AII et SR-
AIII sont générées suite à l’épissage alternatif d’un même
ARN messager (ARNm). À l’exception de SR-AIII qui reste
séquestré dans le réticulum endoplasmique, SR-AI et SR-
AII xent les Ox-LDL, le LPS et les bactéries Gram positif
(Streptococcus agalactiae, Staphylococcus aureus) et
Gram négatif (Escherichia coli) [4]. MARCO présente une
structure proche de SR-A avec un domaine collagène plus
important et se trouve exprimée par les macrophages et
les cellules dendritiques. Ce récepteur xe les Ox-LDL et
les bactéries Gram positif (e.g. Staphylococcus aureus)
et Gram négatif (e.g. Neisseria meningitidis). SRCL est
exprimée dans un large panel de tissus (e.g. poumons,
cœur, côlon) et xe les Ox-LDL modiées et les bactéries
Gram positif (e.g. Staphylococcus aureus) et Gram négatif
(e.g. Escherichia coli) [4].
2.2.2.2. Classe B
Cette classe contient les gènes codant CD36 et SR-B. Le
gène SR-B subit un épissage alternatif pour donner les
protéines SR-BI (CLA-1) et SR-BII. Les SRs de classe B
sont des glycoprotéines de type III (plusieurs domaines
transmembranaires) avec une topologie caractéristique
en « épingle à cheveux » où les extrémités carboxy- et
amino-terminales sont localisées dans la cellule. CD36 est
exprimée par les monocytes, les macrophages, les cellules
endothéliales et les plaquettes. SR-B est exprimée par les
macrophages, les cellules dendritiques, les adipocytes et
les hépatocytes. Les SR de classe B peuvent reconnaître
les Ox-LDL, le collagène et les cellules apoptotiques [5].
De plus, CD36 reconnaît les érythrocytes infectés par Plas-
modium falciparum (pathogène responsable du paludisme)
[6] et SR-BI reconnaît le virus de l’hépatite C associé à
des lipoprotéines.

REVUE FRANCOPHONE DES LABORATOIRES - JUILLET-AOÛT 2010 - N°424 // 43
IMMUNOLOGIE : ACTUALITÉS 2010
2.2.2.3. Classe C
La classe C contient uniquement un gène (dSR-CI) exprimé
chez la mouche Drosophila melanogaster, sans équivalent
connu chez les autres eucaryotes.
2.2.2.4. Classe D
La classe D contient les molécules CD68 (appelée micro-
sialine chez la souris) et les glycoprotéines de membrane
lysosomale (LAMP pour « lysosome-associated membrane
protein ») 1, 2 et 3. CD68 est exprimée par les macropha-
ges, les cellules dendritiques, les cellules de Langerhans
et les ostéoclastes [7]. LAMP-1 et LAMP-2 sont exprimées
par un grand nombre de cellules alors que LAMP-3 est
exprimée uniquement par les cellules dendritiques matures.
CD68 xe les Ox-LDL et les ligands des molécules LAMP
restent à déterminer.
2.2.2.5. Classe E
Un seul gène représente cette classe dont le produit se
nomme « lectin-like oxidized LDL receptor-1 » (LOX-1)
[8]. Ce récepteur fait également partie de la famille des
lectines de type-C et se trouve localisé dans un cluster
de gènes contenant d’autres membres de cette famille
(e.g. Dectin-1, CLEC-1). LOX-1 est exprimée par les cel-
lules endothéliales, les monocytes, les macrophages, les
cellules dendritiques et les cellules des muscles lisses.
LOX-1 xe les Ox-LDL, les bactéries Gram positif (e.g.
Staphylococcus aureus) et négatif (e.g. Escherichia coli)
et les cellules apoptotiques [8].
2.2.2.6. Classe F
Cette classe contient deux gènes codant pour les récep-
teurs appelés « scavenger receptor expressed by endo-
thelial cells » (SREC)-I et SREC-II. La molécule SREC-I est
exprimée par les cellules endothéliales, les monocytes et
les macrophages et présente une distribution tissulaire
similaire à SREC-II (e.g. cœur, placenta, poumons, reins,
rate) [9]. Seule SREC-I reconnaît les Ox-LDL. De plus,
SREC-I et SREC-II contiennent, au niveau de leur domaine
cytoplasmique, plusieurs sites de phosphorylation poten-
tiels qui pourraient être impliqués dans une signalisation
cellulaire après xation d’un ligand [9].
2.2.2.7. Classe G
Cette classe contient un seul membre appelé « scaven-
ger receptor that binds phosphatidylserine and oxidized
lipoprotein » (SR-PSOX). SR-PSOX xe les Ox-LDL et les
bactéries Gram positif et négatif. SR-PSOX est exprimée
par les macrophages, les cellules dendritiques, les cellules
endothéliales et les cellules musculaires lisses [10].
2.2.2.8. Classe H
La classe H contient deux membres identiques à 39 se
nommant « fasciclin, epithelial growth factor (EGF)-like, lami-
nin-type EGF-like and Link domain-containing scavenger
receptor-1 » (FEEL-1) et FEEL-2. Ce sont des glycoprotéines
qui xent les bactéries Gram positif et négatif. De plus,
FEEL-1 et FEEL-2 reconnaissent les protéines glycosylées
nommées « advanced glycation end-products » (AGE) qui
s’accumulent dans les vaisseaux. FEEL-1 et FEEL-2 sont
exprimées dans plusieurs tissus (e.g. rate, moelle osseuse)
mais seul FEEL-1 est exprimée par les monocytes, les
macrophages et les cellules endothéliales.
L’expression des SR peut-être régulée par différents fac-
teurs environnementaux, essentiellement des cytokines
et des composants microbiens.
2.1.2. Les récepteurs aux lectines de type-C
Les récepteurs aux lectines de type-C (C-type lectin recep-
tors ou CLR) constituent une famille de protéines exclusi-
vement retrouvées chez les métazoaires. Le terme lectines
de type-C désigne toute protéine qui reconnaît des carbo-
hydrates de manière dépendante du calcium. Cependant,
cette dénition est mal adaptée puisque certains membres
de cette famille, dont l’appartenance est basée sur des
homologies de structure, xent leurs ligands indépendam-
ment du calcium (ex : Dectin-1). En plus de leur capacité
d’endocytose, ces PRRs possèdent des propriétés de
signalisation. Les CLR sont représentées par des protéi-
nes associées aux cellules, participant à l’endocytose des
microorganismes et à la signalisation intracellulaire, et par
des protéines solubles (ex : les collectines) impliquées dans
l’opsonisation des microorganismes [11]. Cette section
présente les principaux CLRs associés aux cellules.
2.1.2.1. DC-SIGN/CD209
Le récepteur DC-SIGN/CD209 (dendritic cell-specic ICAM-
3-grabbing nonintegrin) est exprimé par les cellules dendri-
tiques et les macrophages alvéolaires et placentaires. Ce
récepteur reconnaît un grand nombre de pathogènes tels
que Mycobacterium tuberculosis, Helicobacter pylori, Leish-
mania mexicana, Schistosoma mansoni, Candida albicans,
Klebsiella pneumoniae et le virus de l’immunodécience
humaine (HIV) [12]. Le rôle de DC-SIGN dans la défense
contre certaines infections virales reste controversé. En
effet, lors d’une infection par HIV ou par le virus de l’hépa-
tite C, DC-SIGN serait une voie de protection du virus et
d’infection « en trans » des cellules voisines (e.g lympho-
cytes T, hépatocytes). Cependant, des travaux récents
montrent que DC-SIGN induit une internalisation de HIV
permettant la destruction de la majeure partie des virions
et la présentation de peptides viraux associés au CMH-I.
Il est ainsi probable que l’ensemble des virions n’est pas
détruit et qu’une petite partie induit une trans-infection des
lymphocytes T. L’interaction du virus HIV avec DC-SIGN
induit la production de chimiokines attirant les lymphocytes
T CD4+ qui sont les principales cibles du virus mais HIV
inhibe la capacité des cellules dendritiques à induire l’ac-
tivation des lymphocytes T. La reconnaissance de HIV par
DC-SIGN génèrerait un environnement favorable au virus
en inhibant la maturation de la cellule dendritique et en
permettant la propagation du virus dans les lymphocytes
T. DC-SIGN interagit avec Mycobacterium tuberculosis
par la reconnaissance de lipoarabinomannane mannosylé
(ManLAM), constituant majeur de la paroi du pathogène.
Le ManLAM est également sécrété par les macrophages
infectés par Mycobacterium tuberculosis et induit une
importante sécrétion de la cytokine immunosuppressive
interleukine 10 (IL-10). Comme observé avec HIV, M. tuber-
culosis empreinte la voie DC-SIGN pour inhiber le système
immunitaire et permettre sa prolifération.

44 // REVUE FRANCOPHONE DES LABORATOIRES - JUILLET-AOÛT 2010 - N°424
2.1.2.2. Récepteur au mannose
Le récepteur au mannose est exprimé par les macro-
phages, les cellules dendritiques et les cellules endothéliales
hépatiques et lymphatiques [13]. Ce récepteur reconnaît
plusieurs pathogènes tels que HIV, Candida albicans,
Pneumocystis carinii et Mycabacterium tuberculosis. Les
données concernant le rôle de cette molécule dans la
présentation des antigènes par les molécules du CMH-II
sont contradictoires. En effet, la capacité du récepteur
au mannose à induire l’endocytose du ManLAM, qui sera
ensuite présenté aux lymphocytes T en association avec
la molécule CD1b pour induire une immunité protectrice,
est contrebalancée par la capacité du ManLAM à bloquer
la formation du phagolysosome, permettant ainsi la survie
de M. tuberculosis dans le macrophage. Le rôle in vivo du
récepteur au mannose dans la défense contre les patho-
gènes est controversé. En effet, de manière surprenante,
les souris décientes pour le gène codant le récepteur au
mannose ne sont pas plus sensibles que les souris sau-
vages à une infection par Candida albicans ou Pneumocystis
carinii. En revanche, ces souris ont un taux plasmatique
élevé d’hydrolases lysosomales, conrmant le rôle de ce
récepteur dans l’élimination d’antigènes du non-soi.
2.1.2.3. Dectin-1
Dectin-1, glycoprotéine exprimée par les macrophages,
les cellules dendritiques et les neutrophiles, reconnaît les
glucanes présents dans la paroi des champignons et des
levures [14]. Dectin-1 interagit avec Aspergillus fumiga-
tus, Candida albicans et Pneumocystis carinii. Les souris
décientes pour le gène codant Dectin-1 présentent une
sensibilité accrue aux infections fongiques, comparative-
ment aux souris sauvages. Dectin-1 induit la sécrétion du
tumor necrosis factor α (TNFα par les macrophages en
réponse à des stimulations fongiques. Dectin-1 est éga-
lement impliquée dans la génération des lymphocytesT
pro-inammatoires (Th17) via l’induction de cytokines pro-
inammatoires interleukine (IL) 6, TNFαet IL-23.
2.2. Les récepteurs de signalisation
2.2.1. Les molécules « toll-like receptors »
Les molécules « toll-like receptors » (TLR) [15] sont des
récepteurs très conservés durant l’évolution, des homolo-
gues étant retrouvés chez les insectes et les vertébrés. Le
premier membre de la famille, nommé toll, a été identié
chez Drosophila melanogaster et intervient dans la mise
en place de l’axe dorso-ventral durant le développement
embryonnaire et dans la protection antimicrobienne. À ce
jour, 13 molécules TLRs sont retrouvées chez les mam-
mifères : 10 chez l’homme (TLR1-10) et 12 chez la souris
(TLR1-9 et TLR11-13). Les molécules TLRs sont des pro-
téines transmembranaires caractérisées par un domaine
riche en leucine (« leucine-rich region » ou LRR) couplé à
un domaine proche de celui du récepteur à l’IL-1, nommé
TIR (« toll/IL-1 receptor »). Le domaine LRR est impliqué
dans la reconnaissance des PAMPs (bactéries, parasites,
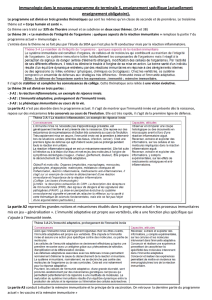
champignons, virus). À ce jour, un ligand a été identié
pour la plupart des TLRs humains (tableau I).
Les molécules TLRs sont exprimées par de nombreux types
cellulaires. Leur distribution subcellulaire est associée à
la nature des ligands qu’elles reconnaissent. Les molé-
cules TLR1, 2, 4, 5 et 6, qui reconnaissent des composés
microbiens, sont exprimées à la surface des cellules alors
que les molécules TLR3, 7, 8 et 9, qui reconnaissent des
acides nucléiques, sont localisées dans les endosomes/
lysosomes. Après la reconnaissance d’un ligand, deux
molécules TLR s’homodimérisent ou s’hétérodimérisent
an d’initier une cascade de signalisation aboutissant à
l’induction d’une réponse pro-inammatoire. Deux voies de
signalisation sont initiées par les TLRs. La première aboutit
à l’activation du facteur de transcription NF-κB impliqué
dans la production des cytokines pro-inammatoires et la
deuxième conduit à la sécrétion des interférons de type I
impliqués dans la protection antivirale.
Les molécules TLR sont impliquées dans de nombreux
processus d’activation des cellules du système immuni-
taire inné tels que (i) l’activation des cellules dendritiques
(augmentation de l’expression des molécules de costi-
mulation, production de cytokines pro-inammatoires),
(ii) l’activation des cellules NK, notamment par TLR2 et
TLR5 (production de défensines et de cytokines pro-
inammatoires (IL-6) et immunostimulatrices (interféron γ
ou IFNγ)), (iii) l’activation des polynucléaires. Les molécules
TLRs sont également exprimées par les cellules épithé-
liales et leur activation induit la production de cytokines
pro-inammatoires. Cependant, les cellules épithéliales
seraient hyporéactives aux stimulations par les ligands
des TLRs an de maintenir l’homéostasie tissulaire dans
les épithéliums en contact permanent avec des microbes
(tractus digestif, génital ou respiratoire).
2.2.2. Les récepteurs intracellulaires hors TLRs
Le système TLR n’étant pas utilisé pour la détection de
pathogènes ayant envahi le cytosol, d’autres familles de
récepteurs, tels que les molécules « NOD-like receptor »
(NLR) et « RNA helicase » (RLR) permettent une reconnais-
sance cytosolique des composés bactériens et viraux,
Tableau I – Agonistes des molécules TLR humaines.
TLR PAMPs
TLR1/TLR2 Triacyl lipopeptides (bactéries, mycobactéries)
TLR2
Lipoprotéines
Acide lipotéichoïque (bactéries Gram positif)
Lipoarabinomannan (mycobactérie)
OmpA (bactéries Gram négatif)
Porine (bactéries Gram négatif)
TLR3 ARN double brin (virus)
TLR4
LPS (bactéries Gram négatif)
Protéines virales (virus respiratoire syncytial)
phosphatidylinositol mannosides (mycobactéries)
TLR5 Flagelline (bactéries à agelle)
TLR6/TLR2 Diacyl lipopeptides (mycoplasme)
TLR7 ARN simple brin (virus)
TLR8 ARN simple brin (virus)
TLR9 ADN hypométhylé (bactéries)
TLR10 Ligand non-connu
Abréviations: OmpA, outer membrane protein A; LPS, lipopolysaccharide.

REVUE FRANCOPHONE DES LABORATOIRES - JUILLET-AOÛT 2010 - N°424 // 45
IMMUNOLOGIE : ACTUALITÉS 2010
respectivement [16]. Les protéines de la famille des NLRs,
représentée par les molécules NOD1 et NOD2, sont syn-
thétisées sous une forme inactive et la xation d’un ligand
induit l’oligomérisation du récepteur et donc la signalisa-
tion. NOD1 et NOD2 reconnaissent respectivement l’acide
γ-D-glutamyl-meso-diaminopimelique (DAP) et le muramyl
dipeptide, constituants du peptidoglycane bactérien. La
reconnaissance d’un ligand par NOD1 et NOD2 induit
l’activation de NF-κB et donc la production de cytokines
pro-inammatoires ainsi que l’expression de peptides
antibactériens, tel que la cryptidine.
La détection des virus indépendante des molécules TLR
est médiée par les RLR qui reconnaissent les ARNs viraux
synthétisés dans le cytoplasme des cellules infectées.
Les protéines « retinoic acid-inducible gene I » (RIG-I) et
« melanoma-differentiation-associated gene-5 » (MDA5)
sont des molécules cytosoliques. Les souris décientes
pour les gènes RIG-I et MDA5 présentent une importante
sensibilité aux infections virales. La signalisation via RIG-I
et MDA5 aboutit à l’activation des facteurs de transcription
impliqués dans la production des interférons de type I et
des cytokines pro-inammatoires.
3. Récepteurs solubles
del’immunité innée
Aux récepteurs associés aux cellules s’ajoutent les com-
posants de l’immunité humorale ou PRRs solubles, tels
que les collectines, les colines et les pentraxines. Cette
famille est constituée de molécules hétérogènes, pré-
sentées comme étant les ancêtres des anticorps. Ces
molécules participent à l’opsonisation des microorganis-
mes, à l’activation du complément, à la reconnaissance
et l’élimination des cellules apoptotiques, et modulent la
réponse inammatoire.
3.1. Les collectines et les ficolines
Les collectines appartiennent à la famille des lectines de
type-C et sont constituées de neuf membres : «mannose
binding lectin » (MBL), conglutinine, « surfactant protein »
(SP)-A, SP-D, les collectines (CL) CL-P1, CL-L1 et CL-K1.
Les collectines CL-43 et CL-46 ne sont exprimées que
chez les bovidés [17].
MBL est une protéine sérique produite par le foie et l’in-
testin grêle. SP-A et SP-D sont principalement produites
par les épithéliums pulmonaires et sont donc retrouvées
au niveau des alvéoles pulmonaires. SP-A est également
produite par la muqueuse intestinale, le thymus ou encore
la prostate et se retrouve dans le liquide séminal et les
muqueuses. CL-L1 et CL-K1 ont une expression ubiquitaire,
à l’exception des muscles squelettiques. CL-P1 est une
collectine particulière car elle est exprimée à la membrane
des cellules endothéliales ; elle a également été qualiée
de SR puisqu’elle reconnaît les Ox-LDL.
La famille des colines est composée de trois membres :
la H-coline (également connue sous le nom d’antigène
Hakata), la L-coline et la M-coline. À l’exception de la
M-coline qui n’est pas retrouvée dans le sérum, H-coline
et L-coline sont des protéines sériques (3 à 4 μg/mL).
Cependant, le fait que la M-coline soit détectée dans les
granules de sécrétion des neutrophiles suggère que cette
protéine pourrait également être sécrétée. H-coline est
produite par le foie et les épithéliums bronchiques, L-co-
line est produite par le foie et la M-coline est produite
par l’utérus, les monocytes, les neutrophiles et les cellules
épithéliales alvéolaires.
Les collectines et les colines possèdent une structure de
base particulière constituée de sous-unités composées par
trois chaînes polypeptidiques identiques (homotrimère).
Chaque polypeptide de collectines contient notamment
une région similaire au collagène et le domaine de recon-
naissance des carbohydrates est localisé à l’extrémité
apicale. Le polypeptide des colines possède une organi-
sation similaire à l’exception d’un domaine brinogène à la
place du domaine carbohydrate. Le nombre de sous-uni-
tés trimériques diffère entre les collectines (SP-D contient
4trimères alors que SP-A et MBL contiennent 6 trimères)
et les colines (L-coline contient 4 trimères alors que
la H-coline contient 3 trimères). Ces molécules recon-
naissent différents sucres, tels que mannose, glucose,
L-fucose, N-acetyl-mannosamine et N-acétyl-glucosa-
mine. Le calcium est indispensable à la reconnaissance
des carbohydrates par les collectines alors qu’il n’est pas
nécessaire pour les colines. Les collectines et les colines
reconnaissent un grand nombre de pathogènes (e.g. virus,
bactéries, champignons). MBL et SP-A reconnaissent par
exemple Staphylococcus aureus, Streptococcus pneumo-
niae, Escherichia coli, Aspergillus fumigatus alors que la
L-coline reconnaît Salmonella typhimurium.
3.2. Les pentraxines
Les pentraxines forment une famille de protéines très
conservées durant l’évolution et sont ainsi retrouvées
chez les mammifères, les arthropodes, les amphibiens,
les oiseaux et les vertébrés inférieurs. Sur la base de leur
structure primaire, les pentraxines ont été divisées en pen-
traxines courtes et en pentraxines longues. Les protéines
de la phase aiguë de l’inammation « C-reactive protein »
(CRP) et « serum amyloid P » (SAP) constituent les pen-
traxines courtes alors que la pentraxine 3 (PTX3) fait partie
des pentraxines longues. CRP a été, durant les années
30, le premier membre de la famille des PRRs découvert
dans le sérum de patient en réponse à une infection [18].
Les pentraxines xent de nombreux ligands du soi et du
non soi (tableau II).
3.2.1. Les pentraxines courtes
Les pentraxines courtes CRP (PTX1) et SAP (PTX2) pos-
sèdent 51 % d’identité de séquence en acides aminés et
proviennent de la duplication d’un gène ancestral. Au niveau
structural, CRP est composée de cinq monomères formant
ainsi un pentamère qui a donné le nom de pentraxine à cette
famille. CRP est produit par les hépatocytes en réponse à
l’IL-6 et son expression est potentialisée par l’IL-1β.
CRP a été mise en évidence et caractérisée sous la forme
d’une fraction protéique capable de se lier à la phosphoryl-
choline, composant majeur de la fraction C de la capsule
du pneumocoque, d’où sa dénomination de C-reactive
protein. CRP et SAP se fixent à de nombreux ligands
(tableau II). CRP engage des liaisons dépendantes du
calcium avec les groupements phosphocholines des mem-
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
1
/
11
100%