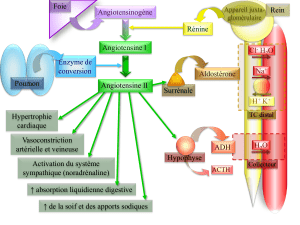
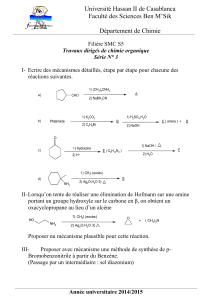
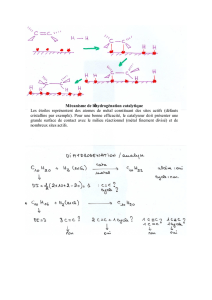
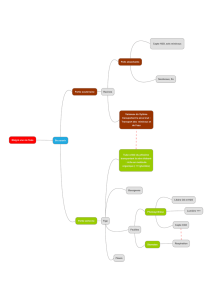
Biocatalyse et éléments lourds - Institut des Métaux en Biologie de

Cours de Fabrice THOMAS
Chimie bio-inorganique des
éléments lourds
I. Introduction
II. Enzymes à Mo
III. Enzymes à W
IV. Formiate dehydrogenase: enzyme à W ou Mo
V. Conclusion: Pourquoi Mo et W ?

I. Introduction
1. Généralités
• Rayons atomiques: Mo, W (1.36-1.37 Ǻ),
• Rayons ioniques: 0.62 (MoVI), 0.65 (WVI) soit ≈
FeIII, > VV, CrVII et < CoII, NiII, CuII, ZnII
• Electronégativités: 2.16 – 2.36 selon Pauling
(Mo – W) soit > bloc d première ligne
• Métaux relativement inertes (surtout W)
•E°(WO
2/W) et (WO3/WO2) < 0 V
ÆDegré ox courants: +2 à +6, seul +4 à +6
dans les systèmes bio

Géométries et structures électroniques
Octaèdre déformé:
DMSO réductase
Pyramide base carrée
déformée: Sulfite Oxydase
Tétraèdre: MoO42-
O
Mo
S
O
S
SCys
Mo
O
O
O
O
O
Mo
S
S
S
S
OSer/SCys/SeCys
d9
d8
d7
d6
d5
d4
d3
d2
d1
AuPtIrOsReWTaHfLa
AgPdRhRuTcMoNbZrY
CuNiCoFeMnCrVTiSc
MoIV: d2; MoVI: d0
WIV: d2; WVI: d0
ÆValables pour degrés rédox +IV à +VI
Seuls bioéléments de la
2eme et 3eme ligne
Géométrie du Mo:
ÆW: Le plus lourd des bio-éléments

Mo est le plus abondant des
métaux de transition dans les
océans! Or la vie vient des
océans
Mo et W parmi les moins abondants des métaux
de transition dans la croûte terrestre!
Molybdenite (MoS2)
Mo
O
O
O
O
Mo soluble
Abondance naturelle

Biodisponibilité
• A l’origine de la vie : Sulfures de Mo,W
peu solubles dans l’eau, donc peu
biodisponibles.
• L’atmosphère devient moins réductrice: Molybdenite
(MoS2)
Oxydation de la molybdenite: 2 MoS2+ 7 O2+ 2 H2O Æ2 MoO42- + 4 SO2+ 4 H+
Forme WO3(H2O)xen solution acide, peu soluble
Forme soluble prédomine
sur une large gamme de
potentiel et de pH
Biodisponible
dans les océans
(Oxydes de Mo: [MoO4]2-,
[Mo7O24]6-, [Mo8O26]4-…)
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
 53
53
 54
54
 55
55
 56
56
 57
57
 58
58
 59
59
 60
60
 61
61
 62
62
 63
63
 64
64
 65
65
1
/
65
100%