GG cours 3

La PCR
Ingrédients requis
séquences à reproduire ou matrice: bien sûr l’ADN génomique isolé est une excellente matrice puisqu’il contient tous
les gènes. Mais la matrice peut aussi être un fragment purifié ou un ADNc etc… Cas particulier, on peut utiliser un
hybride ARN/ADN c’est à dire le stade intermédiaire de la fabrication d’ADNc.
amorces (primers, oligonucléotides): parfaitement complémentaires inverse des extrémités du fragment à amplifier.
Attention au sens de polymérisation! ce sont de petits fragments d’ADNSS qui font en général entre 18 et 30
nucléotides. Quand ils font 18 nucléotides, ce sont des 18mers.
déoxynucléotides: puisqu’on fabrique de l’ADN
Taq polymérase à 72°C + Mg: Le Mg est un cofacteur nécessaire à toutes les polymérases
Principe de la PCR
On a un fragment d’ADN matrice, on veut la séquence que l’on souhaite amplifier.
1ère étape : dénaturation, on va chauffer notre ADN matrice à 95°C : les 2 brins se séparent
2ème étape : hybridation : en abaissant la température, il faut que les brins se ressoudent. Les primers hybrident sur les
brins, il faut être en très large excès en primers. De plus, il faut que la température favorise les primers qui doivent se
mettre sur leur cible pour l’hybrider en complémentaire inverse. Chaque primer a une température spécifique.
3ème étape : la polymérisation à 72°C. la polymérase en présence d’une amorce, Mg, et va pouvoir allonger les primers
jusqu’à qu’il n’y ait plus rien à copier.
Lorsque le 1er cycle est fini, on recommence un 2nd cycle. Et c’est pendant le 3ème cycle qu’on voit apparaitre les premiers
fragments cibles. Au 4ème cycle, on va passer à 8 copies cibles soit 16 copies en tout.
Nombre de cycles : n
Nombre de copies : 2n
Nombre de copies cibles 2n-2n
Classiquement une PCR c’est 25 cycles soit + de 33 millions copies cibles.
Choix des amorces: complémentaires inverses
Température d’hybridation: Tm -(2 à 5°C) Tm : température melting = température de fusion c’est la température où la moitié
des oligonucléotides sont hybridés et l’autre moitié ne le sont pas.
Calcul d’un Tm: 2x(nb A+T) + 4x(nb G+C)
Les deux amorces doivent avoir des températures d’hybridation compatibles. La température d’hybridation doit être inférieure de
2 à 5°C à la Tm.
Ex : 20mers 5A, 5T, 5G, 5C
Tm = 60°C en température de fusion
95°C : dénaturation 30 sec à 1min
55 à 58°C : hybridation 30 sec à 1 min.
Pour une PCR il faut au moins 2 oligonucléotides, sa séquence sera différente. Il est difficile de trouver 2 nucléotides ayant la
même Tm, on se place alors à une T° correcte pour les 2 nucléotides. C’est la T° d’hybridation compatible.
72°C : polymérisation 1 min/kb à amplifier. C’est la T° idéale pour que la polymérase fonctionne. A reproduire 25 fois
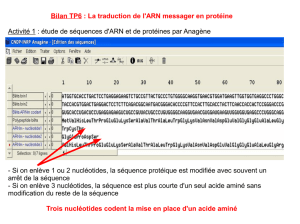
Le séquençage
Il est essentiel de séquencer l’ADN pour connaitre les bornes pour faire la PCR. La technique de séquence la plus couramment
utilisée est la technique de terminaison de chaîne. Pour réaliser on a besoin d’ADN matrice d’amorce, de polymérase, de
didéoxynucléotides, et des déoxydenucléotides.
L’identification de séquences
Dénaturation de la matrice à séquencer
Hybridation avec des amorces spécifiques
Répartition en 4 tubes distincts
Dans chaque tube ajout des 4 dXTP et l’un des 4 ddXTP
Chaque fois qu’un ddXTP est incorporé, la séquence s’arrête
On obtient donc:
- dans le tube contenant les ddATP, des fragments dont la synthèse s’est arrêtée à chaque A possible
- dans le tube contenant les ddCTP, des fragments dont la synthèse s’est arrêtée à chaque C possible
- etc….
En faisant migrer ces fragments on les sépare en fonction de leur taille et on « lit » ainsi la séquence de 5’ vers 3’ du bas vers le
haut du gel
Chaque didéoxynucléotide est “tagué” avec un fluorophore, ce qui permet de détecter les différents produits de séquençage
Séparation: migration de tous les produits ensemble dans un tube capillaire et détection de la fluorescence
Lorsque l’on ne connait pas la séquence nucléotidique ?
Il faut au moins connaitre une partie de la séquence en acide aminé de la protéine codée par le gène d’intérêt.
On peut retrouver la séquence en passant par le code génétique.
Le code génétique est ce qui permet de passer d’une séquence de nucléotides à une séquence d’acide aminé.

4 lettres AUGC doivent donner l’info pour 20 lettres : les 20 acides aminés
Le code doit donc comporter 3 lettres pour coder pour 20 acides aminés
1 lettre 4aa
2 lettres 4² possibilités
3 lettres 43 possibilités
C’est un code : redondant (le même acide aminé peut être codé par plusieurs triplets) ; universel (il existe une petite différence
entre le code génomique et le code mitochondrial) ; non chevauchant.
La plupart des mutations ponctuelles affectent un seul acide aminé le code ne peut donc pas être chevauchant
Il existe un ordre dans l’organisation du code :
3ème base : pour beaucoup de codons, quelque soit la base l’acide aminé est le même.
2ème base : base pyrimidique code pour un acide aminé hydrophobe ; base purique code pour un acide aminé polaire
Dans la nature le plus souvent pypy ou pupu
1ère base : une mutation sur cette base va souvent donner un acide aminé différent
Expériences qui ont conduit à l’élucidation du code génétique
1- Niremberg et Mathei (voir diapo)
Le code génétique traduction (=translation)
Le mécanisme de biosynthèse des protéines
C’est un processus séquentiel : l’ARNm est lu et les acides aminés accrochés les uns aux autres
Il requiert :
des ribosomes
des ARNt chargés
l’ARNm
des enzymes
ATP ou GTP (énergie)
La nécessité d’un adaptateur entre les acides aminés et l’ARNm
ARN: molécule polaire
Acides aminés : polaires ou hydrophobes
ARNt il a globalement la forme d’une feuille de trèfle et il possède 2 régions essentielles à sa fonction la première forme une
boucle anti-codon et c’est à ce niveau qu’il va se fixer à l’ARNm. A l’extrémité 3’ de l’ARNt se trouve le site de fixation de l’acide
aminé.

La structure de l’enzyme d’activation
• l’aminoacyl tRNA synthétase
c’est l’enzyme qui permet de lier l’acide aminé sur l’ARNt
elle possède 3 sites de reconnaissance :
o un pour l’acide aminé
o un pour l’ARNt
o un pour l’ATP
il existe autant d’enzymes que d’acides aminés
La réaction de charge de l’acide aminé sur l’ARNt
A la fin on a relarguage de l’enzyme.
Le premier codon et la séquence de shine-delgarno. C’est au niveau de cette séquence que l’ARN 16S (ribosomial) va pouvoir
s’accrocher.

L’utilisation d’oligonucléotides dégénérés en PCR
On va fabriquer des mélanges d’oligonucléotides.
C’est une population d’oligonucléotides qui ont été fabriqués pour être sur à 100% d’avoir au moins une séquence commune.
Le Nterm correspond au 5’
Les techniques de clonage d’un gène
1
/
4
100%