travaux diriges de l`unite d`enseignement lv 303 « biochimie

UFR DES SCIENCES DU VIVANT
DIVISION DE BIOCHIMIE
ANNEE UNIVERSITAIRE 2013-2014
TRAVAUX DIRIGES
DE L’UNITE D’ENSEIGNEMENT LV 303
« BIOCHIMIE FONDAMENTALE »
LICENCE DE SCIENCES ET TECHNOLOGIES : MENTION SCIENCES DU VIVANT

TD 1-Protéines
2

TD 1-Protéines
3
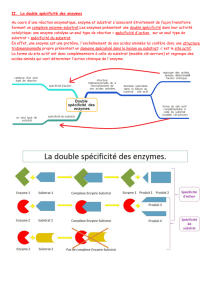
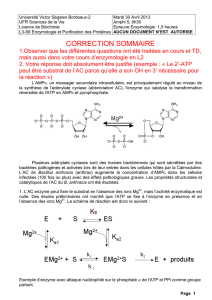
TD 1 : PROTEINES
A- Etude structurale et fonctionnelle de la protéine MutS :
D’après Manelyte L et al , Nucleic Acids Research, 2006, Vol. 34(18), 5270–5279.
La protéine MutS est un facteur essentiel du système de réparation post-réplicatif des
mésappariements de l'ADN (MMR : Mutation Mismatch Repair, voir cours de LV203). MutS est
notamment impliquée dans la reconnaissance et la fixation des mésappariements conduisant alors
au recrutement d'autres facteurs tels que MutL et MutH.
In vivo et in vitro, il a été montré que le domaine C-terminal de MutS (CTD, résidus 801 à 853) joue
un rôle essentiel dans son oligomérisation et dans sa fonction lors d’une réparation.
Première partie : Alignement de séquences
La figure 1 représente un alignement de séquences et une prédiction de structure secondaire
de la région CTD de MutS provenant de 9 espèces bactériennes différentes.
Hélice -Boucle -Hélice
Boucle - Hélice - Boucle - Hélice
Figure 1 : Alignement de séquences et prédiction de structure secondaire (S. II) de MutS chez plusieurs
espèces bactériennes. Les nombres entre parenthèses correspondent au nombre d'acides aminés
présents dans la zone définie par la séquence de référence. En fin de séquence le nombre entre
parenthèse est le nombre d'acides aminés total de la protéine.
Q1 a) Qu’est-ce qui définit la structure primaire d’une protéine ?
Q1 b) Quels sont les motifs de structure secondaire des protéines ?
Q2) Commentez les données présentées figure 1. Que pouvez-vous conclure quant aux structures
primaire et secondaire de la région CTD ?

TD 1-Protéines
4
Deuxième partie : Production de mutants de MutS
Dans le but d’analyser la structure et la fonction du domaine C-terminal de la protéine MutS,
différents variants portant une étiquette 6-Histidine ont été construits et exprimés :
- MutS-CTD : domaine C-terminal de MutS (résidus 801à 853),
- MutS-CTD ∆His6 : MutS-CTD délétée de son étiquette histidine,
- MutS- CTDD835R : MutS-CTD pour lequel le résidu D835 a été remplacé par R.
D’après leur séquence, les protéines MutS-CTD et MutS-CTDD835R ont une masse moléculaire calculée
de 7,7 kDa et MutS-CTD ∆His6 une masse moléculaire de 6,4 kDa.
Les protéines exprimées ont été purifiées à homogénéité par chromatographie d’affinité sur colonne
de nickel et par chromatographie d’exclusion moléculaire sur Sephadex G200. Les protéines purifiées
ont ensuite été déposées sur un gel SDS-PAGE en présence d’un agent réducteur (figure 2). Elles ont
été détectées après coloration au bleu de Coomassie.
Q3 a) Donnez le principe général d'une chromatographie d'affinité. Expliquer l'utilisation du nickel
dans l’expérience.
Q3 b) Donnez le principe de la chromatographie d’exclusion moléculaire (ou gel filtration).
Q3 c) Quelles autres chromatographies connaissez-vous ?
Q4 a) Quel est le rôle de l’agent réducteur utilisé en SDS-PAGE ? Citez-en un.
Q4 b) Quel est le principe de la coloration au bleu de Coomassie ? Quelle autre technique, plus
sensible, est utilisée pour colorer un gel de protéines ?
Figure 2 : Analyse des variants MutS-
CTD purifiés. Gel de polyacrylamide
SDS-
PAGE coloré au bleu de
Coomassie. Les masses des marqueurs
protéiques (M) sont indiquées à gauche
de la figure.
Q5) Commentez brièvement les résultats du gel SDS-PAGE.
Troisième partie : Etude structurale de MutS
Des spectres de dichroïsme circulaire ont été obtenus pour les 3 variants de MutS. Le spectre de
MutS-CTD est donné (figure 3). Les deux autres mutants présentent un spectre superposable.

TD 1-Protéines
5
Q6 a) Quelle information structurale est apportée par un spectre de dichroïsme circulaire ?
Q6 b) Quelle est la structure majoritaire des trois variants de MutS ? Est-ce en accord avec les
données de la figure 1 ?
Quatrième partie : Statut oligomérique de MutS
Les masses moléculaires des différents variants de MutS-CTD ont été estimées par sédimentation à
l’équilibre (à des concentrations de 20 µM). On obtient les valeurs suivantes :
- 29800 +/- 410 Da pour MutS-CTD,
- 25300 +/- 380 Da pour MutS-CTD ∆His 6,
- 16500+ /- 370 Da pour MutS-CTDD835R.
Q7 a) Donnez le principe de la sédimentation à l'équilibre.
Q7 b) Quel est le statut oligomérique des trois variants de MutS-CTD ?
Q8) Quel est l’intérêt d’étudier le variant MutS CTD ∆His 6 dans ces expériences ?
Une chromatographie d’exclusion moléculaire (Superdex G-200) a été réalisée avec différentes
concentrations de MutS-CTD et MutS-CTDD835R (figure 4).
MutS, µM
Figure 4 : Analyse par chromatographie d’exclusion moléculaire des variants MutS-CTD. : MutS-CTD
(O) et MutS-CTDD835R (∆). Dans le graphique, le volume d’élution (en mL) de la protéine est reporté en
fonction de la concentration protéique au moment du dépôt. Les lignes pointillées indiquent les
volumes d’élution des formes de 16500 Da et 29800 Da respectivement sur la base des analyses des
résultats de sédimentation à l’équilibre.
Q9) Pourquoi différentes concentrations des variants MutS-CTD ont-elles été testées en
chromatographie d’exclusion moléculaire ?
Figure 3 : Spectre de dichroïsme
circulaire obtenu pour MutS-CTD.
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
1
/
32
100%