Université Montpellier 2 - Master Sciences pour l`Environnement

Université Montpellier 2 - Master Sciences pour l’Environnement
Mention : Biologie des Plantes, des Microorganismes, Bioingéniéries, Bioprocédés
Spécialité : IMHE (Interactions Microorganismes, Hôtes, Environnements)
Renvoyer la proposition de sujet de stage à : [email protected] / patricia.quemener@univ-
montp2.fr /
Responsable des stages d’Initiation à la Recherche de M1 et de M2 :
Tatiana Vallaeys : [email protected] Tél : 04 67 14 40 11
Administration :
Patricia Quéméner : patricia.quemener@univ-montp2.fr
Sujet de stage préciser le niveau souhaité M1 et/ou M2 (le cas échéant indiquez vos
préférences: Niveau souhaité M1
Hémo-microbiote de Crassostrea gigas :
diversité microbienne et relation avec le système immunitaire des huîtres.
Responsable (s) de stage : ESCOUBAS Jean-Michel Chargé de Recherche (CR1) CNRS
Encadrant (si différent du Responsable de stage notamment dans le cas où le Responsable n’est pas
un Enseignant-Chercheur ou un Chercheur) :
Personnel technique éventuellement impliqué dans la formation du stagiaire :
Tel et Email du Responsable de stage et de l’encadrant (si différent du Responsable de stage) :
Email : jmescoubas@univ-montp2.fr Tél. : 04 67 14 47 11
Laboratoire d’Accueil et nom du Directeur :
Interactions Hôtes Pathogènes Environnements – (UMR 5244)
CNRS-IFREMER-Université de Montpellier 2-Université de Perpignan Via Domitia.
Directeur : MITTA Guillaume
Equipe d’Accueil : Mécanismes d’Interaction et d’adaptation en Milieu Marin (MIMM)
Directrice d’équipe : DESTOUMIEUX-GARZON Delphine
L’équipe MIMM est localisée au sein de l’Université de Montpellier 2 (Bât. 24 1er étage).
Dans quel contexte s’insère le sujet de stage (démarrage d’un projet, travail partiel d’un sujet de
thèse …….) :
Le sujet de stage correspond à projet de recherche émergeant au sein de l’équipe MIMM et pour
lequel une demande de financement a été déposée auprès de l’INSU du CNRS (projets EC2CO 2015).
De plus, il sera adossé à un projet plus vaste qui vient d’être financé par l’ANR et qui vise à décrypter
les facteurs responsables des phénomènes de mortalité des huîtres (ANR DECIPHER 2015-2018).
Techniques qui seront principalement utilisées lors de ce stage :
Microbiologie, microscopie, cytométrie en flux, biologie moléculaire (extractions ADN et ARN, PCR
et RT-PCR).

Description du stage : donner un résumé (contexte, problématique, matériels et méthodes).
Contexte
L’hémo-microbiote correspond à la cohorte de microorganismes associés à l’hémolymphe
(équivalent du sang) d’animaux sains. Contrairement à la plupart des animaux, nombre de mollusques
bivalves et notamment l’huitre creuse Crassostrea gigas ont une hémolymphe qui n’est pas stérile.
L’existence même de l’hémo-microbiote constitue un paradoxe puisque l’hémolymphe contient les
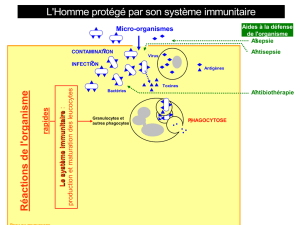
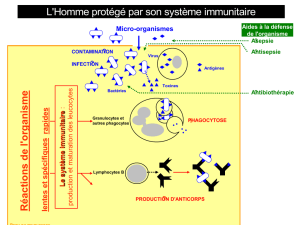
hémocytes, cellules circulantes jouant un rôle clé dans la réponse anti-infectieuse (1). Dans ce
contexte, comment expliquer la cohabitation entre les hémocytes, impliqués dans la reconnaissance et
l’élimination du non-soi et l’hémo-microbiote non-soi par excellence ? Peu de travaux sont consacrés
à l’hémo-microbiote des bivalves et ils sont cantonnés à l’étude des bactéries (2). Pourtant, des études
d’écologie microbienne ont révélées la richesse et la diversité en microorganismes du milieu marin
(virus, bactéries, champignons, algues, …).
Programme de travail
Le projet de recherche a pour objectif de caractériser la diversité microbienne de l’hémo-
microbiote de Crassostrea gigas et d’étudier sa sensibilité à la réponse immunitaire de l’hôte. Pour
cela, le projet sera décliné en deux phases.
La première phase a pour objectif de déterminer la diversité et l’abondance de l’hémo-
microbiote (bactéries, virus, eucaryotes). Pour cela, les communautés microbiennes présentes dans le
plasma (hémolymphe débarrassée des hémocytes) seront analysées par cytométrie en flux et
microscopie (épifluorescence et électronique) afin d’obtenir des données quantitatives et qualitatives
sur les communautés microbiennes de l’hémolymphe (proportions de virus, procaryotes et
eucaryotes). Des expériences de fractionnement du plasma seront également effectuées par
centrifugation et filtration afin de séparer les microorganismes (d’une part les procaryotes et les pico-
eucaryotes ; d’autre part les virus). Les différentes fractions seront analysées en cytométrie et en
microscopie afin de contrôler la nature et le nombre de microorganismes récupérés par rapport aux
échantillons de départ. Les microorganismes ainsi préparés feront l’objet d’analyses via des
approches de métagénomique globale (metabarecoding et shotgun metagenomic sequencing ; dans le
cadre du projet EC2CO 2015).
La deuxième phase a pour objectif d’étudier la sensibilité de l’hémo-microbiote à la réponse
immunitaire de l’hôte. Pour cela, nous suivrons l’impact d’une stimulation de la réponse
immunitaire sur la composition de l’hémo-microbiote. La réponse immunitaire sera induite via
l’injection de bactéries non pathogènes à des lots d’huîtres, puis l’hémolymphe sera prélevé à
différents temps post injection. La composition de l’hémo-microbiote sera analysée via les approches
développées durant la première phase du projet. En parallèle, l’ARN des hémocytes sera extrait pour
vérifier l’induction de la réponse immunitaire (RT-qPCR sur des gènes immunitaires) (3).
Résultat attendu :
Ce projet, de par la mise en œuvre d’approches sans a priori, va permettre d’appréhender la
diversité de l’hémo-microbiote de l’huître Crassostrea gigas en déterminant notamment si des
microorganismes autres que les bactéries (virus, eucaryotes) sont présents dans l’hémolymphe
d’animaux sains. L’étude de la sensibilité de l’hémo-microbiote à la réponse immunitaire de l’hôte
permettra d’appréhender le rôle de cette dernière dans le contrôle, voire la sélection de la composition
de son hémo-microbiote.
Références :
(1) Schmitt, P., Rosa, R. D., Duperthuy, M., de Lorgeril, J., Bachere, E., and Destoumieux-Garzon, D. (2012). The
Antimicrobial Defense of the Pacific Oyster, Crassostrea gigas. How Diversity may Compensate for Scarcity in the
Regulation of Resident/Pathogenic Microflora. Front Microbiol 3, 160.
(2) Wendling, C. C., Batista, F. M., and Wegner, K. M. (2014). Persistence, seasonal dynamics and pathogenic potential of
Vibrio communities from pacific oyster hemolymph. PLoS One 9, e94256.
(3) de Lorgeril, J., Zenagui, R., Rosa, R. D., Piquemal, D., and Bachere, E. (2011). Whole transcriptome profiling of
successful immune response to Vibrio infections in the oyster Crassostrea gigas by digital gene expression analysis.
PLoS One 6, e23142.

1
/
3
100%