Poly Régression multiple

!"#!$#%!&%' ' #&'
Master2(NCC(
Modules(Statistiques(–(V.(Fourcassié(&(C.(Jost(
(
(
(
(
(
(
(
INTRODUCTION(AUX(MODELES(LINEAIRES(GENERAUX(
!"#$#%&'()*$#&%(+,-#'(.(")+/(
(
EXERCICES(
(
(
(
(
(
(
(
(
(
(
SOMMAIRE(
(
1'EXEMPLE(DE(REGRESSION(SIMPLE(:(REGRESSION(DE()0123421(SUR(50678697 ................................ 2'
:'EXEMPLE(DE(REGRESSION(MULTIPLE(:(REGRESSION(DE()0123421(SUR(;659<(ET(=>?@ ................ 10'
3'EXEMPLE(DE(REGRESSION(MULTIPLE(:(REGRESSION(DE()0123421(SUR(=>?@(27("=4..................... 19'
4'EXEMPLE(DE(REGRESSION(MULTIPLE(:(TRAITEMENT(DU(FICHIER(FOURMI_TRANSPORT ............... 23'
5'POUR(ALLER(PLUS(LOIN(:(QUELQUES(OUVRAGES ......................................................................... 31'

!"#!$#%!&%' ' #%'
(
1 Exemple(de(régression(simple(:(régression(de(lifexpef(sur(birthrat(
'
Description(du(Tableau(Country.txt((
(
(
(
!
!
!
!
!
!
!
!
!
!
!
!
1.1 Préparation!du!Tableau!Country.txt!
1.1.1 Charger!le!jeu!de!données!dans!R!et!le!placer!dans!un!tableau!nommé!«!Country!»!
'
()*+',-.**/)0'1'23'456.78)7'9)0'9.**/)0'23'-)6+:0'+*';:<=:)7'8)>8'.+'?)'67)00)26@6:)7'
'
3'A.+*87B'C2'7)@9D8@E?)FGAH#IDD#A.+*87BD8>8GJ'=)@9)7KLMNOJ'0)6KGGJ'*@D087:*P0KGQRGJ'9)<KGDGJ'
087:6DS=:8)KLMNOT'
'
U:0+@?:0)7'?)'V)+'9)'9.**/)0'9@*0'M'
'
A?:W+)7'0+7'?)'E.+8.*','U:0+@?:0)7'1'
>(0=.S-@8@FA.+*87BJ'6?@<)5)*8KX2%!Y%!!XJ';.*8KP)8M<597FX?.PZ.*8XTJ'5@>S:98=K[!J'
5@>=):P=8K\!T(
(
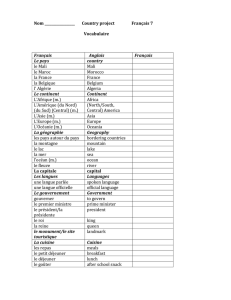
Nom
colonne
Signification
country
pop92
Population, 1992, in millions
urban
Percent urban, 1992
gdp
GDP per capita
lifeexpm
Male life expectancy 1992
lifeexpf
Female life expectancy 1992
birthrat
Births per 1000 population, 1992
deathrat
Deaths per 1000 people, 1992
infmr
Infant mortality rate 1992 (per 1000 live
births)
fertrate
Fertility rate per woman, 1990
region
Region of the world
develop
Status as Developing Country
radio
Radios per 100 people
phone
Phones per 100 people
hospbed
Hospital beds per 10,000 people
docs
Doctors per 10,000 people

!"#!$#%!&%' ' #\'
1.1.2 Compte!le!nombre!de!valeurs!manquantes!dans!le!tableau!Country!pour!chaque!
VI!
(
()*+',']8@8:08:W+)0'1'23'M/0+5/0'23'-/*.5E7)7'?)0'.E0)7^@8:.*0'5@*W+@*8)0'
'
3'0@66?BFA.+*87BJ';+*<8:.*F>TF0+5F:0D*@F>TTTT'_'QR'<.+*80'
'
'<.+*87B''''6.6$%''''+7E@*''''''P96'?:;))>65'?:;))>6;'E:78=7@8'9)@8=7@8''
'''''''!''''''''!''''''''!''''''''!''''''''!''''''''!''''''''&''''''''&''
''':*;57';)787@8)'''7)P:.*''9)^)?.6''''7@9:.''''6=.*)''=.06E)9'''''9.<0''
'''''''!''''''''%''''''''!''''''''!''''''''!''''''''[''''''''`''''''''&'
'
1.1.3 Elimine!les!observations!ayant!des!valeurs!manquantes!
(
()*+','-.**/)0'1'23'a)+'9)'9.**/)0'@<8:;'23'O?:5:*)7'?)0'<@0'<.*8)*@*8'9)0'^@?)+70'5@*W+@*8)0'
'
3'A.+*87B'C2'*@D.5:8FA.+*87BT'
'
1.1.4 Calcule!la!moyenne!et!l’écartHtype!des!VI!
(
()*+',']8@8:08:W+)0'1'23'M/0+5/0'23']8@8:08:W+)0'9)0<7:68:^)0'
'
3'*+5]+55@7BFA.+*87BbJ<FGE:78=7@8GJ'G9)@8=7@8GJ'G9)^)?.6GJ'G9.<0GJ'G;)787@8)GJ'GP96GJ'
G=.06E)9GJ'G:*;57GJ'G?:;))>6;GJ'G?:;))>65GJ'G6=.*)GJ'G6.6$%GJ'G7@9:.GJ'G7)P:.*GJ'G+7E@*GTcJ'
08@8:08:<0K<FG5)@*GJ'G09GTT'
''''''''''''''''''5)@*''''''''''''09'''' *'
E:78=7@8''''\!Dd!````"''''&\D&[&$[%"''&!e'
9)@8=7@8''''&!D%d"`&$!'''''dDd`!$!!e''&!e'
9)^)?.6''''''!D"`&$!d['''''!Dd%"$`!e''&!e'
9.<0''''''''&!D$%d&[[[''''&&D%"\%&dd''&!e'
;)787@8)'''''dD&!\[!$e'''''&D$%"!\!`''&!e'
P96'''''''dde!D$$!d"`%'`\\!De!!e%`\''&!e'
=.06E)9'''''\dD&!&e%"`''''\&D`"`$&\"''&!e'
:*;57'''''''e`D\\"&d%$''''d\De[$[`[!''&!e'
?:;))>6;''''`"D!%[e"&d''''&&D&e$%&%d''&!e'
?:;))>65''''`%De`&$!d['''''$D"$%$"`&''&!e'
6=.*)'''''''&eD&%&d&\e''''%%D"\de"$d''&!e'
6.6$%'''''''deD%!$\$!e'''&ddD[&$$&d$'' &!e'
7@9:.'''''''\\D%[%!`d"''''\&D%e!&[e"''&!e'
7)P:.*'''''''[D`"`&$!e'''''eD&!$\"&`''&!e'
+7E@*'''''''e!Dd\&d%[`''''%dDd`%"eee''&!e'

!"#!$#%!&%' ' #d'
(
1.2 Représenter!la!variable!lifeexpf!en!fonction!de!la!variable!birthrat!
f*'<=)7<=)'g'0@^.:7'0:J'6.+7'?)0'6@B0'7/6)78.7:/0'9@*0'?@'E@0)J'?h)06/7@*<)'9)'^:)'9)0';)55)0')08'
?:/)'@+'8@+>'9)'*@8@?:8/D'
'
()*+'i7@6=)0'23'Q+@P)0'9)'6.:*80'
'
0<@88)76?.8F?:;))>6;jE:78=7@8J'7)PD?:*)K?5J'05..8=KZRk]OJ'?@E)?0KZRk]OJ'E.>6?.80KX>BXJ'06@*K!DeJ'
9@8@KA.+*87BT'
'
'
'
1.3 Calculer!les!coefficients!de!la!droite!des!moindres!carrés!et!leur!intervalle!de!
confiance!
'
()*+']8@8:08:W+)0'23'RV+08)5)*8'9)'5.9l?)0'23'M/P7)00:.*'?:*/@:7)'
'
3'M)P(.9)?D&'C2'?5F?:;))>6;jE:78=7@8J'9@8@KA.+*87BT'
3'0+55@7BFM)P(.9)?D&T'
'
'

!"#!$#%!&%' ' #e'
A@??H'
?5F;.75+?@'K'?:;))>6;'j'E:78=7@8J'9@8@'K'A.+*87BT'
'
M)0:9+@?0H'
''''''(:*'''''''''&m'''''()9:@*'''''''''\m''''''''(@>''
2&"D`!%&\'''2%D["e%$''''!D!\%!e''''\D%%%&['''&"D[&d!!''
'
A.);;:<:)*80H'
'''''''''''''O08:5@8)'']89D'O77.7''8'^@?+)''n7F3o8oT'''''
F4*8)7<)68T''[$D&%$%&'''''&Dd&`&`''''`%D$d''''C%)2&`'ppp''
E:78=7@8''''' 2!D"%`[d'''''!D!d%"`''' 2&"D!!'''C%)2&`'ppp'
222'
]:P*:;D'<.9)0H''!'qppph'!D!!&'qpph'!D!&'qph'!D!e'qDh'!D&'q'h'&''
'
M)0:9+@?'08@*9@79')77.7H'eD"d$'.*'&!\'9)P7))0'.;';7))9.5'
(+?8:6?)'M2]W+@7)9H'!D"\"%J'R9V+08)9'M20W+@7)9H'!D"\d`''
Z208@8:08:<H'%[[D$'.*'&'@*9'&!\'-ZJ''62^@?+)H'C'%D%)2&`'
'
NoteH'n.+7'.E8)*:7'?)'5r5)'5.9l?)'.*'6)+8'@+00:'6@00)7'6@7'?)0'<.55@*9)0'
'
()*+']8@8:08:W+)0'23'RV+08)5)*8'9)'5.9l?)0'23'(.9l?)'?:*/@:7)'
1.4 Ecrire!l’équation!de!la!droite!de!régression!et!interpréter!les!résultats!
'
k:;))>6;'K'4*8)7<)68'9)'?@'97.:8)'9)'7/P7)00:.*'Y'n)*8)'9)'?@'97.:8)'9)'7/P7)00:.*'>'E:78=7@8'
'''K'[$D&\'s'!D"\'E:78=7@8'
'
M)?@8:.*'87l0'0:P*:;:<@8:^)')*87)'?h)06/7@*<)'9)'^:)'9)0';)55)0')8'?)'8@+>'9)'*@8@?:8/'H'?h)06/7@*<)'9)'
^:)'9)0';)55)0'9/<7.t8'?.70W+)'?)'8@+>'9)'*@8@?:8/'@+P5)*8)D'
'
k)'5.9l?)'9)'7/P7)00:.*'?:*/@:7)')>6?:W+)')*^:7.*'"\u'9)'?@'^@7:@*<)'.E0)7^/)'9@*0'?h)06/7@*<)'9)'
^:)'9)0';)55)0D''
'
1.5 Demander!la!table!d’anova!de!l’analyse!de!régresion!
(
()*+'(.9l?)0'23'L)080'9h=B6.8=l0)0'23'L@E?)'9)'?h@*.^@'F-)5@*9)7'RQfUR'LB6)'4'0/W+)*8:)?T'
'
3'@*.^@FM)P(.9)?D&T'
R*@?B0:0'.;'U@7:@*<)'L@E?)'
'
M)06.*0)H'?:;))>6;'
'''''''''''' ' -;'']+5']W''()@*']W''Z'^@?+)'''''n7F3ZT'''''
E:78=7@8''''' ' &''$ed"D!'''''$ed"'''%[[D[[''' C'%D%)2&`'ppp'
M)0:9+@?0'' ' &!\''\d!\D$'''''''\\'''''''''''''''''''''''
222'
]:P*:;D'<.9)0H''!'XpppX'!D!!&'XppX'!D!&'XpX'!D!e'XDX'!D&'X'X'&'
'
k)'Z'9+'5.9l?)'F9/Vg'9.**/'@+'6.:*8'%T'<.77)06.*9'@+'7@8:.'H'^@7:@*<)')>6?:W+/)'6@7'?)'5.9l?)'9)'
7/P7)00:.*'?:*/@:7)#'^@7:@*<)'7/0:9+)??)K'F$ed"D!'#&T'#'F\d!\D$'#'&!\TK'%[[D[[D'
(
L)08)'0:'?h:*8)7<)68'
9)'?@'97.:8)'9)'
7/P7)00:.*')08'v'!'
L)08)'0:'?@'6)*8)'9)'?@'
97.:8)'9)'7/P7)00:.*'
)08'v'!'
n@78'9)'?@'^@7:@*<)'
)>6?:W+/)'6@7'?)'5.9l?)'
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
1
/
22
100%