Enquête sur les maladies héréditaires

Travaux Pratiques Juillet 2011
©2011 Sami Khuri 1
TP 3
Enquête sur les maladies héréditaires - I
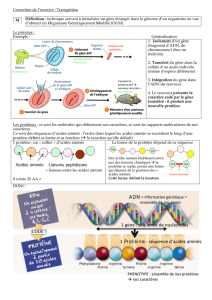
Le but de ces exercices est d’introduire des outils de Bioinformatique. Vous découvrirez un gène
humain important et verrez comment les mutations qui s’y déroulent engendrent la maladie
héritée.
Vous apprendrez comment:
• Traduire une séquence d’ADN en une séquence d'acides aminés
• Rechercher des séquences similaires dans le génome humain
• Relever l'effet des mutations dans cette séquence et les relier avec une maladie
héritée
Résolvez les trois exercices qui consistent à:
A) Traduire une séquence ADN
B) Trouver des protéines similaires dans le génome humain
C) Relever les effets de mutations
A) Traduction d’une séquence d’ADN
Ne pas oublier :
• Le code génétique est composé de trois lettres qui codent pour des acides aminés
• Un gène commence toujours avec le codon initiateur (Start) ATG. ATG code pour
l’acide aminé qu’on appelle la méthionine.
• Il existe trois codons Stop (TAA, TGA, TAG) qui indiquent la fin de protéines
Essayons cet exercice à la main.
Considérer la séquence d’ADN représentée ci-dessous :
>Séquence ADN “inconnue”
atggtgcacctgactcctgaggagaagtctgccgttactgccctgtggggcaaggtgaac
gtggatgaagttggtggtgaggccctgggcaggttgctggtggtctacccttggacccag
aggttctttgagtcctttggggatctgtccactcctgatgctgttatgggcaaccctaag
gtgaaggctcatggcaagaaagtgctcggtgcctttagtgatggcctggctcacctggac
aacctcaagggcacctttgccacactgagtgagctgcactgtgacaagctgcacgtggat
cctgagaacttcaggctcctgggcaacgtgctggtctgtgtgctggcccatcactttggc
aaagaattcaccccaccagtgcaggctgcctatcagaaagtggtggctggtgtggctaat
gccctggcccacaagtatcactaa
Traduisez les premières 57 bases de la séquence inconnue :
atg gtg cac ctg act cct gag gag aag tct gcc gtt act gcc ctg tgg
___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___
ggc aag gtg
___ ___ ___
Les ordinateurs sont généralement plus rapides et plus exacts

Travaux Pratiques Juillet 2011
©2011 Sami Khuri 2
• Rendez vous à http://www.cs.sjsu.edu/faculty/khuri/AVL/AVL_TP_Sequences.doc
• Utilisez la souris pour copier la séquence : Séquence ADN “inconnue”
• Rendez vous à http://us.expasy.org/tools/dna.html, l’outil de traduction ExPasy
• Collez la séquence dans l’espace vide
• Allez à “Output format” (plus bas) et choisissez “Includes nucleotide sequence”
• Cliquez sur le bouton “TRANSLATE SEQUENCE”.
Les points suivants décrivent ce que vous devriez voir :
• La séquence d’ADN et les acides aminés relatifs ligne par ligne
• Les bioinormaticiens utilisent principalement le "code de la lettre individuelle" où
chaque acide aminé est représenté par une lettre unique.
Pourquoi y a-t-il six traductions?
o Il y a six traductions possibles de toute séquence ADN parce qu'il y a six cadres de
lecture possibles selon où vous commencez à lire la séquence
Lequel des six cadres de lecture est la traduction correcte?
o Le premier cadre de lecture avec une Méthionine au commencement et un tiret
(indiquant un codon Stop) à la fin est la traduction correcte.
B) Chercher le génome humain pour trouver des
protéines similaires
Vous allez trouver la séquence telle qu’elle est dans le génome humain. Cela vous donnera
alors l'accès à tout ce que les recherches ont pu trouver au sujet du gène et son produit, la
protéine. Supposez que vous découvrez un ADN ou une séquence de la protéine dans votre
recherche. Vous pensez que cette séquence joue un rôle important dans le système que vous
étudiez. Dans le premier cas, il existe une éventualité assez considérable qu’un autre
chercheur ait déjà trouvé une fonction à votre séquence. Sinon, dans le deuxième cas et si
vous ne trouvez pas d’égal exact pour votre séquence dans le génome humain, vous pourriez
alors trouver une séquence similaire se situant soit dans le génome humain, soit dans un
autre organisme. Cette simple recherche peut vous dire beaucoup au sujet de votre gène ou
protéine.
• Revenez à la production (résultat) de l’outil de traduction. (ExPasy dans ce cas)
• Cliquez sur le lien “5'3' Frame 1”
• Maintenant vous devriez voir uniquement la séquence d’acides aminés (sans la
séquence d’ADN). Copiez entièrement la séquence d’acides aminés à l’exception du
codon Stop (Stop). Ouvrez WordPad et collez la séquence d’acides aminés.
Sauvegardez le fichier puisque nous allons l’utiliser dans la deuxième partie du
problème lorsque nous analyserons les mutations.
• Pour savoir tout ce que les recherches ont pu découvrir à propos de notre séquence,
visitez le centre national pour l'information de la biotechnologie “National Center for
Biotechnology Information” (NCBI) :
http://www.ncbi.nlm.nih.gov

Travaux Pratiques Juillet 2011
©2011 Sami Khuri 3
• Cliquez sur “BLAST” sous “Popular Resources” (à droite de la page)
• Choisissez “protein blast” en dessous de “Basic Blast”
• Collez la séquence d’acides aminés dans l’espace vide
• Choisissez “Refence proteins (refseq_protein)” de “Database” en dessous de
“Choose Search Set”
• Tapez “Homo sapiens (taxid:9606)” dans l’espace vide à côté de “Organism”.
• Cliquez sur le bouton "BLAST"
Vous obtiendrez une nouvelle page avec un tableau au sommet de l'exposition représentant les
séquences les plus similaires à la séquence initiale.
Les barres rouges indiquent les séquences qui, dans la base de données du génome humain, sont
très similaires à notre "séquence inconnue" alors que les barres roses indiquent les séquences qui
sont assez similaires à cette séquence.
• Défilez au-dessous du diagramme et vous verrez une liste de protéines semblables à
notre séquence inconnue dans le génome humain
• La première protéine est le gène de la beta-globine humaine, suivie d’une liste d'autres
gènes de la globine qui sont similaires dans leur séquence d’acides aminés. Ceci reflète
bien le fait qu’ils ont des fonctions similaires quant à la transportation de l’oxygène et
qu’ils ont tous évolué d’une protéine ancestrale commune.
• Défilez vers le bas un peu plus loin et regardez les alignements en pair entre notre
séquence inconnue et les séquences de la base de données GenBank. Notre séquence
inconnue est identique à la séquence de la beta-globine humaine. C'est une des
techniques qu'un bioinformaticien appliquerait pour trouver ce qui est codé par une
séquence particulière (notre séquence inconnue).
• Observez les alignements en pair avec quelques-unes des autres séquences de la globine
pour voir à quel point elles sont similaires ou différentes de notre séquence inconnue.
C) Observation des effets de mutations (Partie 1)
Les mutations de la séquence d’ADN affectent la protéine de différentes façons. Dans cet
exercice vous traduirez la séquence d’ADN du gène de la beta-globine humaine d’une forme
différente trouvé chez les individus souffrant d’une thalassémie.
Mutation 1 : Thalassémie
> Le mutant humain de bêta globine 1
atggtgcacctgactcctgaggagaagtctgccgttactgccctgtgggcaaggtgaac
gtggatgaagttggtggtgaggccctgggcaggttgctggtggtctacccttggaccca
gaggttctttgagtcctttggggatctgtccactcctgatgctgttatgggcaacccta
aggtgaaggctcatggcaagaaagtgctcggtgcctttagtgatggcctggctcacctg
gacaacctcaagggcacctttgccacactgagtgagctgcactgtgacaagctgcacgt
ggatcctgagaacttcaggctcctgggcaacgtgctggtctgtgtgctggcccatcact
ttggcaaagaattcaccccaccagtgcaggctgcctatcagaaagtggtggctggtgtg
gctaatgccctggcccacaagtatcactaa

Travaux Pratiques Juillet 2011
©2011 Sami Khuri 4
Un seul nucléotide fut supprimé de la “séquence inconnue” initialement donnée pour produire le
mutant ci-dessus. Pour détecter l’effet de la mutation sur la protéine beta-globine, nous aurons
besoin de la traduire en une séquence d’acides aminés exactement comme nous avons fait dans
la première partie du problème.
Commençons par la traduction des 57 premières bases à la main
atg gtg cac ctg act cct gag gag aag tct gcc gtt act gcc ctg tgg
___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___ ___
gca agg tga
___ ___ ___
Pour confirmer notre résultat, rendez vous à
http://www.cs.sjsu.edu/faculty/khuri/AVL/AVL_TP_Sequences.doc
pour obtenir la sequence “Le mutant humain de bêta globine 1” et puis à
http://us.expasy.org/tools/dna.html et traduisez le mutant. Comme auparavant, le premier cadre
de lecture est correcte. Cliquez sur le lien “5'3' Frame 1”.
Encore une fois, vous devriez voir la séquence d’acides aminés uniquement, sans la séquence
d’ADN. Copiez la séquence d’acides aminés en entier et collez-la en dessous de la séquence
d’acides aminés obtenue à la partie A du problème.
Examinez avec soin la deuxième séquence d’acides aminés et comparez-la à celle obtenue à la
partie A.
Ce que vous devriez voir est résumé ainsi :
• Les 16 premiers acides aminés sont identiques à ceux précédemment obtenus.
• Ils sont suivis par deux acides aminés qui n’étaient pas présents dans la protéine : alanine
(A) et arginine (R)
• Un codon Stop.
• Le premier codon Stop est suivi par des acides aminés qui sont différents de la séquence
beta-globine normale. Néanmoins, une fois que le codon Stop est atteint, le reste de la
séquence n’est plus essentiel.
• Ce type de mutation est ce que l’on appelle un changement de cadre, et donnent comme
résultats des protéines tronquées ou raccourcies à cause du codon stop.
Des mutations de ce type sont communes chez les personnes souffrant d’une thalassémie.
Comme les êtres humains ont deux copies de chaque gène, il est possible que les personnes
souffrant de cette mutation aient un taux de bêta-globine relativement bas résultant d’une copie
normale du gène.
1
/
4
100%