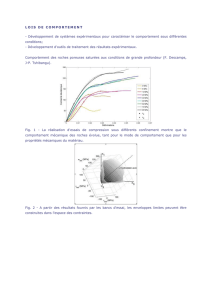
Figures

1.
2.
3.
Fig.2: Ségrégation de deux gènes (A et B) lors de la méiose.
Fig.1: Schémas des différents stades de la méiose d'une cellule végétale.
1. Interphase et prophase de la division réductionnelle d'une cellule végétale (2n=4); 2. (a)
Métaphase avec crossing-over, (b) anaphase et (c) télophase de la division réductionnelle,
suivi de (d) l'anaphase et de (e) la télophase de la division équationnelle. Les chromosomes
homologues sont de couleurs différentes et les chromatides sœurs d'un chromosome de
même couleur. 3. Autre possibilité de division réductionnelle (puis de division
équationnelle): au cours de la méiose 1, le mélange des informations héréditaires est le fruit
non seulement de la recombinaison chromosomique et mais aussi du mélange des
chromosomes d'origines paternelle (en bleu) et maternelle (en rouge).
http://www.afblum.be/bioafb/meiose/meiose.htm

Type de
Transmission Description Exemple
Autosomique
Dominante Une personne atteinte a 50% de risque de
transmettre la maladie à ses enfants. Maladie
de Huntington,
Achondroplasie
Autosomique
Récessive
Des parents sains mais porteurs d’un allèle
muté ont 25% de risque à chaque grossesse
d’avoir un enfant atteint.
Drépanocytose,
Mucoviscidose
Dominante liée à l’X
Pour une mutation dominante d’un gène du
chromosome X, le risque de transmission
diffère selon le sexe: un homme atteint ne
transmet pas la maladie à son fils, une femme
atteinte a 50% de risque de la transmettre à ses
enfants.
Maladie de Fabry
Récessive liée à l’X
Egalement dues à des mutations d’un gène du
chromosome X, les hommes sont plus souvent
atteints. Une femme atteinte à 50% de risque
d’avoir un fils atteint ou une fille porteuse.
Hémophilie A, Maladie
de Duchenne,
Daltonisme
Mitochondriale
De transmission maternelle, ces maladies
résultent de mutation de l’ADN mitochondrial.
Vu que seuls les ovocytes apportent les
mitochondries de l’embryon, seules les
femmes peuvent les transmettre.
Neuropathie optique de
Leber
Tableau 1: Les différents types de maladies génétiques.
Fig.3: Transmission d’une maladie autosomique récessive.
Eddie Baret © Alliance Sanfilippo

First cousins Double first cousins Half first cousins First cousins
once removed Second cousins
Type Degree of Relationship Proportion of Genes
in Common Coefficient of Inbreeding
in Child (F)
Monozygotic twins NA 1 NA
Parent-child 1st 1/2 1/4
Brother-sister (including
dizygotic twins) 1st 1/2 1/4
Brother-half sister 2nd 1/4 1/8
Uncle-niece or aunt-nephew 2nd 1/4 1/8
Half uncle-niece 3rd 1/8 1/16
First cousins 3rd 1/8 1/16
Double first cousins 2nd 1/4 1/8
Half first cousins 4th 1/16 1/32
First cousins once removed 4th 1/16 1/32
Second cousins 5th 1/32 1/64
Coefficients de consanguinité pour les descendants d'un certain nombre de croisements consanguins. Si une personne
est consanguine à travers plus d'une ligne de descente, les coefficients distincts sont sommés afin de trouver son
coefficient de consanguinité total. NA, non applicable.
Fig.4: Coefficient de consanguinité ou mesure de la proportion de gènes
hérités de manière homozygote entre deux individus.

Fish et al. J Cell Science, 2008
a. b. c.
Fig. 5: Divisions des cellules progénitrices neuronales.
a. Expansion latérale par division symétrique d’un progéniteur apical donnant lieu à deux
progéniteurs. b,c. Croissance radiale. b. Division asymétrique neuronale d’un progéniteur
apical donnant soit un neurone soit un autre progéniteur, ou 3. Division asymétrique
différenciative d’un progéniteur apical produisant un progéniteur apical et un progéniteur
basal qui génère ensuite deux neurones.
http://www.ateurope.org/fr/ataxie-telangiectasie/le-gene-atm/cycle-cellulaire.html
Fig6a. Le cycle cellulaire.
Représentation schématique des quatre phases du cycle cellulaire. R représente la possibilité
qu’a la cellule de sortir du cycle cellulaire et d’entrer en phase de quiescence en cas de
carence par exemple.

http://edusofad.com/www/demo/wged-scp/demo/scie1m01a3.php
Fig6b. Représentation des différentes étapes de la mitose.
Fig.7: Activation du point de contrôle G2 / M après des dommages de l'ADN.
En réponse aux dommages de l'ADN, la voie de signalisation ATM/ATR est activée, ce qui
conduit à la phosphorylation et à l'activation de Chk1 et Chk2 et à la phosphorylation
inhibitrice subséquente de Cdc25. Cdc25 phosphorylé est séquestré dans le cytoplasme par la
protéine 14-3-3, ce qui empêche l'activation du complexe cyclinB/Cdk1 par Cdc25 et
provoque l'arrêt en G2. La voie ATM/ATR active également la signalisation dépendante de
p53. Cela contribue au maintien de l'arrêt en G2 par la régulation positive de 14-3-3 qui
séquestre Cdk1 dans le cytoplasme. De plus, p53 induit l’activation de p21, un inhibiteur de
CDK qui se lie et inhibe le complexe cyclinB/Cdk1. P: phosphorylation.
Wang et al. Molecular Cancer, 2009
X
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
1
/
32
100%



![III - 1 - Structure de [2-NH2-5-Cl-C5H3NH]H2PO4](http://s1.studylibfr.com/store/data/001350928_1-6336ead36171de9b56ffcacd7d3acd1d-300x300.png)