Corrigé ET1 mai 09

BIO241 Examen terminal 1ère session (26 mai 2009)
Documents et calculatrices non autorisés
ATTENTION : Les parties A et B doivent être traitées sur des copies
séparées
7 pages
Notation sur 100 points
PARTIE A
Sujet I : Cours de Mr M. Robert-Nicoud (20 points)
La Chymotrypsine – Structure et fonction
1/ Quelle est la réaction catalysée par la chymotrypsine (type de réaction,
nature du substrat, site d’action, produits de la réaction) ?
Décrire (formule développée) la région du substrat reconnue spécifiquement par
l’enzyme.
2/ Quelles sont les caractéristiques de la structure quaternaire de cette enzyme ?
Décrire la structure du domaine qui permet à l’enzyme de reconnaître de façon
spécifique son substrat.
3/ Quels sont les 3 acides aminés impliqués dans la réaction de catalyse au
niveau du site catalytique de la chymotrypsine ?
Donner les caractéristiques des groupements fonctionnels sur les chaînes
latérales de ces acides aminés.
4/ Dans quels types de réactions ces 3 acides aminés sont-ils impliqués lors de la
catalyse ?
Quel est l’acide aminé impliqué dans la formation d’un intermédiaire covalent
enzyme-substrat au cours de la catalyse ?
5/ La chymotrypsine est synthétisée sous forme d’un précurseur inactif
(chymotrypsinogène).
Quels sont les mécanismes moléculaires impliqués dans l’activation de ce
précurseur ?
Sujet II : Cours de Mme F. Cornillon (40 points)

On souhaite étudier une nouvelle protéase P isolée du tube digestif d'un insecte
amazonien. Cette protéase existe sous deux formes Pi et Pa ayant des séquences
en acides aminés identiques. Seule la forme Pa possède une activité protéasique.
Pour élucider le mode d'activation de la protéine Pi, inactive en protéine Pa,
active, une expérience à l'aide de DIFP a été réalisée.
La découverte de la neurotoxicité du DIPF (diisopropylphosphofluoridate) a
conduit à utiliser ce réactif comme agent inactivateur d'enzyme. Le DIPF réagit
avec une sérine du site actif, selon la réaction suivante :
1. Les deux protéases Pa et Pi sont incubées séparemment en présence de
32P-DIPF pendant 30 minutes à 37°C, puis dialysées pour éliminer l'excès de
réactif radiomarqué. Les deux protéases sont alors analysées par électrophorèse
sur gel de polyacrylamide, en présence de SDS (sodium dodecyl sulfate) avec ou
sans 2-mercaptoéthanol.
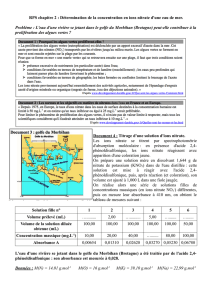
a - Après coloration au bleu de Coomassie, la photographie du gel obtenu
donne le résultat suivant :
Piste 1 Piste 2 Piste 3 Piste 4 Piste 5
enzyme
enzyme

Les marqueurs de poids moléculaire déposés dans la piste 1, sont les suivants
: βgalactosidase 120 000 g.mol-1 ; albumine du sérum de bœuf 70 000 g.mol-1 ;
ovalbumine 42 000 g.mol-1 ; trypsine 25 000 g.mol-1 ; myoglobine 16 000 g.mol-1
; peptone 9 000 g.mol-1.
La protéine Pi est déposée sur les pistes 2 et 3, respectivement en absence et
en présence de 2-mercaptoéthanol. De même, la protéine Pa est déposée sur les
pistes 4 et 5, respectivement en absence et en présence de 2-mercaptoéthanol.
Que pouvez-vous tirer de l'analyse de cette électrophorèse sur le mode
d'activation de la protéine P ? (5 points)
sachant que le 2-mercaptoéthanol réduit les ponts disulfure et que le SDS
dénature les protéines en les chargeant négativement de telle sorte qu'elles
migrent toutes vers l'anode selon leur MM
pistes 2 et 3 : bande à 90 000 g.mol-1. La protéine P sous forme inactive est
formée d'une seule chaine polypeptidique.
Pistes 4 : bande à 90 000 g.mol-1. et 5 : bandes à 60 000 et 30 000 g.mol-1.
La protéine P sous forme active est formée de 2 chaines polypeptidiques reliées
par au moins un pont S-S.
L'activation de Pi en Pa correspond à la rupture d'une liaison peptidique mais
avec maintient dans la forme active des deux chaines par un pont S-S.
b - Après autoradiographie du gel, les bandes radioactives sont révélées sur
un film photosensible par une coloration noire. Le résultat est le suivant :
Piste 1 Piste 2 Piste 3 Piste 4 Piste 5

Compléter votre analyse précédente (question a) de l'électrophorèse, par
l'étude des résultats obtenus par autoradiographie pour affiner vos conclusions
sur le mode d'activation de la protéine P. (5 points)
bandes noires dans les pistes 4 et 5 : seule la protéine sous forme active fixe
le DIFP radioactif ,
c'est une protéase à Ser active comme la chymotrypsine.
Dans la forme inactive, le site actif est inaccessible au DIFP.
bande noire de la piste 5 correspond à la chaine polypeptidique à 30 000
g.mol-1 : le site actif de l'enzyme contenant la ser active se situe sur la chaine à
30 000 g.mol-1
l'activation de l'enzyme par rupture d'une liaison peptidique induit un
changement de conformation rendant accessible le site actif situé sur la chaine à
30 000 g.mol-1
Schéma bienvenu
2. L'étude enzymatique de la protéine P s'effectue sur un substrat artificiel, le
para-nitrophénylacétate (pNPA) dont le produit d'hydrolyse absorbe à 410 nm
avec un coefficient d'extinction molaire à 410 nm de 4 000 M-1.cm-1.
a – Ecrire la réaction catalysée par la protéine P sur le substrat artificiel.
pNPA + H2O pNP + acétate (1 point)
b – L’extrait enzymatique est trop concentré en activité pour être utilisé en
l’état. Une dilution au 300ème est nécessaire.
Quel sont les volumes respectifs en solution tampon et en extrait enzymatique
que vous devez prendre pour effectuer cette dilution sachant qu'un volume
minimum de 600 µL d'extrait dilué est nécessaire pour la suite de vos manipulations
?
2 µL d'extrait + 598 µL de tampon (1 point)
c - Le protocole expérimental pour mesurer l'activité enzymatique de
l'enzyme P est le suivant. Le spectrophotomètre est programmé pour faire
une cinétique pendant 3 minutes à 410 nm. Le zéro d’absorbance est fait
à 410 nm à l’aide d’1 mL de tampon phosphate de sodium 0,1 M pH 7.
Dans une cuve de 1 mL, 450 µL de tampon phosphate 0,1 M pH 7 et 500 µL de
pNPA à 1 mM sont mis en présence de 50 µL d’extrait enzymatique dilué et
homogénéisés rapidement avant de démarrer la cinétique.
Quelle critique apportez-vous au choix du contenu de la cuve utilisé pour faire le
zéro d'absorbance ?

Il ne contient pas de pNPA et donc ne permet pas de s'affranchir de l'absorbance
éventuelle du pNPA. (1 point)
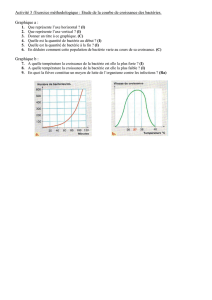
Le résultat expérimental obtenu est le suivant :
d – Donner un titre à la courbe expérimentale obtenue et l'annoter. Vous la
rendrez avec votre copie. Commenter la courbe.
Titre : Détermination de la vitesse initiale de 50 µL de protéine P diluée au
1/300, pour une concentration en substrat pNPA de 0,5 mM à pH7,4 et t° 25°C.
A410nm = f(t)
(3 points)
annotations : tangente à l'origine tracée et légendée Vo ; valeur Vo = ∆A/∆t
=(1,2-0,4)/(1-0)= 0,8 min-1
(3points)
commentaires : hyperbole, pente à l'origine nous donne Vo, plateau reflète que
l'équilibre de la réaction est atteint.
(2 points)
e – Donner la définition de l'activité enzymatique en précisant les conditions
expérimentales idéales que l'on choisit pour effectuer sa mesure.
AE ( 1mL d'enzyme, pH optimum, t°optimale, à [Substrat] fixée si possible
saturante) = nbre de nmole de substrat transformé par unité de temps, en
nmol.s-1.mL-1 , ou nkatal.mL-1
Vo
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
1
/
11
100%