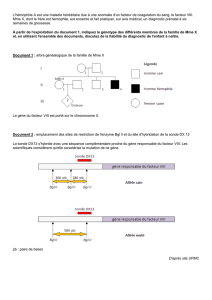
gène a

Notion de gène, expression, mutation, réparation
La fonction du gène Relation un gène/une fonction: Beadle et Tatum
La fonction dominante ou récessive
La complémentation de deux gènes
Génétique Fondamentale
Génétique Mendélienne
Génétique moléculaire
Ségrégation indépendante Un gène, deux gènes
Liaison génétique Interaction génique Epistasie et synergie
Transformation bactérienne Transformation, recombinaison,
Transductions localisées et généralisées
Conjugaison: facteur F
Opérons
Analyse
Génétique

Notion de gène
Promoteur Stop
Procaryote
Promoteur Stop
Intron
Exon1 Exon2
Codant
Eucaryote
Gène: Unité fonctionnelle et physique élémentaire de l’hérédité
qui transmet l’information d’une génération à la suivante.
Un fragment d’ADN, constitué d’une région transcrite
et de séquences régulatrices.

Transcription
RNAP
RNAP
-35 -10
+1
Promoteur: exemple E.coli
Fonction du gène
Modèle simple bactérien
Promoteur Stop
Traduction
Codon
initiation
Codon
Stop
Unité fonctionnelle: gène
La structure du gène
reflète sa fonction: Assurer
l’expression du matériel
génétique.
maturation
L’architecture d’une protéine est la clé
de la fonction des gènes.

Importance de l’architecture d’une protéine:
Notion de site actif
++
enzyme substrat
enzyme produit
La structure tridimensionnelle de la protéine
(enzyme) définit sa fonction biochimique.
Si la structure tridimensionnelle de la
protéine est modifiée, sa fonction
biochimique peut elle aussi être modifiée.
La mutation du gène peut induire ce type
de modification.

Altération d’une base
Mutation: au niveau de l’ADN
Mutation: processus par lequel des gènes passent d’une forme allélique à une autre.
ACGTC
TGCAG
ACGTC
TGCAG
ACGTC
TGTAG
ACGTC
TGCAG
ACGTC
TGTAG
ACATC
TGTAG
ACGTC
TGCAG
ACGTC
TGCAG
réplication
réplication
Génération 1
Génération 2
Altération d’une base.
Etape facilitée par les agents mutagènes:
- rayons UV
- rayons X
- rayons beta et gamma
- acide nitreux
- nitrosoguanidine
*
*
*
Exemple d’une mutation ponctuelle:
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
1
/
45
100%