Aucun titre de diapositive

Mécanismes d’adaptation des virus à leur hôte
La multiplication des virus impliquent l’utilisation des
structures de la cellule hôte (parasitisme absolu):
Liens étroit virus-hôte:
adaptation du virus à l’hôte
adaptation de l’hôte au virus

L’exemple du VIH
Les rétrovirus exploitent spécifiquement les mécanismes
cellulaires pour éviter les défenses de la cellule hôte
Le VIH = bon modèle
=> exploite les facteurs cellulaires pour sa réplication
=> évite les facteurs cellulaires de restriction
(facteurs antiviraux).
Le VIH est parfaitement adapté pour promouvoir sa
réplication.

Mécanismes d’adaptation du VIH à l’Homme
I) Echappement du VIH à la réponse immunitaire
innée :
cytokines et chemokines, cellules NK
adaptative
réponse CTL
réponse humorale
II) Restriction au niveau des gènes
facteurs d’hôtes qui restreignent la réplication du HIV
III) Evolution du virus au cours de l’infection
« Fitness »

Dissociation
de la gp120
Antigènes
cellulaires
CD4
co-
récepteur
Cellule cible
gp120
gp41
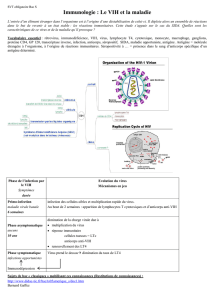
Virus de l’Immunodéficience Humaine
p24

Model of HDF roles in the HIV life cycle. With the stages of the HIV life cycle as a framework, each HDF was placed at the position most
likely to elicit HIV dependency. The function and subcellular location of HDFs were determined with the use of multiple databases
(rationale, table S4). Some proteins are in multiple locations to represent more than one possible role in the HIV life cycle. Newly
identified HDFs (red or blue, the latter if they inhibited HIV in part two only); previously implicated HDFs detected in the screen (green),
or not detected but with a relevant interaction (gray); HIV protein (black): matrix (MA), reverse transcriptase (RT), integrase (IN),
envelope (gp41, gp120) (ENV). Unfolded protein response, UPR. (Brass et al., Science, 2008,319, 921.)
Facteurs de l’hôte impliqués dans la réplication du VIH
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
 23
23
 24
24
 25
25
 26
26
 27
27
 28
28
 29
29
 30
30
 31
31
 32
32
 33
33
 34
34
 35
35
 36
36
 37
37
 38
38
 39
39
 40
40
 41
41
 42
42
 43
43
 44
44
 45
45
 46
46
 47
47
 48
48
 49
49
 50
50
 51
51
 52
52
 53
53
 54
54
 55
55
 56
56
 57
57
 58
58
1
/
58
100%