eleve - SVT Académie d`Orléans

Rappel : L'étude de l'influence de la température sur l'activité enzymatique a montré qu'aux fortes températures celle-ci
s'interrompait irréversiblement.
Hypothèse interprétative émise : lorsque la température devient trop élevée, l'agitation moléculaire est telle qu'elle
provoque la rupture de liaisons dans les molécules enzymatiques (et/ou du substrat). La structure de ces molécules est alors
modifiée et ne permet plus la catalyse de la réaction chimique
Objectif du TP : Rechercher s'il existe une relation entre la structure moléculaire d'une enzyme et son activité.
Pour cela on se propose de comparer les modèles moléculaires :
d'enzymes ayant des activités très différentes, le lysozyme et l'ADN polymérase (le lysozyme est une enzyme qui
hydrolyse les liaisons entre des composés associés aux peptides des parois bactériennes et qui présente donc une activité
bactéricide. L'ADN polymérase réalise la duplication de l'ADN).
d'enzymes ayant la même activité mais prélevées dans des organismes appartenant à des espèces différentes (lysozyme
humain et de poulet)
d'enzymes dont les séquences protéiques sont très voisines (deux molécules de lysozyme humain mais dont l'une est
mois efficace).
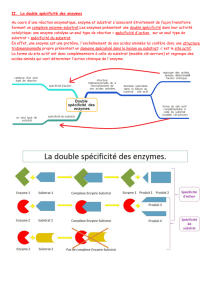
Le complexe enzyme substrat , notion de site actif
Comparaison de la structure moléculaire du lysozyme du blanc d’œuf de poule et de l'ADN polymérase
Objectifs
Actions
Lancer la visionneuse de molécules (Molusc)
Dans le dossier Molusc ouvrir le fichier index.htm à partir du
Navigateur (Netscape ou Internet explorer). Dans la page
d’accueil choisir l’option :comparaison de molécules.
Charger les deux modèles moléculaires :
-1hew.pdb, lysozyme du blanc d’œuf de poule avec son
substrat (en fait ici une molécule de trisaccharide, le Tri-N-
acétyl-chito-triose.
- pdb1bpy.pdb : ADN polymérase et son substrat (l'ADN)
- clic sur bouton Modèle 1, Parcourir, 1hew.pdb, ouvrir,
charger. La première molécule apparaît à gauche de l’écran.
- clic sur bouton Modèle 2 et refaire les mêmes procédures
pour pdb1bpy.pdb.
Traiter les molécules de façon à distinguer l’enzyme de son
substrat.
- clic sur bouton Les deux modèles (pour que les commandes
qui suivent s’appliquent aux deux modèles), colorer par
chaîne. Les molécules enzymatiques apparaissent en bleu
foncé, les molécules de substrat en couleurs différentes
Modifier le type de représentation des molécules
- clic sur Afficher puis sur sphères
Supprimer les molécules d'eau qui entourent les molécules
- clic sur effacer , eau
Déplacer la molécule dans l'espace
- placer le curseur de la souris sur la molécule, déplacer la
souris en maintenant le bouton gauche enfoncé
Faire un zoom
- maintenir la touche majuscule du clavier enfoncée tout en
effectuant l'opération précédente
(En cas d'erreur de manipulation choisir l'option Réinitialiser dans le menu Edition.)
Comparaison de la structure moléculaire du lysozyme du blanc d’œuf de poule et du lysozyme humain
Refaire les mêmes procédures avec deux autres molécules 1JWR.pdb, lysozyme humain et 193L.pdb lysozyme du blanc
d’œuf de poule ; ces deux molécules sont présentées sans leur substrat (Les colorer selon leur structure)
Après avoir comparé deux à deux les structures moléculaires des enzymes étudiées et observé la forme tridimensionnelle de
leur substrat, proposez une explication au fait qu’une enzyme n’agisse que sur un type de molécule (spécificité de substrat).
Les molécules enzymatiques présentent un site actif. Proposez une définition de cette expression.
RELATION ENTRE L’ACTIVITE D’UNE ENZYME ET SA STRUCTURE

Séquence des acides aminés et activité de l’enzyme
Certaines personnes possèdent un lysozyme ayant une faible activité catalytique même dans des conditions de
température et de pH favorables. On cherche à en trouver les causes à travers l’étude de la molécule du lysozyme recueillie
chez ces personnes, puis isolée et purifié. le fichier 134L.pdb est le modèle de cette molécule.
Charger le fichier 134L.pdb à la place de193L.pdb.
Objectifs
Actions
Comparer la séquence des acides aminés des
deux molécules.
Clic sur bouton Mode séquence. Notez les différences dans les séquences.
Pour connaître le nom complet des acides aminés et leur position dans la
chaîne polypeptidique laisser le curseur de la souris sur leurs abréviations.
Repérez dans le modèle en 3D de la molécule
la position des acides aminés qui diffèrent en
les sélectionnant
Choisir sphères dans le menu Afficher, vert dans le menu Colorer,
remplacer dans Sélection puis cliquer sur les acides aminés en question
dans les séquences protéiques.
Observer la position des acides aminés sélectionnés par rapport au site
actif.
Quitter le mode séquence
Fermer la fenêtre en cliquant sur la croix en haut à droite
Retrouver la forme initiale du modèle
moléculaire
Choisir l'option réinitialiser dans le menu Edition
Faire le bilan de vos observations.
Vous allez maintenant comparer les dimensions du site actif des deux molécules. Pour cela vous allez sélectionner des
atomes appartenant à des couples d’acides aminés situés de part et d’autre des bords du creux constituant le site actif, les
faire apparaître sous forme de sphères colorées pour mieux les repérer et noter les distances qui les séparent dans le tableau
ci-dessous
Objectifs
Actions
Sélectionner les différentes paires d’atomes
Exemple de la paire ASN 44 . CB et ALA 111 . CB *:
Sélectionner tout. Veiller à ce que l'option Les deux modèles soit
bien sélectionnée.
Dans le cadre prévu pour entrer les commandes, taper :
select*44.CB, *111.CB Exécuter
Afficher sphères
colorer En…rouge (utiliser d’autres couleurs pour les autres
couples)
Recommencer toutes ces opérations pour les couples suivants
Mesurer les distances entre atomes de même
couleur :
Option
souris
distances
Cliquer ensuite à l’aide du bouton gauche sur les atomes sélectionnés de la
molécule. Le nom des atomes apparaît dans le cadre en bas à droite de
l’écran ainsi que la distance qui les sépare (exprimée en Angström, 1A° =
10-10m). Cliquer deux fois sur le deuxième atome pour que son nom
apparaisse.
* Signification des nomenclatures utilisées : VAL 110. CB : C= carbone, B= en position β de l’acide aminé Valine (VAL)
situé en 110ème position dans la séquence protéique de l’enzyme.
Distance en A°
VAL 110.CB
GLY 48.CA
ALA 108.O
ASN 60.CB
ARG 115.N
ASP 49.CB
ALA 111.CB
ASN 44.CB
1JWR (actif)
134L (peu actif)
Analyser les résultats et émettre une hypothèse pour expliquer la perte d’activité du lysozyme 134L.
Conclusion :
Faire une synthèse sur les relations établies entre la structure et l'activité d'une enzyme.
Les observations faites sur les modèles moléculaires étayent-elles l'hypothèse formulée au départ ?
1
/
2
100%