Partie II - Exercice 2 - Raymond Rodriguez SVTperso

Bac blanc, TS
Proposition de corrigé pour la partie I
Introduction
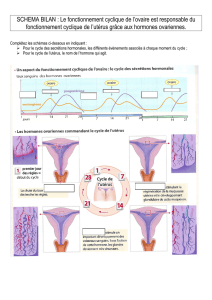
L’appareil génital féminin qui se met en place depuis le stade embryonnaire
jusqu’à la puberté, a un fonctionnement cyclique : le phénotype féminin
fonctionnel se caractérise ainsi par un développement et une régression cyclique
de l’endomètre, par l’ovulation et par des variations des concentrations en
hormones ovariennes (progestérone et oestrogènes). Par ailleurs, il a été établi que
ces changements sont en rapport avec la reproduction c’est-à-dire de la formation
puis la libération des gamètes et la gestation. On a aussi montré que les hormones
ovariennes interviennent directement.
Ici, on se propose de rechercher le moment où elles interviennent et de
préciser leur mode d’action.
I influence des hormones ovariennes sur la mise en place et la maturation
du phénotype féminin
I – 1) hormones ovariennes et établissement du phénotype
À partir du 7e mois de gestation, les gonades indifférenciées des
individus dont les chromosomes sexuels sont XX deviennent des
ovaires et un appareil génital féminin se développe. De plus,
l’injection d’hormones ovariennes à des fœtus ou des embryons
génétiquement mâles n’entraîne pas leur féminisation. Les hormones
ovariennes n’interviennent donc pas dans l’établissement du phénotype
sexuel féminin. C’est l’absence d’hormones mâles qui détermine cette
différenciation.
I – 2) hormones ovariennes et phénotype fonctionnel
En revanche, les hormones ovariennes sont indispensables à
l’apparition des premières règles et des premières ovulations donc à la
mise en fonctionnement de l’appareil reproducteur caractéristique de
la puberté. Cette période est en effet marquée par une augmentation
brutale des concentrations sanguines en hormones ovariennes.
De plus, les hormones ovariennes permettent le développement,
la maturation et le maintien des caractères sexuels primaires et
secondaires.
II hormones ovariennes, ovaires et libération des gamètes
II – 1) hormones ovariennes et ovulation
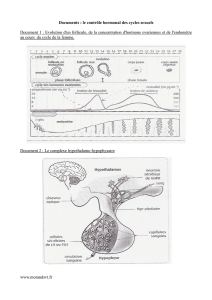
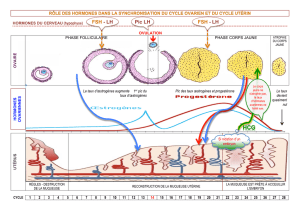
Les hormones ovariennes stimulent la formation des follicules.
Lorsque le follicule est arrivé à maturité, la libération de
l’ovocyte (ou des ovocytes - penser aux faux jumeaux) est associée à
un pic de LH et un pic d’œstrogènes, vers le 14e jour du cycle –

courbe du type de celle qui figure à la page 277, possible : on y voit le
pic de LH , d’oestrogènes… –
De plus, des expériences montrent :
1) que des concentrations élevées en LH sont indispensables pour
que l’ovulation ait lieu (décharge ovulante) ;
2) que les oestrogènes (à fortes concentrations) stimulent la
sécrétion de LH par l’hypophyse : rétrocontrôle positif.
Toutefois, les concentrations en hormones ovariennes et en LH
ne « s’emballent » pas : un rétrocontrôle négatif intervient ensuite.
II - 2) hormones ovariennes et rétrocontrôle négatif
Durant la phase lutéale, la concentration en progestérone est
élevée. Or des injections de progestérone inhibent l’activité du
complexe hypothalamo-hypophysaire : rétrocontrôle négatif cette fois !
Les concentrations « élevées » en hormones ovariennes s’oppose donc à
l’apparition d’un pic de LH durant la phase lutéale. ATTENTION : ces
concentrations restent malgré tout faibles et se mesurent tout de
même en ng.L-1.
Schéma possible page 282 : il résume les relations entre
l’utérus, les ovaires et le complexe hypothalamo-hypophysaire.
III Hormones ovariennes, utérus et gestation
III – 1) influence des hormones ovariennes sur l’endomètre
Si l’on procède à une ablation des ovaires, la muqueuse utérine
ne se développe pas (ou bien régresse si l’intervention survient au
début de la grossesse). Or la muqueuse utérine est indispensable à la
gestation chez les Mammifères. Une ou plusieurs injections
d’hormones ovariennes suffit à rétablir le développement de la
muqueuse utérine. Les hormones ovariennes favorisent donc le
développement de l’endomètre et par conséquent la reproduction ou
plus exactement la gestation. – schéma possible –
III – 2) hormones ovariennes et apparition des règles
Si la fécondation n’a pas lieu, le corps jaune régresse. Ceci
entraîne une diminution brutale des concentrations en hormones
ovariennes et par conséquent l’apparition des règles.
Conclusion
Les hormones ovariennes n’exercent pas d’influences notables
sur l’établissement du phénotype féminin mais elles sont
déterminantes pour sa maturation et pour ses fonctions dans la
reproduction. Ouvertures possibles : applications médicales et
contraception.

Éléments de correction du Bac blanc
partie II-1 (mai 2005)
Il ne s’agit surtout pas de faire une description exhaustive du document mais de trouver les 2 types
d'informations demandés par le sujet.
I. Les événements géologiques ayant affecté la région
Commencer par ce qui est le plus visible, puis aller progressivement dans les détails. Être précis dans
les informations extraites des documents : citer les documents, nommer les roches...
A. Des accidents tectoniques
Sur la carte, on voit que les couches du terrain ne respectent pas la topographie ce qui atteste des
bouleversements tectoniques : plissements du terrain. Ceci est confirmé par la coupe géologique qui
montre que la région se situe au dessus d’un synclinal.
Sur la carte on peut voir une grande faille de direction NW-SE qui passe également par la coupe AB.
B. Un substratum de nature sédimentaire
La légende du document permet de préciser la nature des terrains qui sont essentiellement d’origine
sédimentaire : marnes, calcaire, grès, conglomérat... Ces roches témoignent donc d’un autre phénomène
géologique : la sédimentation.
N.B. : une ambiguïté du sujet concerne les schistes. Dans son sens strict, le terme de schiste s’applique
aux roches ayant acquis une schistosité (débit en feuillets) sous l’influence de contraintes tectoniques :
ce sont alors des roches métamorphiques. Au sens large (qu’il vaut mieux éviter) le terme s’applique à
toute roche susceptible de se débiter en feuillets (ex : les schistes houillers du Pays de Galles qui sont
des roches sédimentaires !). La lecture de la légende doit lever cette ambiguïté puisque ces schistes
sont dans la colonne “Roches sédimentaires”.
C. Des recoupements de structure
Dans la zone supérieure de la carte, un terrain recoupe la série (direction NW-SE). La coupe permet
d’affirmer qu’il s’agit d’un filon de granite. Il est donc le témoin d’un événement magmatique.
Sur la carte, on peut également voir deux anomalies dans la disposition des roches sédimentaires : dans
la partie Est, une vaste formation recoupe la série sédimentaire. Il s’agit des schistes avec veines de
charbon (cf. coupe). Ce même document montre que ces schistes se sont déposés sur un terrain
fortement érodé. D’autres traces d’érosion sont visibles sur cette même coupe, notamment au nord du
filon granitique.
En résumé, une sédimentation est à l’origine des roches de la région.
Ces dépôts ont été affectés par des phénomènes tectoniques (plis et failles), des intrusions
magmatiques (filon de granite) et a subit une érosion intense.
Comment retracer l’histoire de ces événements ?
II. Une datation des événements géologiques
A. Datation relative de la série sédimentaire
Aucune information ne permet de fournir ici une datation absolue ; en revanche, s’agissant de roches
sédimentaires, le principe de superposition peut être appliqué.

Puisque les roches les plus récentes recouvrent les plus anciennes, l’ordre des dépôts se déduit de la
coupe :
a) formation du grès rouge ;
Éviter de dire dépôt des grès puisque un grès est un sable dont les éléments ont été, par la suite,
soudés entre eux au cours de la diagenèse
b) formation des conglomérats ;
Idem : les conglomérats sont des débris de roches qui ont ensuite été cimentés entre eux.
c) dépôt de marnes sableuses ;
d) formation des schistes ;
e) dépôt de calcaire renfermant une zone dolomitique ;
f) formation du grès jaune ;
g) formation des schistes à veines de charbon.
B. Datation des événements tectoniques et intrusifs
Les phénomènes tectoniques ou intrusifs sont postérieurs aux terrains qu’ils affectent.
Sur la carte et sur la coupe on peut voir que le synclinal affecte toute la série sédimentaire à l’exception
des schistes à veines de charbon. Le plissement est donc postérieur à la formation du grès jaune mais
antérieur à la formation des schistes à veines de charbon.
Le même raisonnement s’applique à la faille qui recoupe les terrains déjà plissés (cf. carte et coupe).
Elle est donc postérieure au plissement mais antérieure aux schistes à veines de charbon qui ne sont pas
affectés par cette cassure.
Sur la coupe, on voit que le filon granitique recoupe toute la série sédimentaire jusqu’au calcaire à
banc de dolomite. La carte permet de voir que même les terrains plus récents sont affectés (jusqu’au
grès jaune) à l’exception des schistes à veines de charbon. Compte tenu de l’orientation du filon
(semblable à celle de la faille visible) tout laisse penser que l’intrusion magmatique a eu lieu dans une
faille contemporaine de celle située plus à l’ouest. Dans ce cas l’intrusion serait donc postérieure au
bouleversement tectonique.
De même, on peut voir sur la coupe que l’érosion affecte la série sédimentaire et le filon granitique
mais pas les schistes à veines de charbon. Cette phase érosive a donc eu lieu après le phénomène
intrusif et les plis et failles.
On voit également sur la coupe que les schistes à veines de charbon ont également subit une érosion
mais celle-ci est indépendante de la précédente et donc très récente.
III. Géochronologie de la région (bilan)
Les différents événements se seraient donc déroulés dans l’ordre suivant :
1) sédimentation
a) formation du grès rouge ;
b) conglomérats ;
c) dépôt de marnes sableuses ;
d) formation des schistes ;
e) dépôt de calcaire renfermant une zone dolomitique ;
f) formation du grès jaune ;
2) accidents tectoniques “souples” : plissement des terrains ;
3) accidents tectoniques cassants : failles ;
4) intrusion de granite dans certaines failles : filons ;
5) érosion ;
6) formation des schistes à veines de charbon. ;
7) érosion.
Modifications à apporter :
- indiquer en intro qu'un plan n'est pas attendu
- IA : remplacer synclinal par pli (synclinal)
- Titre IB : Des roches de nature sédimentaire au lieu D'un substratum...

Éléments de correction
Partie II - Exercice 2
DOCUMENT 1
Soient P1 le parent de phénotype sauvage [b+, c+ r+] et P2 le
parent de phénotype mutant [b, c, r].
On sait, que puisqu'ils sont de lignée pure, ils sont
homozygotes. Le parent P1 ne possède donc que les allèles
sauvages b+, c+ et r+ alors que le parent P2 ne possède que les
allèles mutants b, c et r.
A ce stade on ne peut pas écrire les génotypes parentaux car
on ne sait pas si B, C et R sont sur le même chromosome ou
sur des chromosomes différents.
Le croisement de P1 et P2 produit des individus hybrides F1,
tous de même génotype hétérozygote. Cela signifie que pour
chaque gène B, C et R les individus F1 portent un allèle
sauvage et un allèle mutant.
Puisque les hybrides hétérozygotes F1 ont tous le phénotype
sauvage [b+, c+, r+], ce phénotype sauvage est dominant sur
le phénotype mutant. On a donc respectivement [b+] qui
domine [b], [c+] qui domine [c] et [r+] qui domine [r].
DOCUMENTS 2 et 3
On appelle croisement test (= test cross ou croisement en
retour) le croisement d'un individu quelconque, ici F1, avec un
individu de phénotype récessif pour chacun des caractères
étudiés, ici P2.
Dans le cas d'un dihybridisme (étude de deux caractères
comme dans les documents 2 et 3) où un F1 est utilisé, le test
cross permet de localiser les deux gènes l'un par rapport à
l'autre. C'est à dire savoir si les deux gènes étudiés sont liés (=
portés par le même chromosome) ou indépendants (= portés
par des chromosomes différents).
À l'issue d'un tests-cross, les proportions phénotypiques
obtenues traduisent les proportions et la nature des gamètes
produits par l'individu de génotype quelconque (ici F1) :
- si deux gènes sont liés on sait qu'on doit obtenir beaucoup plus
de phénotypes parentaux (phénotype identique à l'un des
parents) que de phénotypes recombinés (combinaison de
phénotypes parentaux dus à des crossing-over).
- si deux gènes sont indépendants (portés par des chromosomes
différents) on sait qu'on doit obtenir des phénotypes parentaux
et recombinés en quantité équivalente.
DOCUMENT 2 - Cas des gènes B et C
Le nombre de phénotypes parentaux obtenus (107 + 109)
étant très supérieur au nombre de phénotypes recombinés (38 +
40), on en déduit que B et C sont liés.
Voir schéma n° 1 (début de prophase 1, appariement des
chromosomes)
Croisement F1 x P2
F1
P2
phénotypes
[b+, c+]
[b, c]
génotypes
b+c+ // b c
b c // b c
Si B et C sont liés, dans certaines cellules, le chromosome
portant B et C subit des recombinaisons (crossing-over). Ces
dernières sont sans conséquence pour P2, qui ne peut produire
qu'un seul type de gamète (bc), mais elles permettent à F1 de
produire 4 types de gamètes non équiprobables. En effet, le
crossing over ne se produisant pas dans toutes les cellules, F1
produit plus de gamètes de type parental (b+c+ et b c) que de
gamètes recombinés (b+ c et b c+).
Voir schéma n° 1 (fin de prophase 1 (crossing-over) et
télophase 2)
Tableau de croisement du document 2
gamètes parentaux
gamètes recombinés
36 %
37 %
13 %
14 %
gamètes
F1
P2
b+ c+
b c
b+c
b c+
b c 100%
b+c+//bc
bc//bc
b+c//bc
bc+//bc
phénotypes
[b+, c+]
[b, c]
[b+, c]
[b, c+]
nombre
107
109
38
40
%
36 %
37 %
13 %
14 %
phénotypes
parentaux
73 %
phénotypes
recombinés
27 %
DOCUMENT 3 - Cas des gènes C et R
Le nombre de phénotypes parentaux obtenus (72 + 75) étant
égal au nombre de phénotypes recombinés (73 + 74), on en
déduit que C et R sont indépendants.
Voir schéma n° 2 ( prophase 1).
Croisement F1 x P2
F1
P2
phénotypes
[c+, r+]
[c, r]
génotypes
c+ // c , r+ // r
c // c, r // r
Si C et R sont indépendants, F1 produit 4 types de gamètes
équiprobables : 2 types de gamètes parentaux (c+ r+, c r), et 2
types de gamètes recombinés (c+ r, c r+). P2 produit un seul
type de gamète (c r).
Voir schéma n° 2 ( télophase 2).
Tableau de croisement du document 3
gamètes parentaux
gamètes recombinés
24%
26 %
25 %
25 %
gamètes
F1
P2
c+ r+
c r
c+ r
c r+
c r
c+// c
r+ //r
c // c
r // r
c+ // c
r // r
c // c
r+ // r
phénotypes
[c+, r+]
[c, r]
[c+, r]
[c r+]
nombre
72
75
73
74
%
24%
26 %
25 %
25 %
phénotypes
parentaux
50 %
phénotypes
recombinés
50 %
CONCLUSION
Les gènes B et C sont portés par le même chromosome, alors
que le gène R est porté par un autre chromosome.
On est maintenant en mesure d’écrire les génotypes parentaux :
P1 b+ c+//b+ c+ r+//r+ et P2 b c// b c r//r
et F1 b+ c+//b c r+//r
 6
6
 7
7
1
/
7
100%