TP : Explication Moléculaire Double Spécificité Enzyme

TP 3 : EXPLICATION MOLECULAIRE DE LA DOUBLE SPECIFICITE D’UNE ENZYME
Mise en situation et recherche à mener
Les enzymes, digérant les sucres, fonctionnent comme toute autre enzyme. Nous étudierons, ici la carboxypeptidase, une enzyme digestive
pancréatique responsable de la digestion des polypeptides. Nous savons que les enzymes sont étroitement spécifiques d’une réaction chimique
donnée, réalisée sur un type de substrat défini. Donc le substrat va être en quelque sorte reconnu par l’enzyme.
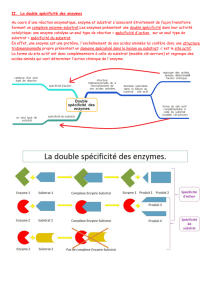
On cherche à mettre en évidence que l’enzyme possède une structure tridimensionnelle particulière qui permet d’expliquer sa double
spécificité.
Ressources
Le site actif de l’enzyme, lieu d’interaction avec le substrat, est composé :
-d’un site de reconnaissance du substrat, impliquant quelques acides
aminés et intervenant dans la spécificité au substrat ;
-d’un site catalytique, impliquant deux ou trois acides aminés, où se
réalise la réaction chimique avec le substrat et qui intervient donc dans la
spécificité d’action.
Pour la carboxypeptidase six acides aminés constituent son site actif et
interviennent dans la catalyse, on distingue :
- trois acides aminés constituent le site catalytique : Histidine 69, Acide
glutamique 72, Histidine196 ,
- trois acides aminés constituent le site de reconnaissance : Arginine 145,
Tyrosine 248 et acide glutamique 270.
Matériel
-Rastop : logiciel de visualisation en 3D
-une fiche méthodologique du logiciel
Molécules de Rastop
- carboxypeptidase : CPASEUL.PDB
- carboxypeptidase et son substrat : CPASUB.PDB
- site actif seul de l'enzyme : siteseul.pdb
- site actif avec le substrat : sitesub.pdb
- carboxypeptidase mutée et son substrat : mut_cpasub.pdb
Etape 1 : Concevoir une stratégie pour résoudre une situation-problème (durée maximale : 10 minutes)
Exigences
1) Proposer une démarche d’investigation permettant d’établir les caractéristiques structurales de l’enzyme qui explique sa
double spécificité. Appeler l’examinateur pour vérifier votre proposition et obtenir la suite du sujet.
Concevoir une démarche
réalisable

Etape 2 : Mettre en œuvre un protocole de résolution pour obtenir des résultats exploitables
2) Mettre en œuvre le protocole afin de préciser le rôle de la structure des enzymes dans leur
fonctionnement.
Appeler le professeur pour vérifier les résultats et éventuellement obtenir une aide.
- Maîtrise du logiciel Rastop
Etape 3 : Présenter les résultats pour les communiquer
3) Sous la forme de votre choix, traiter les données obtenues pour les communiquer.
- Maîtrise du logiciel Word ou powerpoint
Etape 4 : Exploiter les résultats obtenus pour répondre au problème
4) Exploiter les résultats pour montrer que la structure tridimensionnelle des enzymes permet
leur double spécificité
- Explication cohérente avec les observations
réalisées.

Protocole
1) Comparaison de la carboxypeptidase et de la carboxypeptidase et son substrat :
a) Ouvrir les deux molécules ( CPASEUL et CPASUB) en fenêtres mosaïque verticale
b) Faire un affichage en sphère VDW pour ces deux molécules.
c) Colorer l’enzyme et le substrat de deux couleurs différentes : menu : « Atomes », « colorer
par », « Chaine» .
d) Afficher le squelette carboné de l’enzyme dans la fenêtre CPASUB ; pour cela :
- Sélectionner substrat en cliquant sur l’icône « AbC», taper « *S » (tous les
atomes qui appartiennent au substrat sont sélectionnés). Le nombre d'atomes sélectionnés
s'affiche en bas dans la zone grise
- Sélectionner l'enzyme en utilisant le bouton "inverser la sélection"
- Aller dans « atomes -représentation- effacer » (seul le substrat reste affiché)
- Aller dans « Rubans » « squelette carboné »
e) Afficher également l’enzyme seul en squelette carboné dans la fenêtre CPASEUL; pour cela :
- Sélectionner la totalité de l'enzyme (icône "tout sélectionner")
- Aller dans " atomes- représentation- effacer" (toute la molécule disparait)
- Aller dans "Rubans - squelette carboné"
- Colorer en bleu le squelette carboné de l'enzyme
f) Identifier les caractéristiques du complexe enzyme-substrat
g) Copier les images dans votre fiche réponse Word ou powerpoint
h) Fermer les images sans les enregistrer.
2) Comparaison des sites actifs : site actif seul et site actif avec un substrat :
a) Ouvrir les deux molécules (siteseul et sitesub) en fenêtres mosaïque verticale.
b) Afficher le substrat en sphères VDW rouges et les acides aminés du site actif en bâtonnets.
c) Identifier les acides aminés des sites catalytique et de fixation en les colorant, dans les deux
fichiers, de deux couleurs différentes : respectivement jaune et rose.
d) Identifier les caractéristiques du site actif en présence du substrat
e) Copier les images dans votre fiche réponse
f) Fermer les images sans les enregistrer
3) Comparaison d’une carboxypeptidase et substrat avec une carboxypeptidase mutée et
substrat :
a) Ouvrir les deux molécules (CPASUB et mut_cpasub.pdb) en fenêtres mosaïque verticale.
b) Afficher le substrat en sphères VDW rouges et l'enzyme en bâtonnets bleus.
c) Sélectionner les acides amines des sites catalytique et de fixation et les colorer avec le même
code couleur que précédemment.
d) Identifier les différences entre les deux sites actifs
e) Copier les images dans votre fiche réponse
f) Fermer les images sans les enregistrer

FICHE METHODE :
Utilisation maîtrisée du logiciel RASTOP.
Icônes d’aspect des atomes (sphères, étoiles, fil de fer…)
Déplacer et explorer la molécule.
a- Pour manipuler les molécules plus aisément, utiliser les raccourcis clavier et la souris :
o rotation de la molécule : bouton gauche et déplacement de la souris ;
o déplacement de la molécule : bouton droit maintenu enfoncé ;
o zoom avant/arrière : rester appuyer sur ctrl et utiliser la roulette de la souris.
b- Pour réinitialiser les paramètres d'origine de la molécule, cliquer sur le bouton Restaurer.
c- Pour faire tourner automatiquement la molécule sur elle-même, cliquer sur le bouton Pivoter.
d- Pour explorer le cœur de la molécule masqué par d’autres atomes :
o Créer un plan de coupe de la molécule en cliquant sur le bouton Front.
o Cliquer sur le bouton flèche de droite (à côté de Front), afin de déplacer ce plan en profondeur.
o Cliquer sur le bouton flèche de gauche (à côté de Front), afin de reconstituer la molécule.
Pour obtenir des informations sur un atome ou une partie de la molécule, cliquer sur la molécule ; des informations
intéressantes, comme le type et le numéro de l'atome sélectionné, ainsi que le nom de la chaîne, le nom de l'acide
aminé et son numéro (pour les protéines) s’affichent dans cette fenêtre.
Ouvrir une
molécule
Coloration de la
molécule selon
différents critères
Sélectionner une partie de la
molécule :
- *S : sélection de tous les
atomes du substrat.
- 145 : sélection de tous les
atomes de l’acide aminé numéro
145. Pour sélectionner plusieurs
acides aminés taper 34 ,145 ,67
(sélection des acides aminés 34,
145 et 67).
Fenêtres
ouvertes
juxtaposées
Modification de
l’aspect des liaisons.
Modification de
l’affichage de la
structure spatiale de
la molécule
Affichage des
molécules dans des
fenêtres juxtaposées
Inverser la sélection :
Sélectionne tous les
atomes non sélectionnés
par l’onglet AbC. Si vous
faites *S puis inverser la
sélection, vous
sélectionnerez tous les
atomes de l’enzyme.
Ouvrir la palette
de couleur
1
/
4
100%