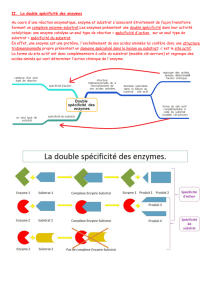
Structure et fonction du complexe enzyme-substrat, avec Rastop

SVT 1ère S, Thème I, Chapitre I.
TP 4 : Structure et fonctionnement du complexe enzyme-substrat
Objectif : Comprendre le fonctionnement du complexe enzyme-substrat au niveau moléculaire.
Matériel : On propose l’étude de la pepsine, une protéase catalysant la digestion des oligopeptides dans l’estomac des Vertébrés.
Le logiciel Rastop permet une modélisation en 3 dimensions de cette enzyme lorsqu’elle est, ou pas, fixée à son substrat
(complexe enzyme-substrat). Un livre est nécessaire par binôme. Rédigez une fiche de TP personnelle.
Méthodes : Activités : Consignes techniques correspondantes :
Exploiter
un modèle.
1. Affichez le complexe pepsine-
substrat de façon à étudier sa
structure générale. La coloration par
chaîne/segment permet de distinguer
l'enzyme de son substrat (chaîne
courte). Résumez vos observations
par une phrase et/ou un schéma.
a) Cliquer Fichier, ouvrir, 1pso.pdb.
b) Faire un clic-glissé pour faire tourner la molécule ; un shift-clic-
glissé pour zoomer et dézoomer. Pas de geste brusque !
c) Cliquez sur l’icône Sphères VDW (1 atome = 1 sphère) puis sur
Rubans (squelette carboné seul) puis sur Bâtonnets (liaisons
chimiques seules). Les couleurs identifient les atomes.
d) Cliquer Atomes, Colorer par…, Chaînes. Le substrat et l’enzyme
prennent 2 couleurs différentes.
2. Superposez la pepsine seule à ce
complexe pepsine-substrat afin de
voir l'effet de la liaison du substrat
sur la forme 3D de l'enzyme.
Comparez précisément les formes et
interprétez.
Remarque : Le logiciel ne permet pas
d’annuler la dernière action faite, en
cas d’erreur ineffaçable, il faut…
fermer le fichier et tout recommencer !
a) Cliquez sur l’icône Sélectionner une chaîne, cliquer sur l’enzyme
seule puis sur l’icône Rubans. Laissez le substrat en bâtonnets.
b) Cliquer sur Restorer en bas au milieu de l’écran, la molécule
reprend sa position initiale dans l’espace.
c) Cliquer Fichier, Ajouter, 1psn.pdb.
d) Cliquez à nouveau sur l’icône Rubans. La seconde enzyme (sans
substrat) s’affiche dans une autre couleur que la première.
e) Cochez ou décochez le bouton Univers en bas à gauche de
l’écran pour faire pivoter les deux enzymes ensemble ou
seulement la dernière molécule chargée (1psn).
f) Sélectionnez Rot ou Trans/zoom et faites glisser les curseurs des
axes X, Y et Z pour opérer des rotations ou translations.
Formuler
une
hypothèse
explicative
.
3. Sur le complexe pepsine-substrat
(1pso) seul, effectuez une restriction
de l'observation aux atomes de
l'enzyme situés à une distance de
moins de 6 nm (= 1500 RU pour
Rastop Unity) autour du substrat.
Relevez les acides aminés qui leur
correspondent (négligez les atomes
apparaissant en rouge lors d’une
coloration par chaîne) et leurs type
de chaîne latérale (livre p.363).
Formulez une hypothèse quant au
rôle de ces acides aminés dans le
complexe.
a) Cliquez sur l’icône Sélectionner une molécule, cliquer sur
l’enzyme seule (psn) puis sur Molécule et Effacer la molécule.
b) Cliquez sur l’icône Sélectionner une chaîne, cliquer sur le
substrat seul (48 atomes sont sélectionnés).
c) Cliquer sur Editer, Sélectionner, Distant de… et inscrivez 1500.
d) Cliquer sur Editer puis Restreindre, les atomes éloignés du
substrat de plus de 6 nm disparaissent.
e) Cliquer Atomes, Colorer par… et Groupe (les acides aminés
contigüs prennent des couleurs différentes).
f) Affichez les atomes en Bâtonnets, sélectionnez le substrat seul en
cliquant dessus pour mettre sa forme en Sphères VDW.
g) Décochez l’icône de Sélection par chaîne ; en cliquant sur un
groupe d’atomes d’une même couleur, vous voyez apparaître le
nom de l’acide aminé qui le porte dans la ligne d’information en
bas de la fenêtre. Voir votre livre p.363 pour la traduction.
4. Affichez d'éventuelles liaisons
hydrogène et ponts disulfures
(liaisons faibles électrostatiques, non
covalentes, entre atomes d'acides
aminés différents) afin de connaître
leur rôle dans le complexe pepsine-
substrat. Faites une hypothèse quant
à ce rôle.
5. Sachant que les liaisons hydrogène
et autres liaisons faibles sont
facilement brisées par des chocs
moléculaires dus à la chaleur ou par
une variation de pH dans la solution,
proposez une explication à
l’existence de conditions physico-
chimiques optimales d’activité
pour toute enzyme.
a) Cliquer sur l’icône carrée bleu clair Sélectionner tout.
b) Cliquer dans le menu Liaisons, Liaisons hydrogènes, Afficher.
Vous pouvez les masquer en cliquant Effacer.
c) Même procédé pour afficher les ponts disulfures éventuels et les
faire disparaître.
d) Sélectionnez uniquement la chaîne enzyme en cliquant sur un de
ses carbones et réaffichez-la en ruban.
e) Réaffichez si besoin les liaisons à étudier avec les procédés
décrits ci-avant.
f) N’effectuez AUCUN enregistrement avant de fermer le fichier
1pso.pdb.
Faire une
synthèse.
6. Résumez quelles caractéristiques moléculaires du complexe enzyme-substrat permettent d’expliquer la
spécificité de substrat.
1
/
1
100%