Poly de TD Gpops 2016 - Cours en Ligne

G
ENETIQUE DES POPULATIONS
Support de travaux-dirigés
2016-2017
UFR de Génétique Évolutive et Amélioration des Plantes
Karine Alix-Jenczewski, karine.alix@
agroparistech.fr
Philippe Brabant, [email protected]
Julie Fiévet, [email protected]
Pierre Gérard, [email protected]
Jean-Pierre Henry, jphenry@
agroparistech.fr

AVERTISSEMENT
Le présent document sert de support aux travaux dirigés. Il présente des
exercices qui seront développés au cours des séances de TD, et il résume dans
des encadrés introductifs ou de bilan de chaque séance, les notions essentielles à
retenir en génétique des populations.
Il ne dispense pas de la lecture du cours polycopié intitulé "Génétique des
Populations". Vous serez supposés avoir lu et compris ce polycopié de cours en
vue de l'examen, à l'exception de certains passages traitant de notions non vues
en TD, et qui vous seront précisés en temps utile. Aussi, il vous est vivement
recommandé de faire cette lecture au fur et à mesure de l'avancement des TD.
Les chapitres et pages du polycopié correspondant aux notions traités aux
diverses séances sont précisées dans ce document-ci, à la fin du texte
correspondant à chaque séance.

Génétique des populations - 1ère année AgroParisTech – 10/2016
1
La génétique des populations s'intéresse à la composition génétique des populations et à ses
variations dans l’espace et dans le temps. Elle fournit un cadre formel permettant de décrire
cette composition, de comprendre les mécanismes qui déterminent l'évolution des
populations, pour reconstruire leur histoire ou prédire leur évolution future. Ses applications
concernent les divers domaines de, l’évolution, la sytématique, la gestion des populations
naturelles ou domestiques (sélection, conservation, lutte...), la gestion de la biodiversité, et de
domaines qui ont trait à la diversité génétique humaine (médecine, épidémiologie, pharmaco-
génétique, médecine légale...)
Quelques définitions :
Gène : unité d'information biologique, transmise au cours des générations, codant pour une
fonction. Correspond à une séquence d'une macromolécule (ADN ou ARN) transmise
telle quelle (à de rares modifications près), transcrite et généralement traduite par un
système biochimique idoine, ce qui permet l'expression d'une activité biologique.
Locus : historiquement, position du gène sur le chromosome. En génétique des
populations, ensemble de gènes homologues (classe d'homologie). Deux chromosomes
ou deux gènes sont homologues s’ils s’apparient et s’excluent mutuellement à la
méiose : on ne trouvera donc pas deux gènes homologues présents en même temps dans
une cellule haploïde.
Gènes allèles : deux gènes homologues sont dits allèles quand ils ont des formes
différentes. Un allèle est donc une classe de gènes homologues qu’on ne distingue pas
entre eux au niveau phénotypique. Le phénotype est ce qu’on observe, il dépend donc
du niveau d’observation. Dès lors, le nombre d’allèles identifiés à un locus dépend lui
aussi du niveau d’observation. Par exemple, pour une enzyme on peut trouver deux
allèles lorsqu’on s’intéresse à sa fonctionnalité (fonctionnelle vs non fonctionnelle),
mais au sein de chacun de ces deux allèles il est possible d’observer différentes
séquences protéiques et en remontant jusqu’aux gènes différentes séquences
nucléotidiques. Pour une même protéine, le nombre d'allèles trouvés au niveau des
séquences est en général beaucoup plus élevé que le nombre observé au niveau
macroscopique.
Gènes identiques par descendance : gènes issus de la transmission d'un même gène
ancestral, sans qu'aucune mutation ne se soit produite dans la chaîne de parenté.
Haplotype : séquence (pouvant couvrir plusieurs gènes et séquences non codantes, et
même tout un chromosome) qu'on reconnaît comme différente d'une autre séquence
homologue. Plus particulièrement utilisé pour désigner le génotype haploïde pour un
ensemble de gènes ou séquences étroitement liés sur un chromosome (donc tendant à
être hérités en bloc) et différent d'un autre en plusieurs sites. Le terme s'est imposé avec
le développement des méthodes moléculaires permettant de caractériser les séquences
d'ADN.

Génétique des populations - 1ère année AgroParisTech – 10/2016
2
Diversité génétique d’une population traditionnelle de maïs
révélée par un marqueur RFLP :
sonde BA1/enzyme HindIV
Figure 1 : Trois séries d’électrophorèses réalisées à partir d’un échantillon d’individus d’une
population traditionnelle de maïs et révélant un marqueur RFLP. Chaque puits contient
l’ADN d’un individu. Les puits notés "Témoin" sont des témoins de migration.
Deux allèles sont présents au locus considéré. On peut reconnaitre les homozygotes au fait
qu’ils ne présentent qu’une bande haute ou basse et les hétérozygotes qui ont les deux bandes.

Génétique des populations - 1ère année AgroParisTech – 10/2016
3
S
EANCE N
°
1
Structure génétique des populations
et Loi de Hardy-Weinberg
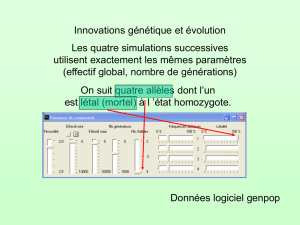
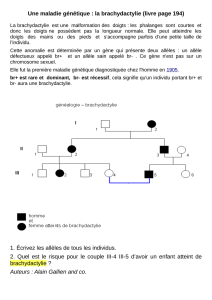
La brachydactylie envahira-t-elle l’espèce humaine ?
A un locus, la composition génétique d'une population est entièrement définie par les
fréquences génotypiques et les fréquences alléliques à ce locus. Les fréquences alléliques
se déduisent facilement des fréquences génotypiques.
Exemple : dans une population avec 50 individus dont 25 de génotype A
1
A
1
, 20 de génotype
A
1
A
2
et 5 de génotype A
2
A
2
,
- les fréquences des génotypes A
1
A
1
, A
1
A
2
et A
2
A
2
sont respectivement 0,5, 0,4 et 0,1 ;
- il y a au total 100 gènes, dont 70 gènes A
1
et 30 gènes A
2
; donc les fréquences des allèles A
1
et A
2
(notées traditionnellement p et q) valent respectivement p = 0,7 et q = 0,3.
Exercices :
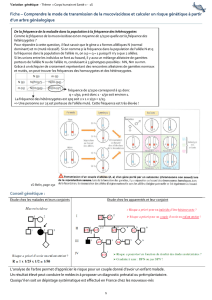
1) Calculer les fréquences alléliques dans les populations suivantes :
AA Aa aa (Effectif total)
Population 1 : 28 24 48 (100)
Population 2 : 16 48 36 (100)
Population 3 : 40 0 60 (100)
Population 4 : 0 80 20 (100)
Qu'en concluez-vous ?
2) Une population est analysée à un locus particulier, où elle présente la composition
suivante :
Génotypes AA Aa aa
Fréquences D
0
H
0
R
0
(D
0
, H
0
et R
0
sont les fréquences des trois génotypes à la génération 0. Ce sont des valeurs quelconques, dont
la somme fait 1; "D" pour "dominant", "H" pour "hétérozygote", "R" pour "récessif".)
Comment évoluent dans cette population les fréquences alléliques d’une part, et les
fréquences génotypiques d’autre part, si les gamètes A et a s'unissent au hasard (situation
appelée "panmixie") ?
Quelles sont les autres hypothèses implicites de ce modèle ?
3) Que pensez-vous de la suggestion de Yule (cf. p. suivante) concernant la brachydactylie
(cf. illustration au verso) ?
 6
6
 7
7
 8
8
 9
9
 10
10
 11
11
 12
12
 13
13
 14
14
 15
15
 16
16
 17
17
 18
18
 19
19
 20
20
 21
21
 22
22
1
/
22
100%